北京中科测试 Metagenome 分析结题报告
| 合同编号 | HXXXSCXXXXXXXX |
| 合同名称 | XXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXX |
| 分期编号 | X101SCXXXXXXXX-XXX-XXXX |
| 报告时间 | 2025-09-18 |
| 售后服务电话 | 010-82726200 |
| 售后服务邮箱 | bjzklh@163.com |
| 售后服务云平台 | https://wwww.bjzklh.com/ 说明:您可以登录云平台并进行数据分析; |
| 温馨提示 | Report仅供展示部分结果,全部分析内容详见Result文件夹; 在您拿到Result前Report中的Result超链接无效,确认结算后,释放结果文件Result中Report的结果目录超链接有效。 |
1 项目简介
微生物群体存在于世界各类生态系统中,从个体体表到肠道,从高原空气到深海海底淤泥,从冰川冻湖到火山岩浆,微生物无处不在,并扮演着不可或缺的角色。对微生物的研究从 Antoni van Leeuwenhoek 发明显微镜开始的数百年中,主要基于纯培养的研究方式。在数以万亿计的微生物种类中,仅 0.1%-1% 的物种可培养(Chen et al., 2005),极大地限制了对微生物多样性资源的研究和开发。
宏基因组学(Metagenomics),是由Handelman (Handelsman et al., 1998)最先提出的一种直接对微生物群体中包含的全部基因组信息进行研究的手段。之后,宏基因组学的定义为 “绕过对微生物个体进行分离培养,应用基因组学技术对自然环境中的微生物群落进行研究”的学科。它规避了对样品中的微生物进行分离培养,提供了一种对不可分离培养的微生物进行研究的途径,更真实地反映样本中微生物组成、互作情况,同时在分子水平对其代谢通路、基因功能进行研究(Tringe et al., 2005)。
近年来,随着测序技术和信息技术的快速发展,利用下一代测序技术(Next Generation Sequencing, NGS)研究宏基因组学,能快速准确得到大量生物数据和丰富的微生物研究信息,从而成为研究微生物多样性和群落特征的重要手段(Tringe et al., 2005; Raes et al., 2007)。如致力于研究微生物与人类疾病健康关系的人体微生物组计划(Human Microbiome Project, HMP),研究全球微生物组成和分布的全球微生物组计划(Earth Microbiome Project, EMP)都主要利用高通量测序技术进行研究。
项目整体流程图如下:
图1 项目整体流程
2 建库测序流程
检测合格的DNA样品用Covaris超声波破碎仪随机打断成长度约为350 bp的片段,经末端修复、加A尾、加测序接头、纯化、PCR扩增等步骤完成整个文库制备。文库构建流程图如下:
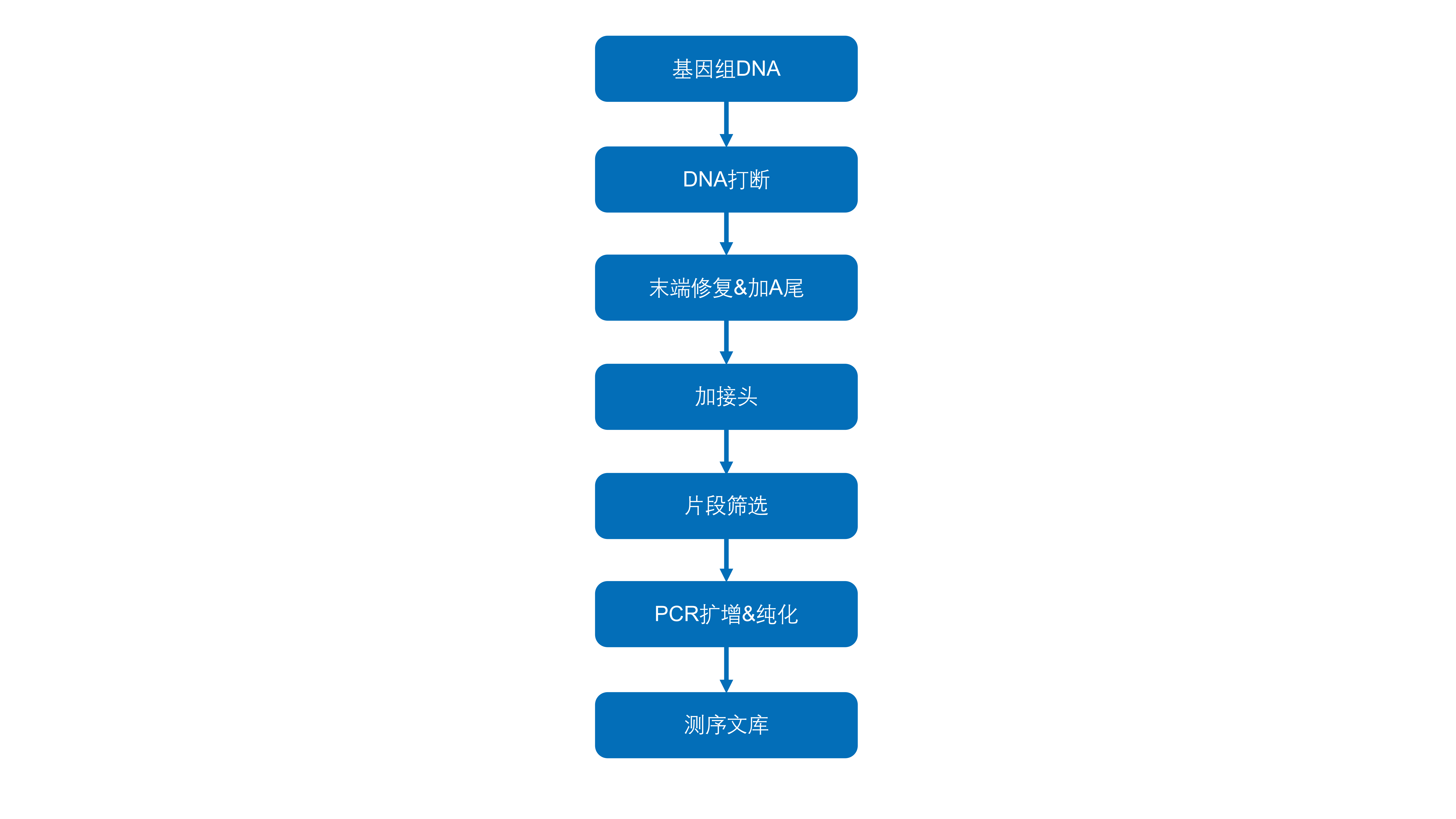
图2 文库构建流程图
文库的检测主要包括2种方法:
(1) AATI检测文库片段的完整性及插入片段大小
(2) qPCR检测文库有效浓度
库检合格后,把不同文库按照有效浓度及目标下机数据量的需求 pooling 后进行双端测序(PE150)。
3 信息分析流程
测序得到的原始数据(Raw Data)存在一定比例的低质量数据,为了保证后续信息分析结果的准确可靠,首先要对原始数据进行质控及宿主过滤,得到有效数据(Clean Data)之后,根据样品质控后的 Clean Data ,进行组装后,得到scaftigs。基于组装后的 scaftigs ,进行基因预测与去冗余,构建 gene catalogue,从而进行相应的gene catalogue 在各样品中的丰度信息计算。
物种丰度方面,将gene catalogue与Micro_NR库进行比对,获得每个基因(unigene)的物种注释信息,并结合基因丰度表,获得不同分类层级的物种丰度表。
功能注释方面,根据常用的功能数据库,从gene catalogue出发,进行代谢通路数据库(Kyoto Encyclopedia of Genes and Genomes, KEGG)、同源基因簇数据库(Evolutionary genealogy of genes: Non-supervised Orthologous Groups, eggNOG)、碳水化合物酶数据库(Carbohydrate-Active enzymes Database, CAZy)、细菌致病菌毒力因子数据库(Virulence Factor Database, VFDB)、病原与宿主互作数据库(Pathogen-Host Interactions, PHI)功能注释和丰度分析。
基于物种丰度和功能丰度,进行丰度聚类分析,PCA和NMDS降维分析,Anosim分析,样品聚类分析;当有分组信息时,可以进行MetagenomeSeq和LEfSe多元统计分析以及代谢通路比较分析,挖掘样品之间的物种组成和功能组成差异。
抗性基因注释方面,根据抗生素抗性基因数据库(Comprehensive Antibiotic Resistance Database, CARD),转移元件数据库(Mobile Genetic Elements, MGEs)进行注释,可以获得抗性基因丰度分布情况以及这些抗性基因的物种归属和抗性机制。
信息分析流程如下图:
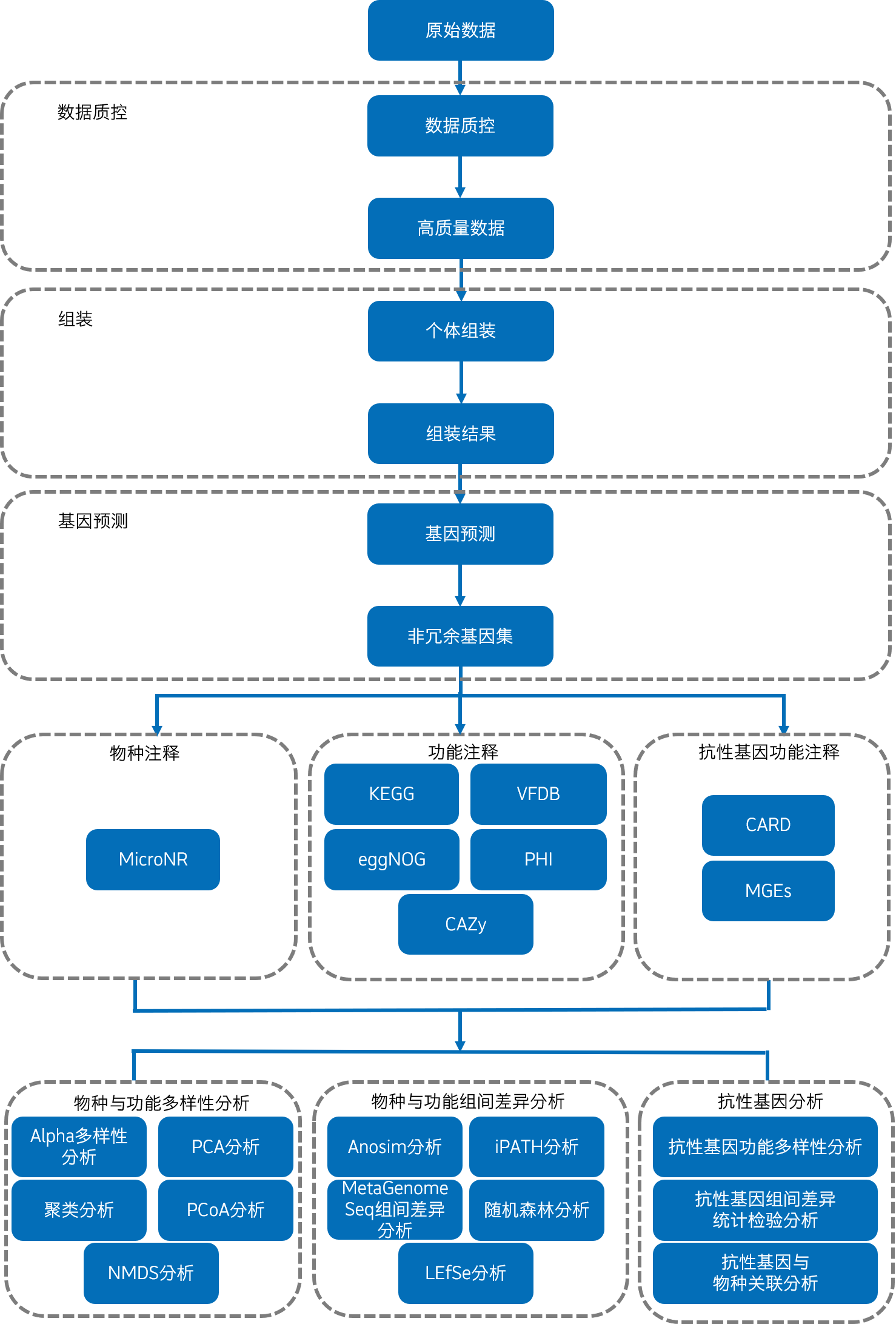
图3 信息分析流程图
说明:当样品数目小于3个时,无法进行PCA,PCoA,NMDS,聚类分析,丰度聚类热图分析;当分组内的生物学重复数目小于3个时,诸如Anosim,MetaGenomeSeq,LEfSe等统计分析皆没有统计学意义,将不进行此类分析。
4 分析结果
4.1 数据处理
对原始测序数据进行预处理,包括:当任一测序read中含有接头序列,去除此paired reads;当任一测序read中含有的低质量(Q<=5)碱基数超过该条read碱基数的50%时,去除此paired reads;当任一测序read中N含量超过该read碱基数的10%时,去除此paired reads。如果样品存在宿主污染,需与宿主序列进行比对,过滤掉可能来源于宿主(Karlsson et al., 2012; Karlsson et al., 2013; Scher et al., 2013) (默认采用 Bowtie2 软件),从而获取用于后续分析的有效数据(clean data)。上述处理步骤均是对read 1和read 2进行操作。测序数据预处理统计结果见下表。
表4.1 数据处理统计结果
| SampleID | Raw_Base(G) | Clean_Base(G) | Clean_Q20(%) | Clean_Q30(%) | Clean_GC(%) | Effective(%) | NonHost_Base(G) |
|---|---|---|---|---|---|---|---|
| A1 | 6.07 | 6.03 | 97.10 | 91.62 | 50.34 | 99.22 | 6.01 |
| A2 | 6.52 | 6.47 | 97.84 | 93.69 | 51.00 | 99.32 | 6.20 |
| A3 | 6.32 | 6.29 | 97.56 | 92.87 | 50.01 | 99.50 | 6.25 |
| A4 | 6.19 | 6.16 | 97.66 | 93.12 | 50.64 | 99.43 | 6.13 |
| A5 | 6.12 | 6.06 | 97.58 | 92.93 | 49.01 | 99.05 | 5.47 |
| A6 | 7.04 | 7.00 | 98.22 | 94.71 | 50.63 | 99.41 | 6.72 |
| B1 | 11.67 | 11.46 | 97.83 | 93.90 | 47.04 | 98.21 | 6.43 |
| B2 | 9.68 | 9.50 | 97.67 | 93.46 | 48.33 | 98.12 | 5.93 |
| B3 | 6.10 | 6.02 | 96.96 | 91.34 | 51.11 | 98.70 | 5.99 |
| B4 | 6.24 | 6.18 | 97.42 | 92.56 | 48.49 | 99.08 | 5.38 |
| B5 | 6.10 | 6.03 | 96.94 | 91.25 | 51.13 | 98.92 | 5.92 |
| B6 | 7.52 | 7.41 | 97.24 | 92.23 | 48.42 | 98.65 | 5.78 |
| C1 | 6.47 | 6.43 | 97.24 | 92.09 | 50.59 | 99.31 | 6.04 |
| C2 | 7.98 | 7.90 | 97.67 | 93.28 | 47.86 | 98.95 | 6.27 |
| C3 | 8.29 | 8.18 | 97.69 | 93.38 | 47.00 | 98.70 | 6.06 |
| C4 | 6.17 | 6.12 | 97.66 | 93.20 | 45.85 | 99.20 | 5.75 |
| C5 | 8.90 | 8.75 | 97.57 | 93.19 | 46.48 | 98.33 | 5.19 |
| C6 | 7.93 | 7.85 | 97.78 | 93.62 | 48.02 | 98.96 | 6.62 |
显示注释
Raw_Base(G):测序原始数据的总碱基数,即为测序序列的个数乘以测序长度算得,以G为单位
Clean_Base(G):过滤之后的有效数据量,过滤后测序序列的个数乘以测序序列的长度,以G为单位
Clean_Q20(%):Clean_Base 中 Phred 数值大于20的碱基占总体碱基的百分比
Clean_Q30(%):Clean_Base 中Phred 数值大于30的碱基占总体碱基的百分比
Clean_GC(%): 碱基G和C的数量总和占总的碱基数量的百分比
Effective(%):有效数据( Clean_Base )与原始数据( Raw_Base )的百分比
NonHost_Base(G):剔除宿主序列的Clean_Base
结果目录:
各样品QC结果详细信息见:result/01.QC/QC_summary.xls
4.2 组装
经过预处理后得到clean data,使用MEGAHIT组装软件进行组装分析(Assembly Analysis) (https://www.metagenomics.wiki/tools/assembly/megahit)。
对于单样品组装生成的Scaftigs,过滤掉500 bp以下的片段,并进行统计分析和后续基因预测(Karlsson et al., 2013; Li et al., 2014; Zeller et al., 2014; Sunagawa et al., 2015)。
表4.2 各样品组装结果scaftigs基本信息统计(>= 500 bp)
| SampleID | Total len(bp) | Scaftigs num | Average len(bp) | N50 len(bp) | N90 len(bp) | Max len(bp) |
|---|---|---|---|---|---|---|
| A1 | 165,447,841 | 69,857 | 2,368.38 | 10,965 | 708 | 478,642 |
| A2 | 213,570,341 | 111,489 | 1,915.62 | 4,092 | 656 | 646,982 |
| A3 | 235,682,388 | 121,901 | 1,933.39 | 3,883 | 666 | 646,981 |
| A4 | 144,363,110 | 50,624 | 2,851.67 | 15,391 | 785 | 478,624 |
| A5 | 183,960,238 | 86,142 | 2,135.55 | 9,028 | 657 | 412,322 |
| A6 | 249,341,826 | 138,844 | 1,795.84 | 3,263 | 642 | 646,982 |
| B1 | 319,519,425 | 166,683 | 1,916.93 | 3,518 | 672 | 481,015 |
| B2 | 132,072,777 | 89,750 | 1,471.56 | 1,905 | 596 | 818,692 |
| B3 | 121,835,665 | 76,525 | 1,592.10 | 2,583 | 610 | 819,022 |
| B4 | 247,135,836 | 148,448 | 1,664.80 | 2,709 | 626 | 488,277 |
| B5 | 92,541,796 | 54,664 | 1,692.92 | 2,594 | 638 | 819,154 |
| B6 | 185,528,748 | 117,961 | 1,572.80 | 2,107 | 630 | 498,778 |
| C1 | 200,981,089 | 105,572 | 1,903.73 | 4,001 | 648 | 818,507 |
| C2 | 265,057,699 | 148,610 | 1,783.58 | 3,019 | 654 | 460,710 |
| C3 | 309,520,243 | 169,297 | 1,828.27 | 3,159 | 664 | 437,747 |
| C4 | 170,647,887 | 87,284 | 1,955.09 | 4,858 | 655 | 488,694 |
| C5 | 225,060,591 | 140,455 | 1,602.37 | 2,417 | 620 | 646,981 |
| C6 | 317,578,445 | 168,282 | 1,887.18 | 3,369 | 673 | 479,734 |
显示注释
Total len(bp):组装得到的scaftigs的总长
Scaftigs num:组装得到的scaftigs总条数
Average len(bp):scaftigs的平均长度
N50 len(bp):将scaftigs按照长度进行排序,然后由长到短加和,当加和值达到scaftigs总长的50%时的scaftigs的长度值
N90 len(bp):将scaftigs按照长度进行排序,然后由长到短加和,当加和值达到scaftigs总长的90%时的scaftigs的长度值
Max len(bp):组装得到的最长scaftigs的长度值
结果目录:
按照长度500 bp进行过滤后,所有样品的Scaftigs序列:result/02.Assembly/Sample_Name/*.scaftigs.fa
按照长度500 bp进行过滤后,所有样品的Scaftigs统计表:result/02.Assembly/total.scaftigs.stat.info.xls
各样品对应的组装结果见:result/02.Assembly/Sample
各样品中Scaftigs长度的分布图见:result/02.Assembly/Sample/*.{svg,png}
4.3 基因预测及丰度分析
4.3.1 基因预测及丰度分析步骤
使用 MetaGeneMark (http://topaz.gatech.edu/GeneMark/)对各样品的scaftigs (>= 500 bp)进行 ORF 预测(Karlsson FH et al., 2012; Mende DR et al.,2012; Li J et al., 2014; Oh J et al., 2014; Qin N et al., 2014),并过滤掉预测结果中长度小于100 nt的信息(Qin J et al., 2010; Zhu W et al., 2010; Nielsen HB et al., 2014; Zeller G et al., 2014; Sunagawa S et al., 2015),均采用默认参数。
对ORF预测结果,采用CD-HIT软件(http://www.bioinformatics.org/cd-hit/)进行去冗余(Li W et al., 2006; Fu L et al., 2012),以获得非冗余的初始gene catalogue,此处将非冗余的连续基因编码的核酸序列称之为genes (Zeller G et al., 2014)(Li J et al., 2014; Qin N et al., 2014)。
使用Bowtie2将各样品的clean data 比对至初始gene catalogue,计算得到基因在各样品中比对上的reads数目(Qin J et al., 2010; Li J et al., 2014)。
过滤掉各个样品中reads数目<= 2的基因(Zeller G et al., 2014),获得最终用于后续分析的gene catalogue (unigene)。从比对上的reads数目及基因长度出发,计算得到各基因在各样品中的丰度信息(Cotillard A et al., 2013; Buchfink B et al., 2015; Villar E et al., 2015),如下公式所示:
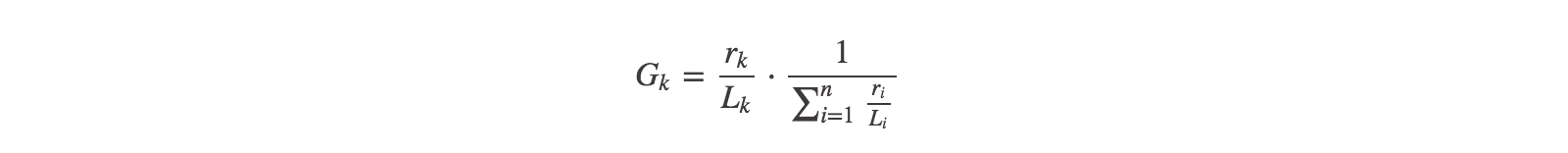
说明:r为比对上基因的reads 数目,L 为基因的长度
基于gene catalogue 中各基因在各样品中的丰度信息,进行基本信息统计,core-pan基因分析,样品间相关性分析,及基因数目韦恩图分析。
4.3.2 gene catalogue 基本信息统计
表4.3 Gene catalogue基本信息统计表
| ORFs_NO. | 1,319,089 |
|---|---|
| Integrity_start | 293,325(22.24%) |
| Integrity_end | 251,360(19.06%) |
| Integrity_none | 127,814(9.69%) |
| Integrity_all | 646,590(49.02%) |
| Total_Len.(Mbp) | 897.37 |
| Average_Len.(bp) | 680.3 |
| GC_Percent | 50.60 |
显示注释
ORFs_NO.:gene catalogue 中基因的数目
Integrity_start:只含有起始密码子的基因数目及百分比
Integrity_end:只含有终止密码子的基因数目及百分比
Integrity_none:没有起始密码子也没有终止密码子的基因数目及百分比
Integrity_all:完整基因(既有起始密码子也有终止密码子)数目的百分比
Total_Len. (Mbp):gene catalogue 中基因的总长,单位是百万
Average_Len. (bp):gene catalogue 中基因的平均长度
GC_Percent:预测的 gene catalogue 中基因的整体 GC 含量值
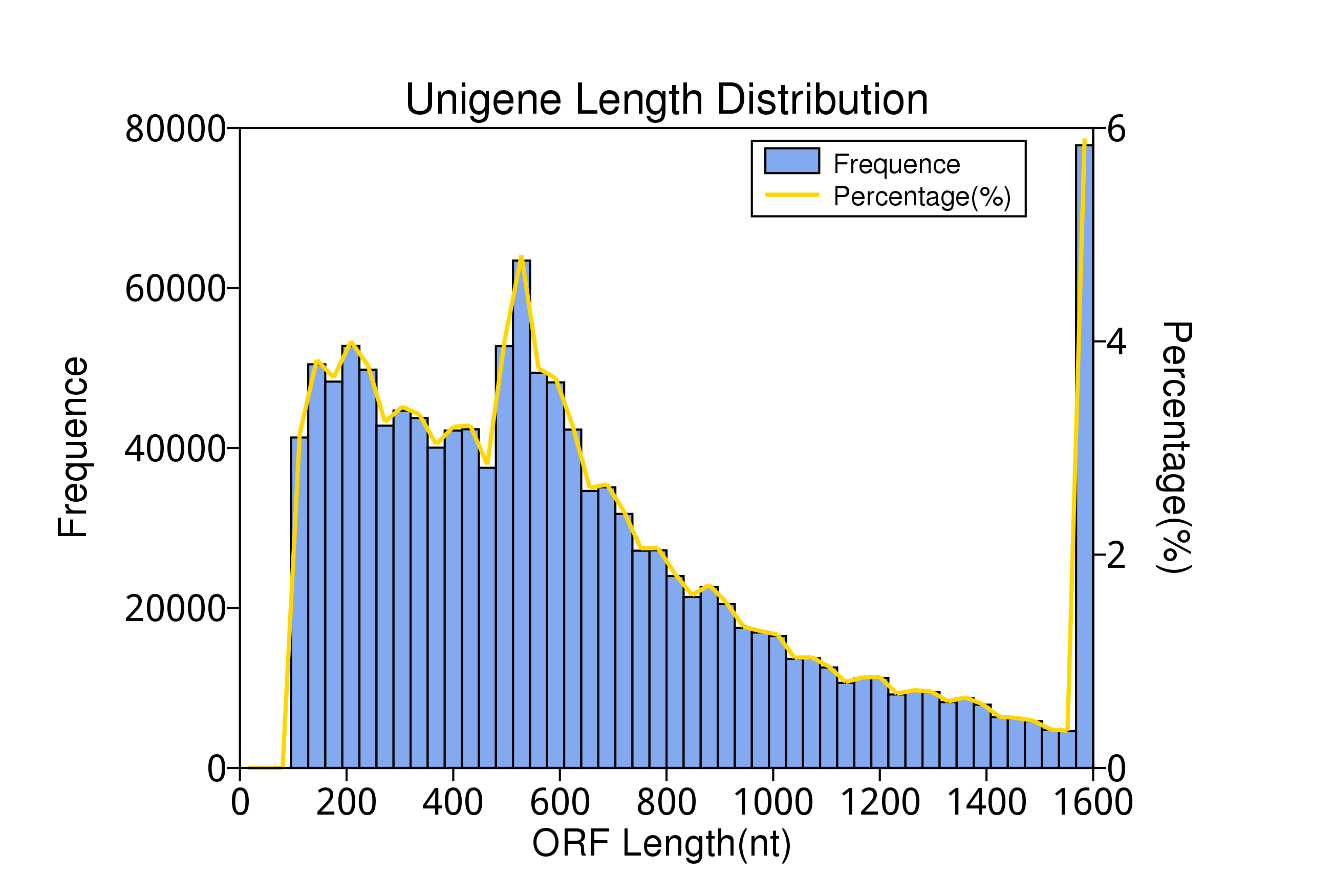
图4.1 Unigene长度分布统计
结果目录:
Unigene长度分布统计见:result/03.GenePredict/GenePredict/UniGenes/*.{svg,png}
4.3.3 Core-pan 基因分析
从基因在各样品中的丰度表出发,可以获得各样品的基因数目信息,通过随机抽取不同数目的样品,可以获得不同数目样品组合间的基因数目,由此构建和绘制的Core 和 Pan 基因的稀释曲线,图片展示如下:
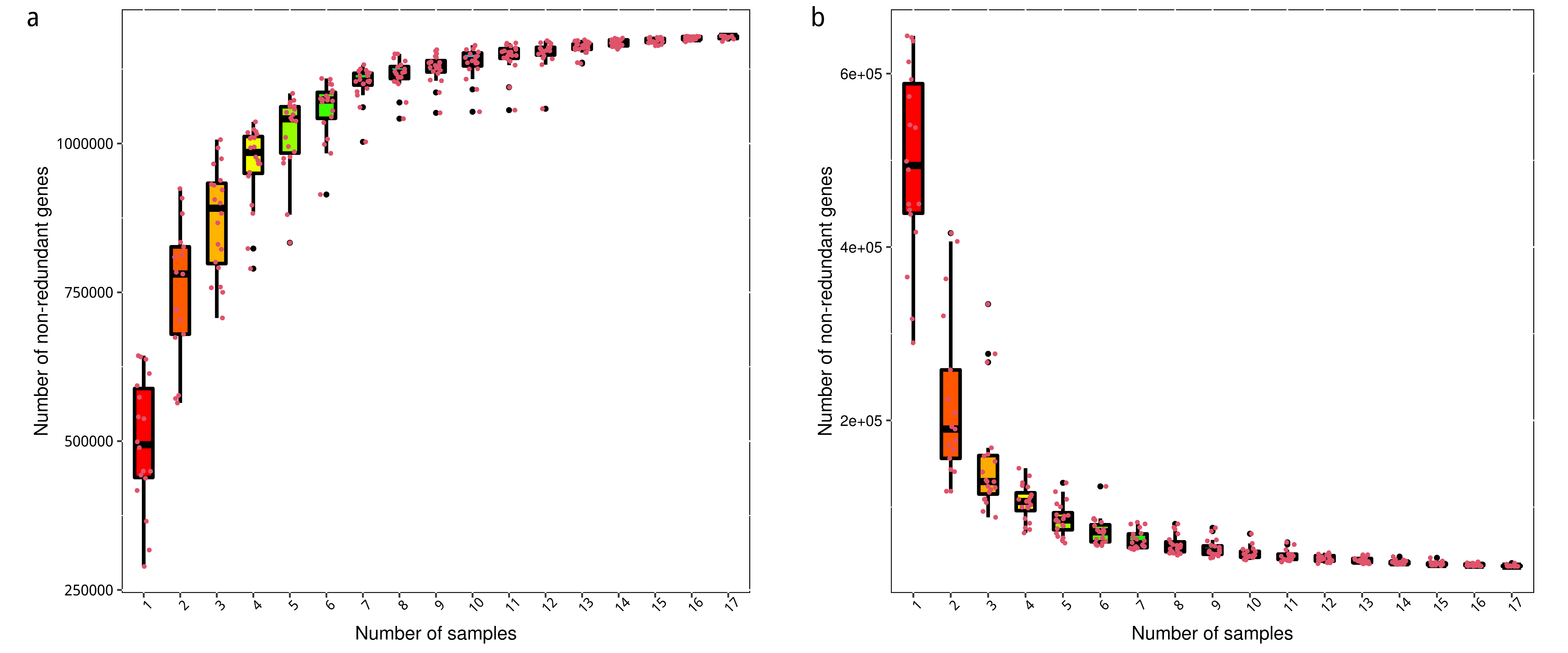
图4.2 Core-pan基因稀释曲线
说明:a) Pan基因稀释曲线;b) Core基因稀释曲线;横坐标表示抽取的样品个数;纵坐标表示抽取的样品组合的非冗余的基因数目
结果目录:
Core-pan 基因稀释曲线图见:result/03.GenePredict/GeneStat/core_pan/*.{png,pdf}
4.3.4 基于基因数目的样品间相关性分析
生物学重复是任何生物学实验所必须的,高通量测序技术也不例外。样品间基因丰度相关性是检验实验可靠性和样本选择合理性的重要指标。相关系数越接近1,表明样品之间基因丰度模式的相似度越高。
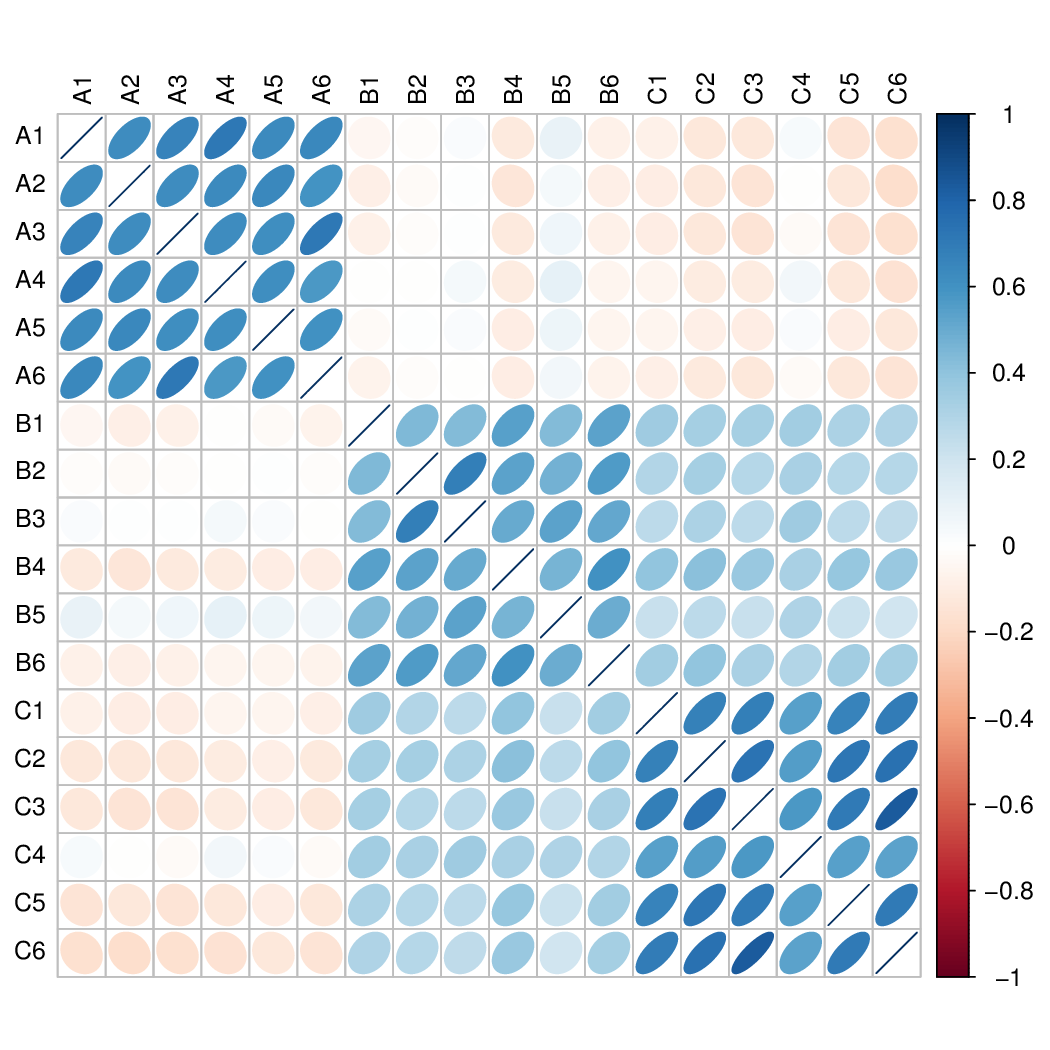
图4.3 样品间相关系数热图
说明:图中,不同颜色代表 spearman 相关系数的高低;相关系数与颜色间的关系见右侧图例;颜色越深代表样品间相关系数的绝对值越大;椭圆向右偏表明相关系数为正,左偏为负;椭圆越扁说明相关系数的绝对值越大
结果目录:
样品间相关系数热图见:result/03.GenePredict/GeneStat/correlation/*.{png,pdf}
4.3.5 基因数目差异分析
为了考察组与组间的基因数目差异情况,绘制了组间基因数目差异箱图,展示结果如下:
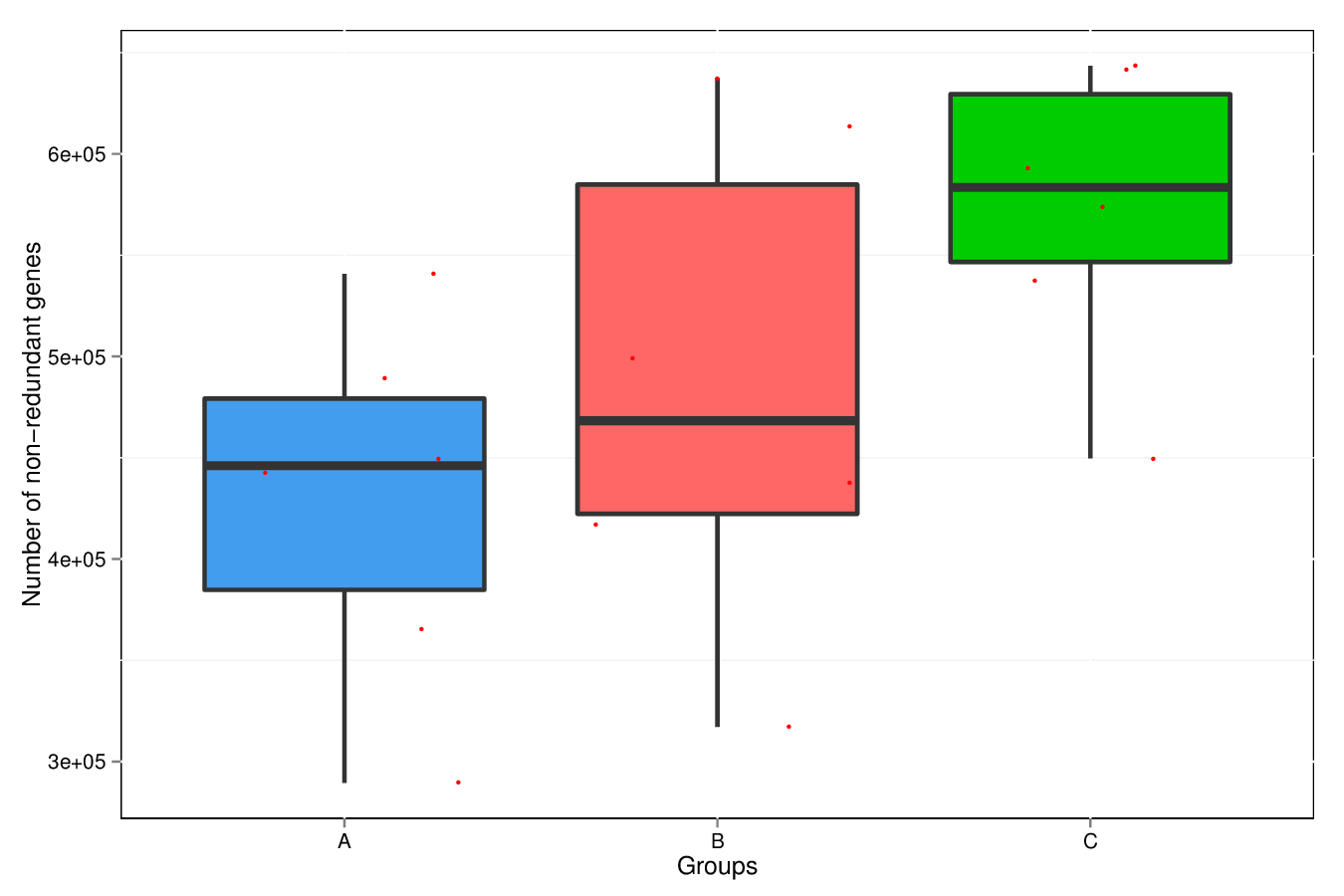
图4.4 组间基因数目差异箱图
说明:横坐标为各个分组信息;纵坐标为基因数目
结果目录:
组间基因数目差异箱图见: result/03.GenePredict/GeneStat/genebox_group1/*.{png,pdf}
4.4 物种与功能注释
4.4.1 Micro_NR物种注释
目前常用的功能数据库主要有:
NR数据库(https://www.ncbi.nlm.nih.gov/):非冗余蛋白质的氨基酸序列数据库,包含SwissProt、PIR (Protein Information Resource)、PRF (Protein Research Foundation)、PDB (Protein Data Bank)蛋白质数据库非冗余的数据以及从GenBank 和RefSeq 的CDS 数据翻译来的蛋白质数据。
Micro_NR数据库:从NR数据库中抽提出的细菌(Bacteria)、真菌(Fungi)、古菌(Archaea)和病毒(Viruses)四部分微生物序列用作微生物分析。
4.4.1.1 物种注释基本步骤
使用DIAMOND软件(https://github.com/bbuchfink/diamond/) (Buchfink B et al., 2015),将unigene与从NCBI的NR数据库(https://www.ncbi.nlm.nih.gov/)中抽提出的细菌(Bacteria)、真菌(Fungi)、古菌(Archaea)和病毒(Viruses)序列进行比对(Karlsson FH et al., 2013)。
对于每一条序列的比对结果,选取evalue <= 10e-5的结果,由于每一条序列可能有多个比对结果,采取 LCA 算法(应用于MEGAN软件的系统分类(https://en.wikipedia.org/wiki/Lowest_common_ancestor) 来确定该序列的物种注释信息(Huson DH et al., 2011)。
从LCA注释结果及基因丰度表出发,获得各个样品在各个分类层级(界门纲目科属种)上的丰度信息及基因数目表,对于某个物种在某个样品中的丰度,等于注释为该物种的基因丰度的加和(Karlsson FH et al., 2012; Li J et al., 2014; Feng Q et al., 2015);对于某个物种在某个样品中的基因数目,等于在注释为该物种的基因中,丰度不为0的基因数目。
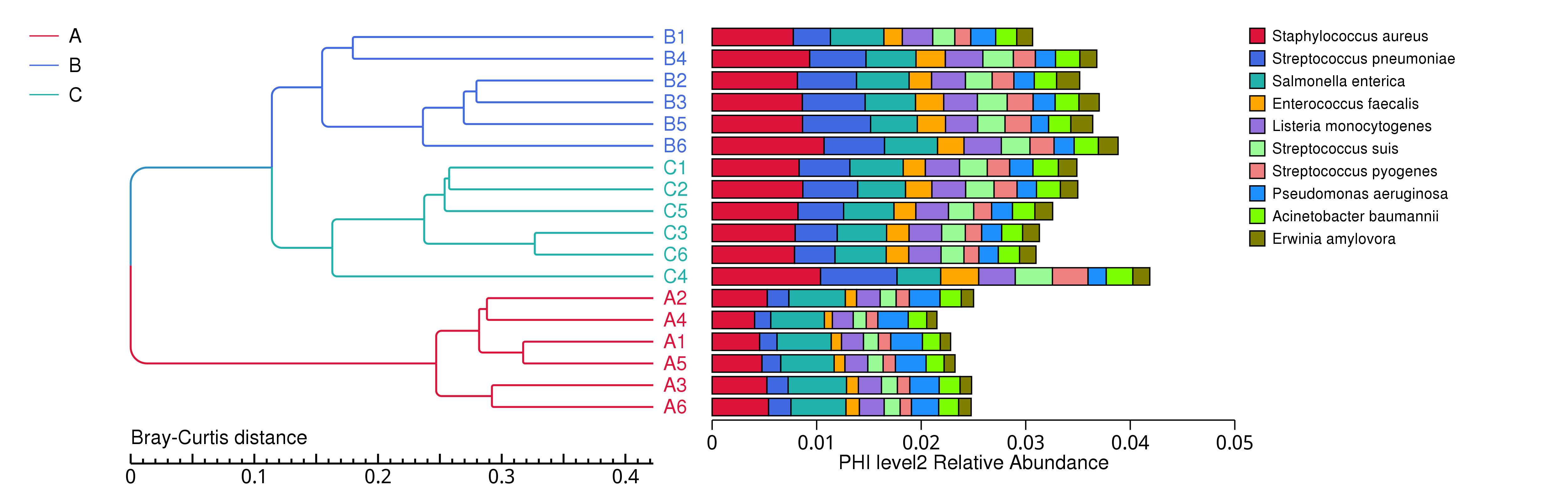
4.4.1.2 物种相对丰度概况
为了综合而直观的展示各样品中,不同分类层级的物种相对丰度,我们采用 Krona对物种注释结果进行可视化展示(Ondov et al., 2011),Krona 示例图如下所示:
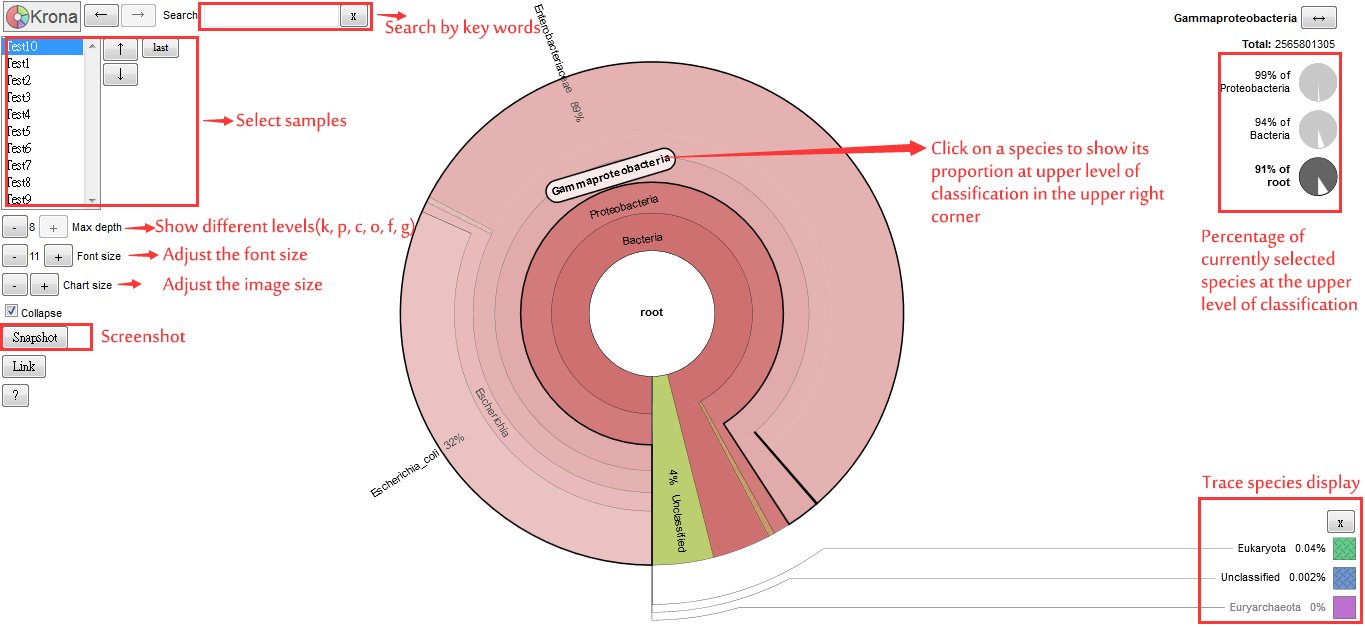
图4.5 使用 Krona 对物种注释结果进行展示(示例图)
说明:图中,圆圈从内到外依次代表不同的分类级别(界门纲目科属种);扇形的大小代表不同物种的相对丰度比例;更多详细的信息请参考KRONA 展示结果详解(https://github.com/marbl/Krona/wiki/)
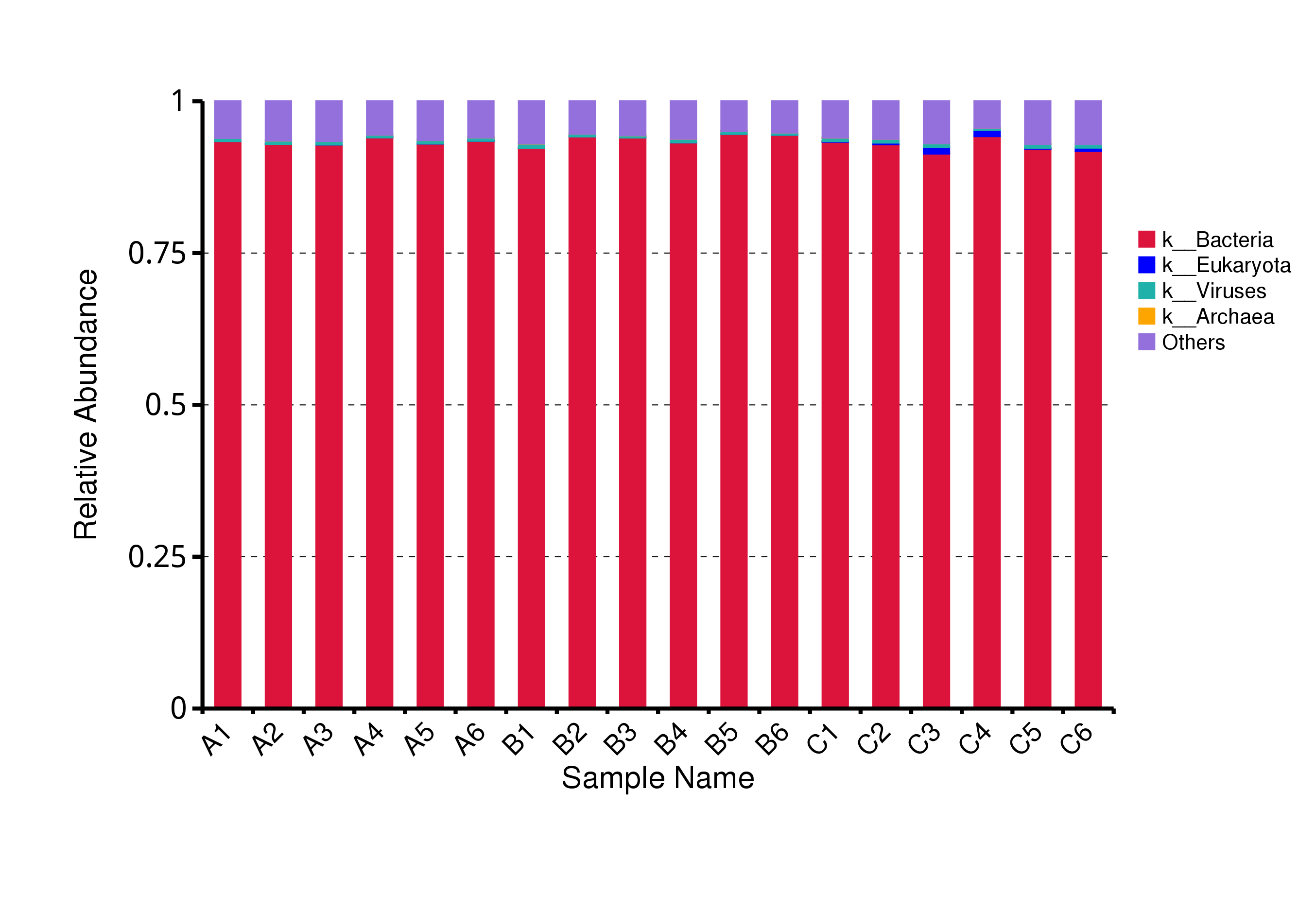
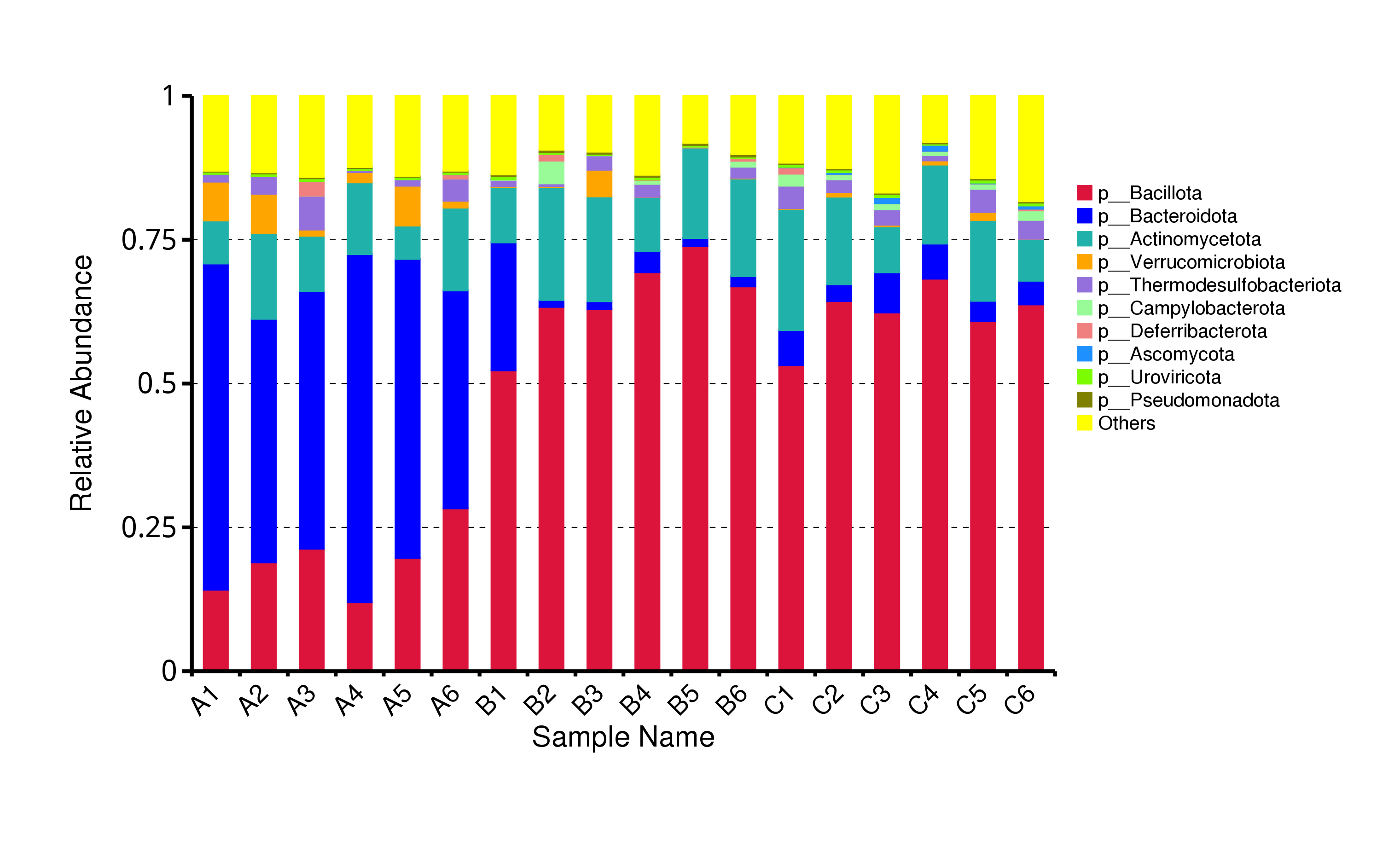
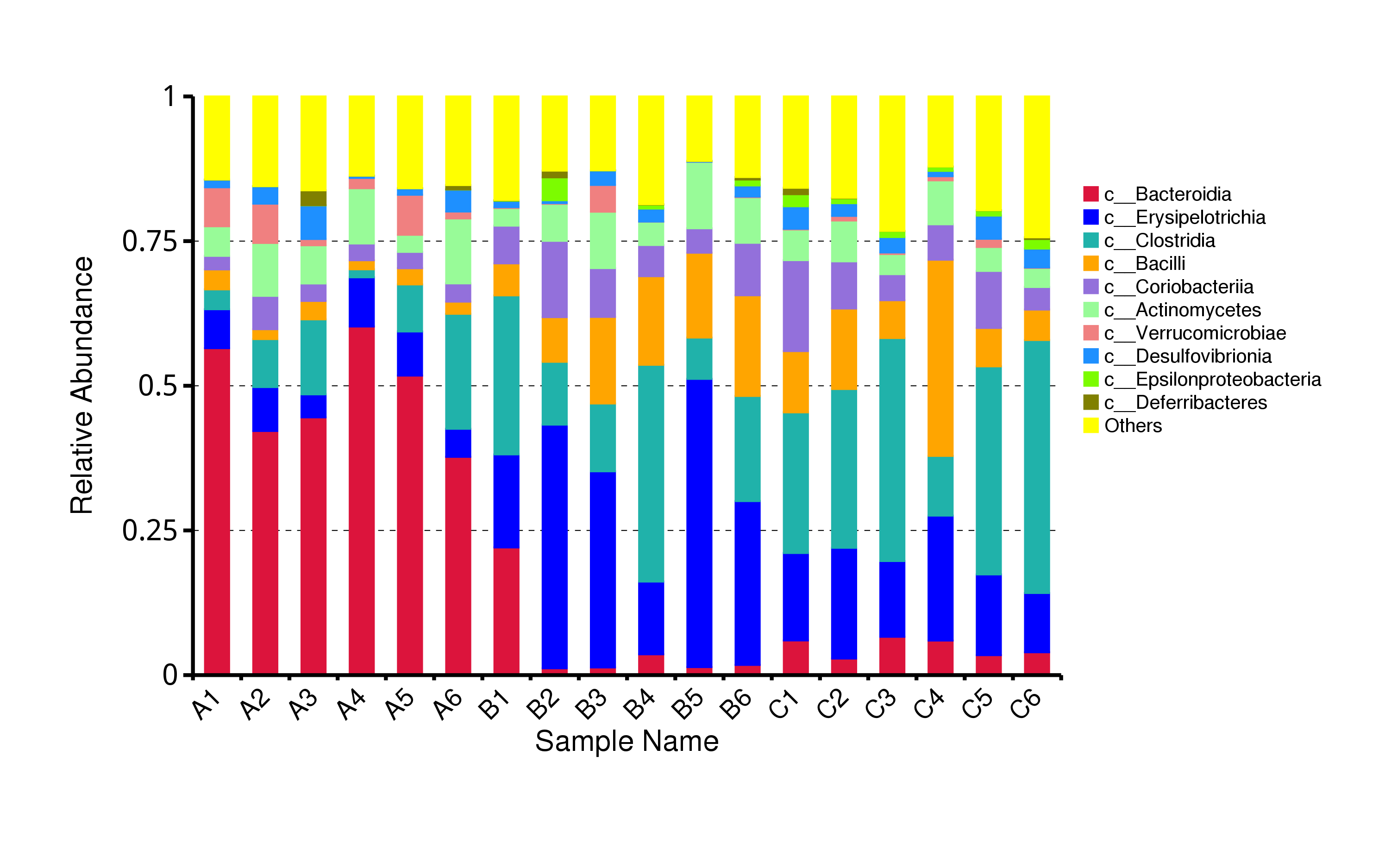
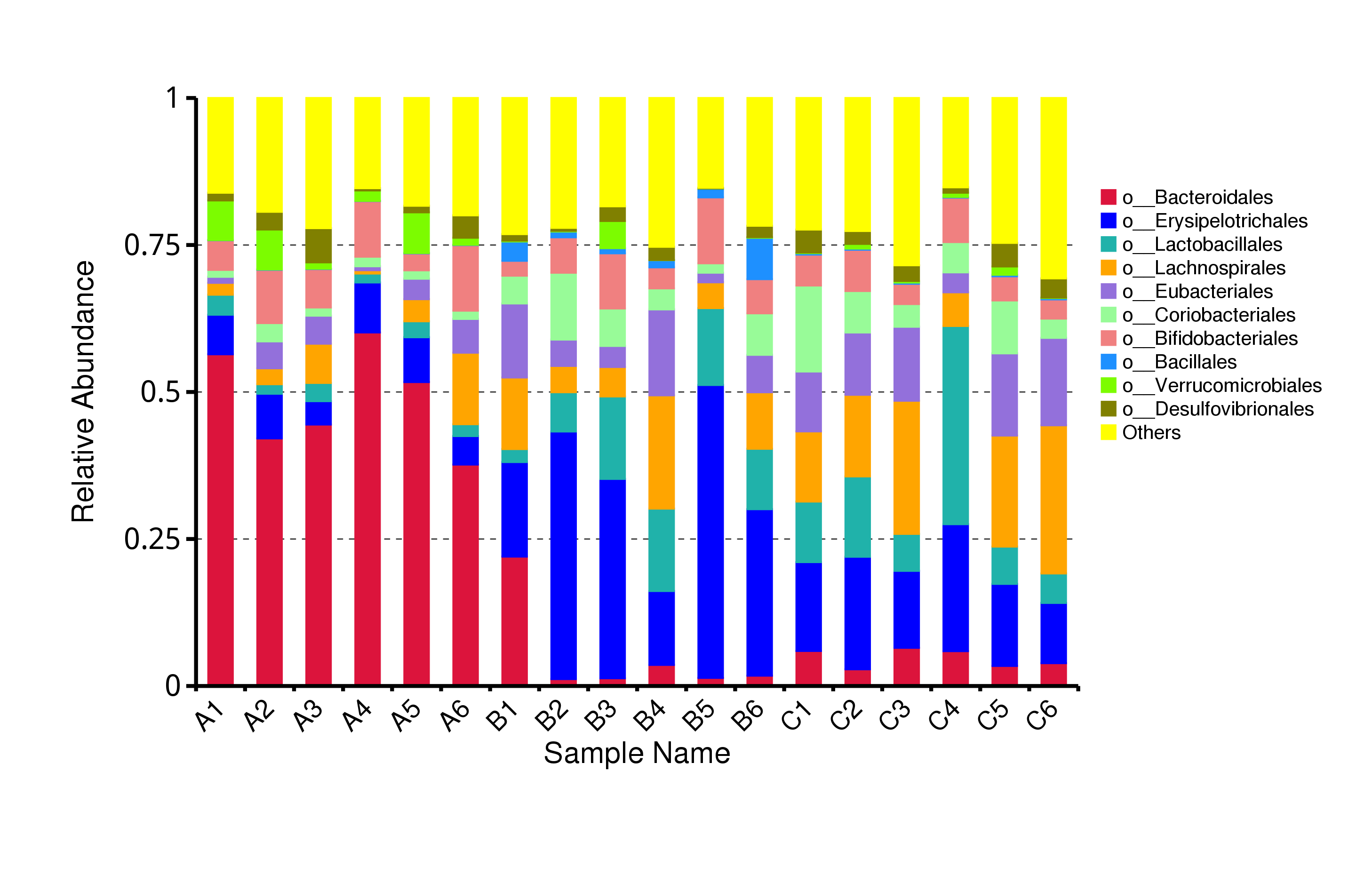
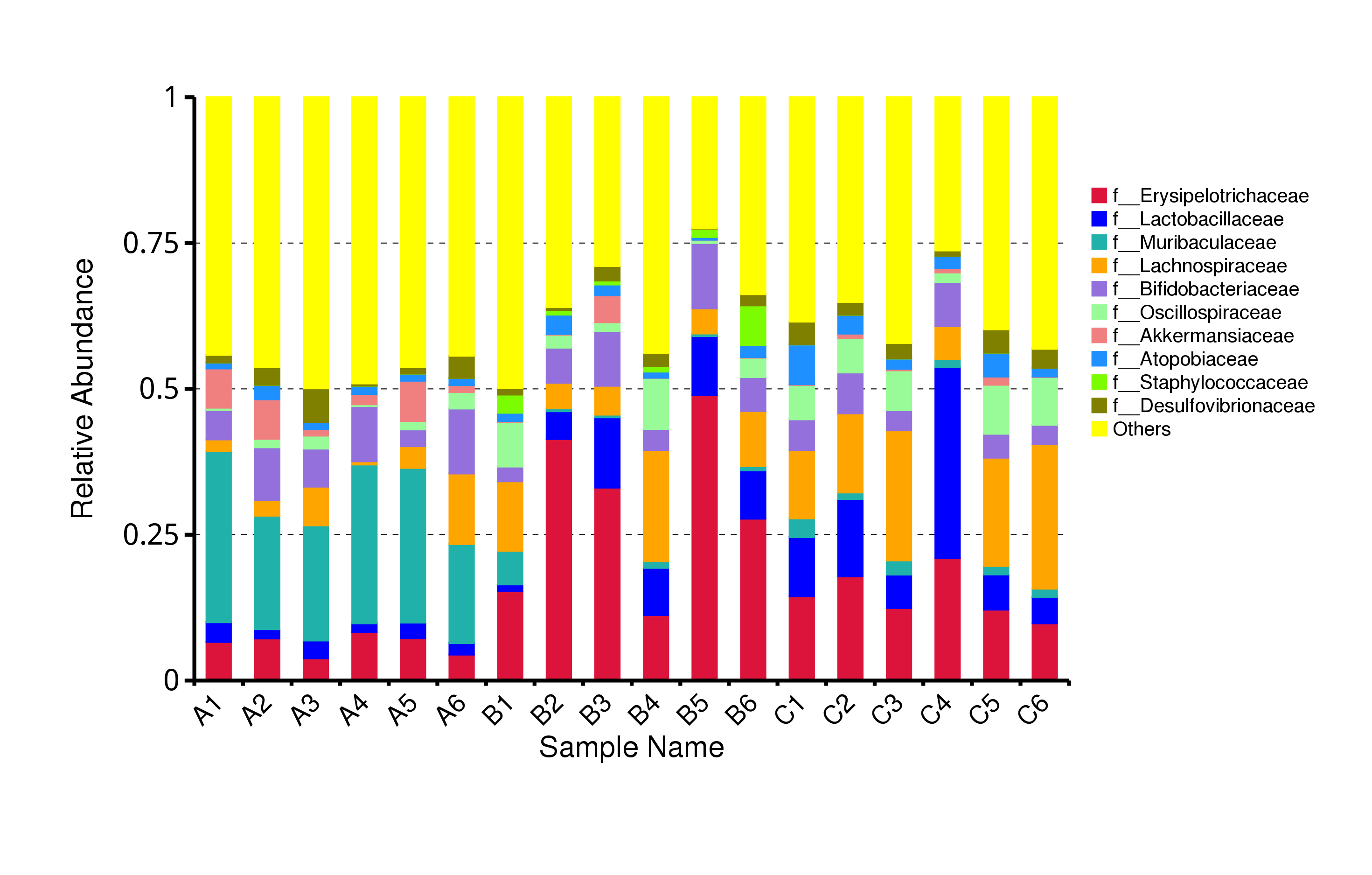
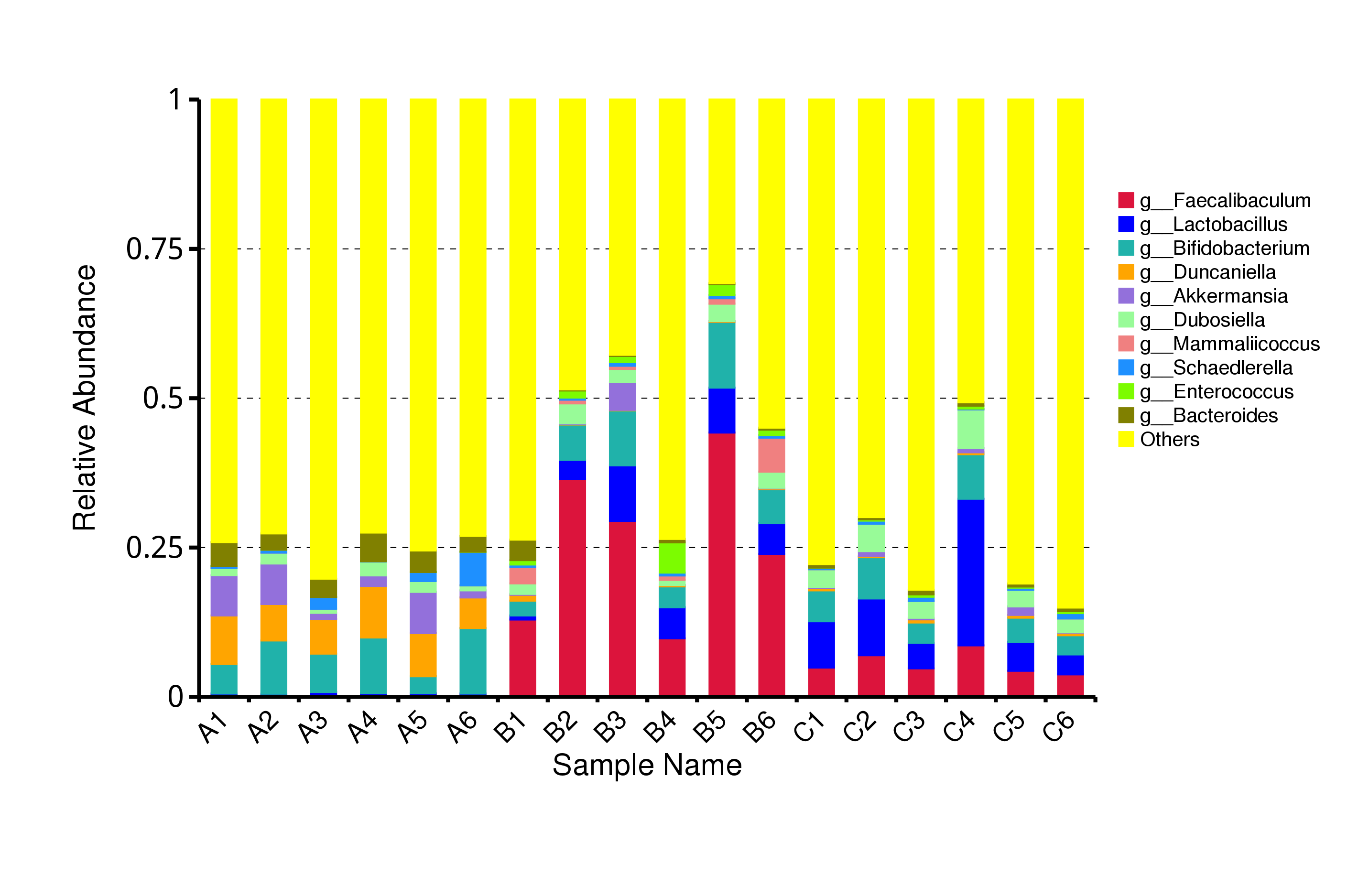
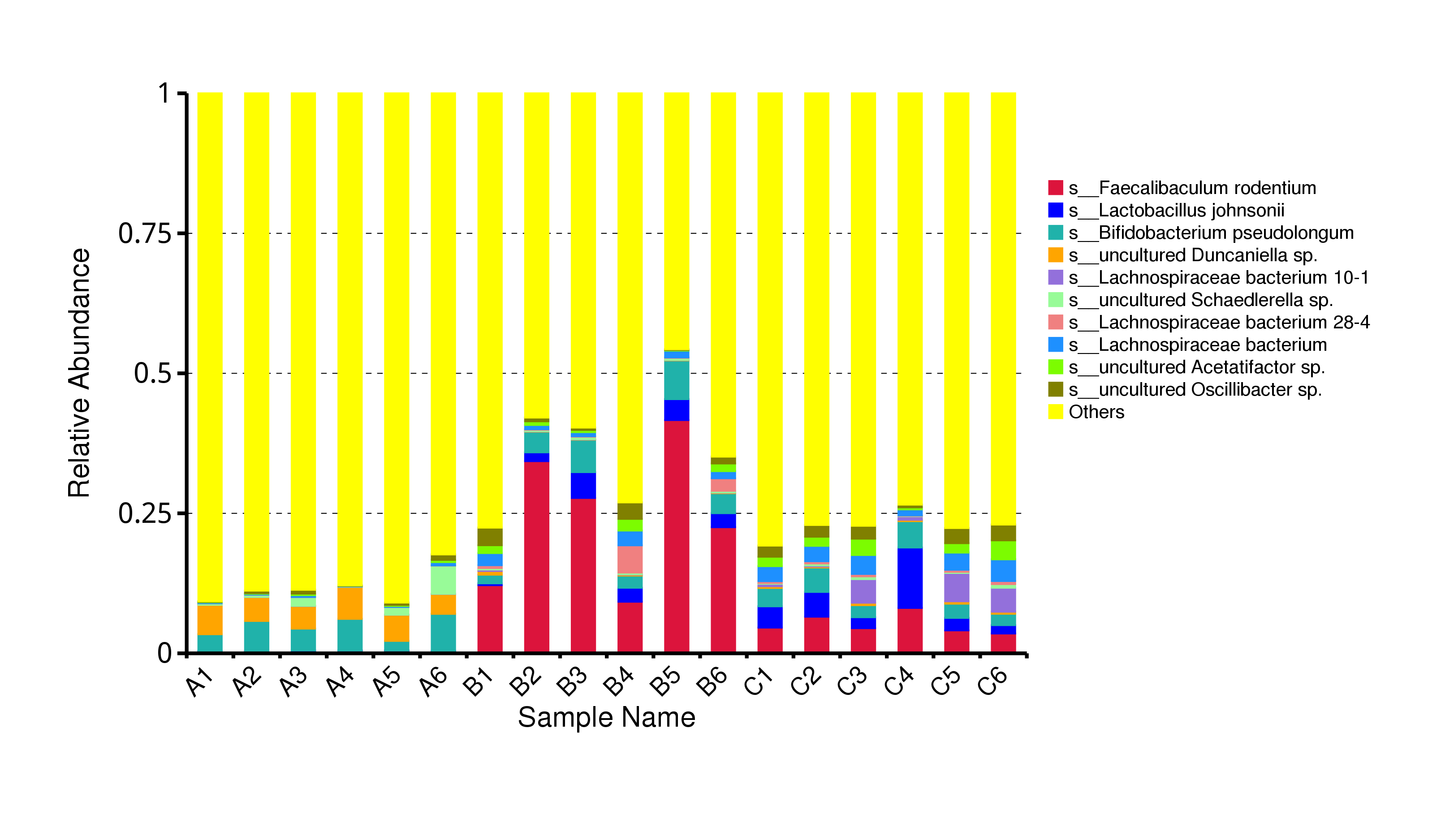
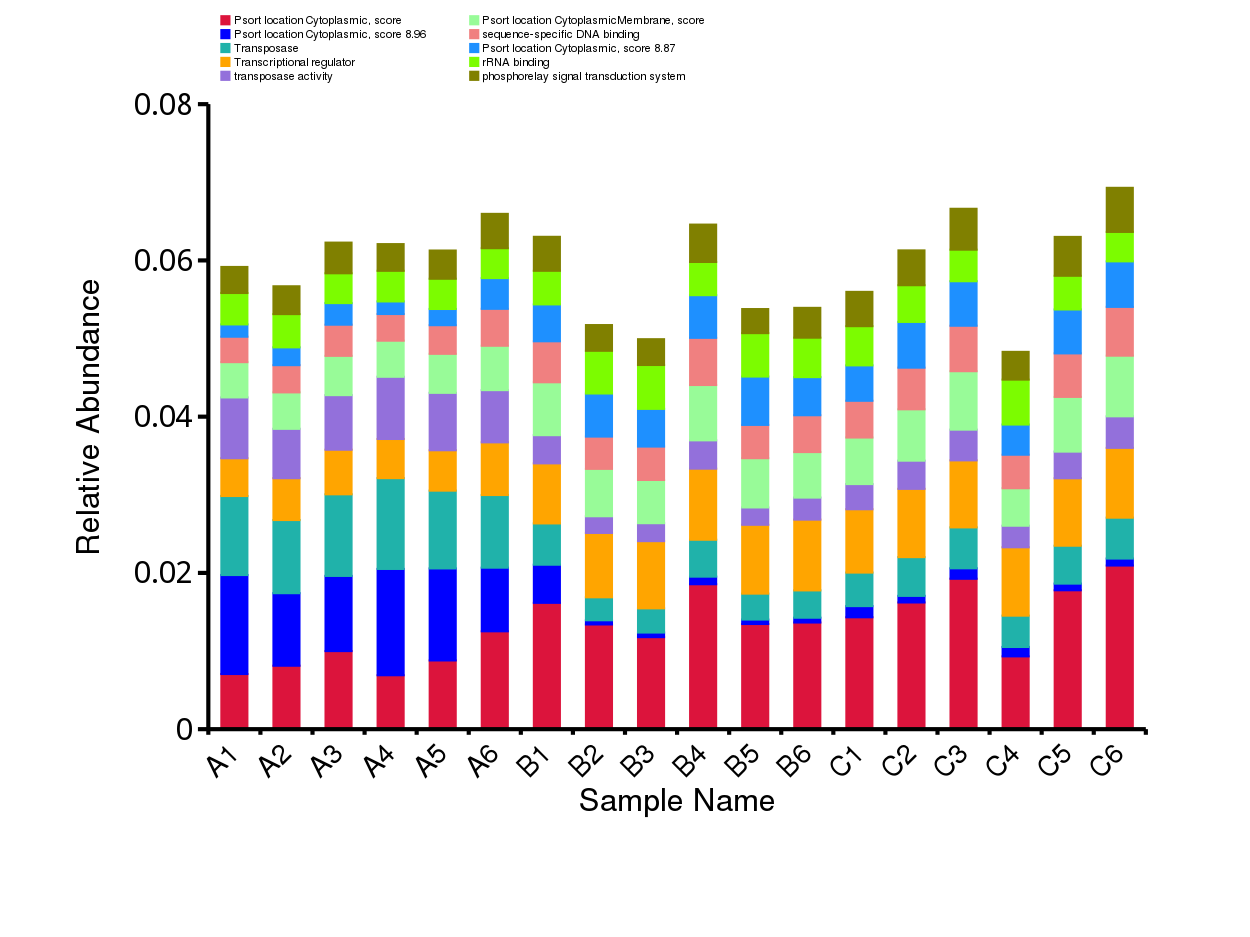
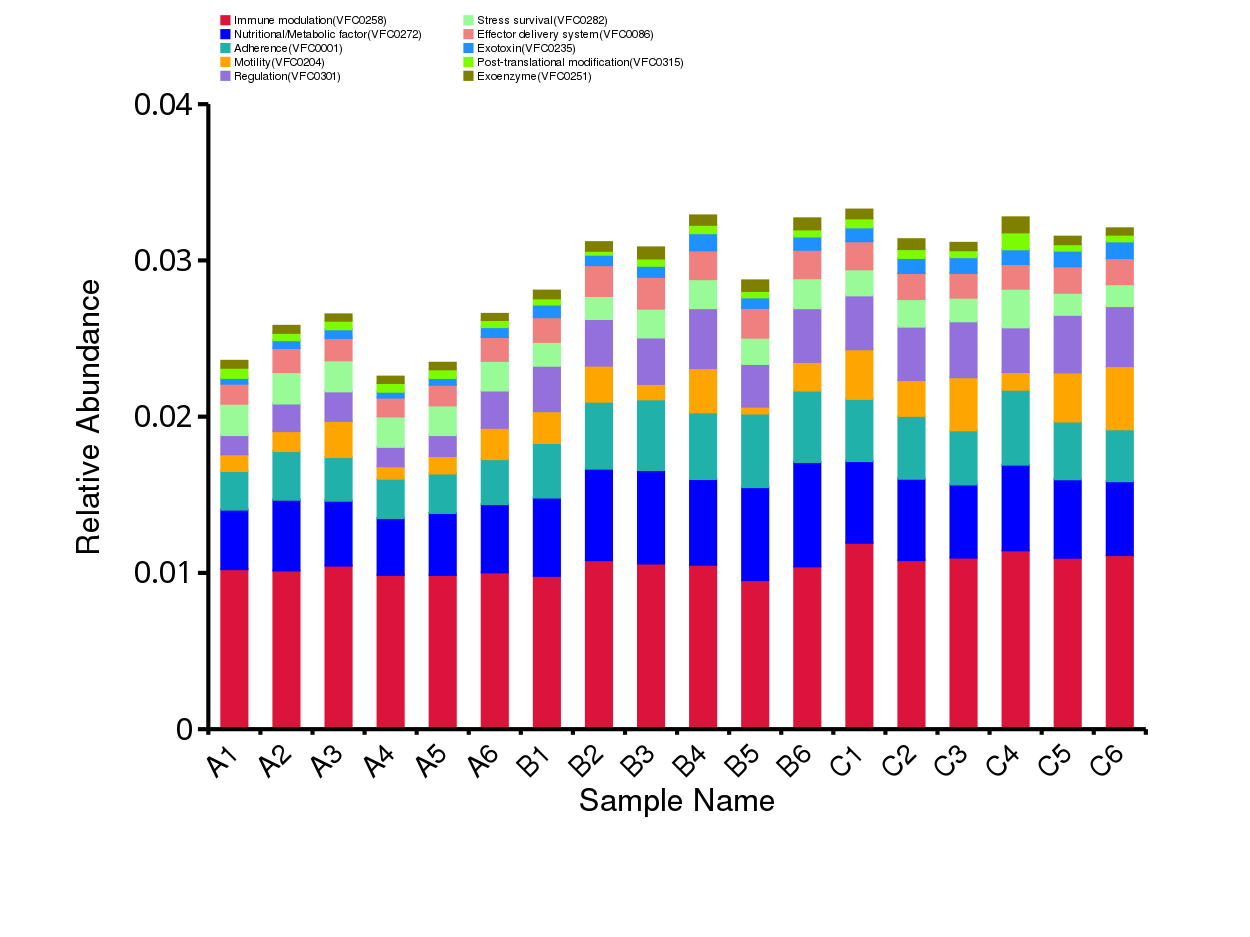
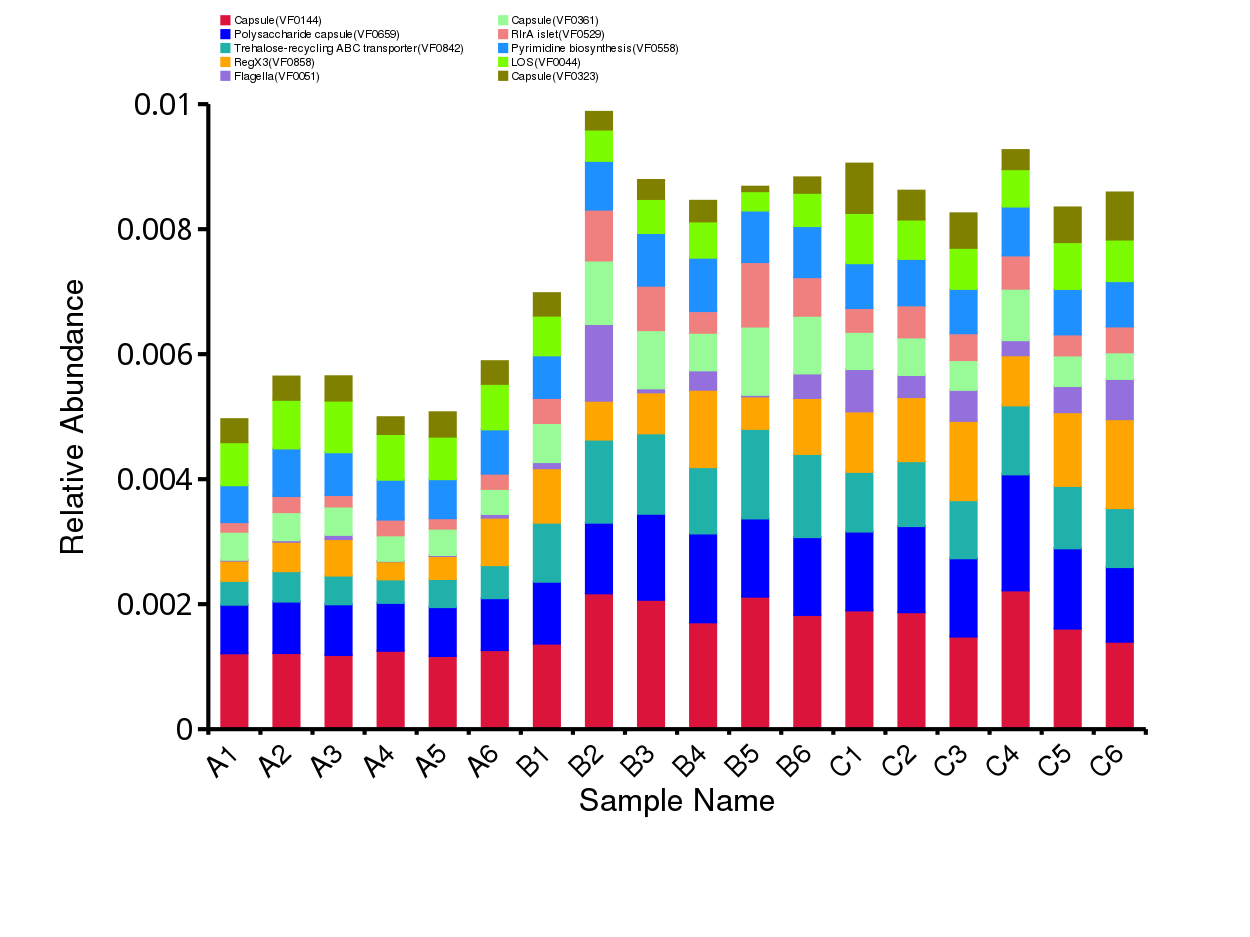
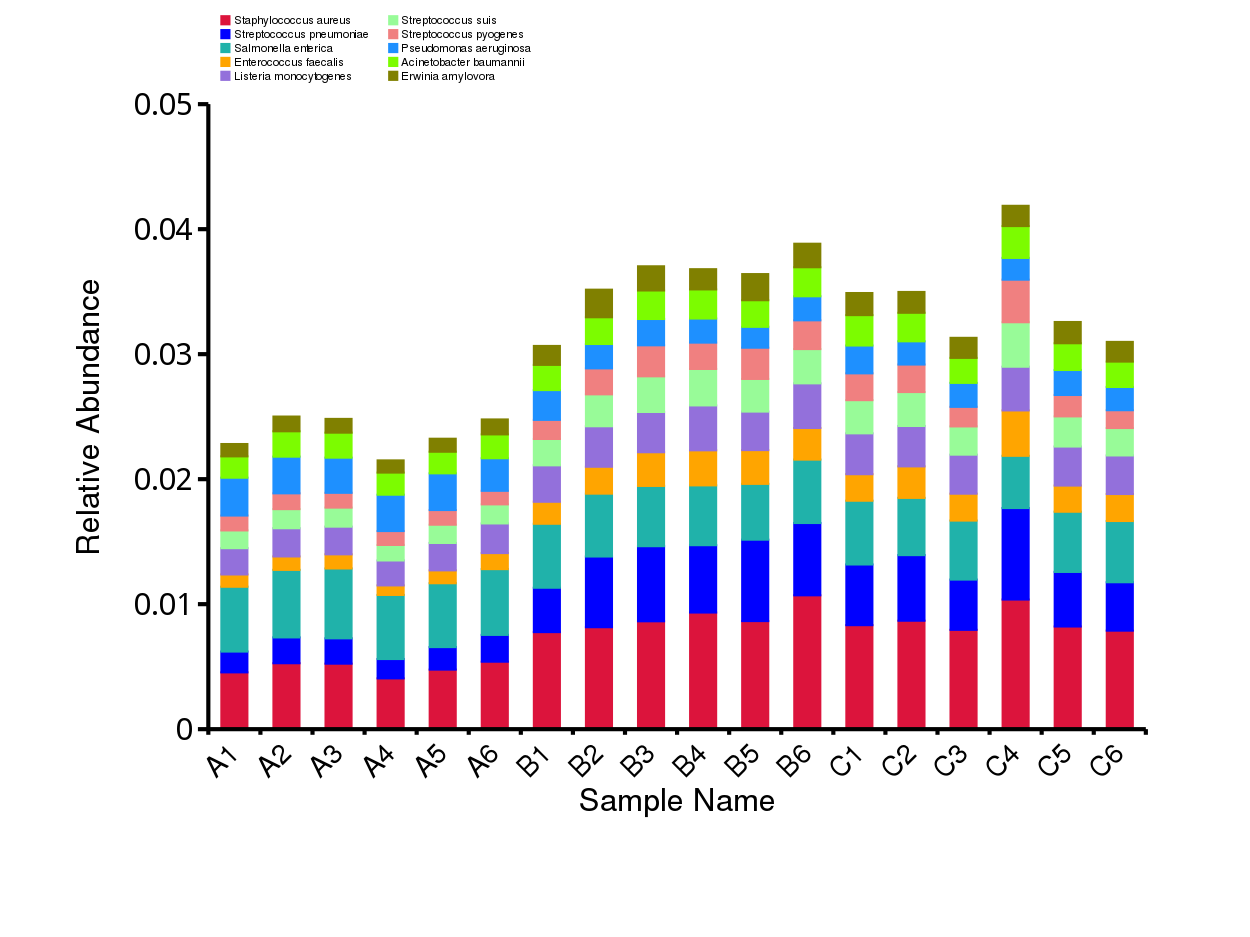
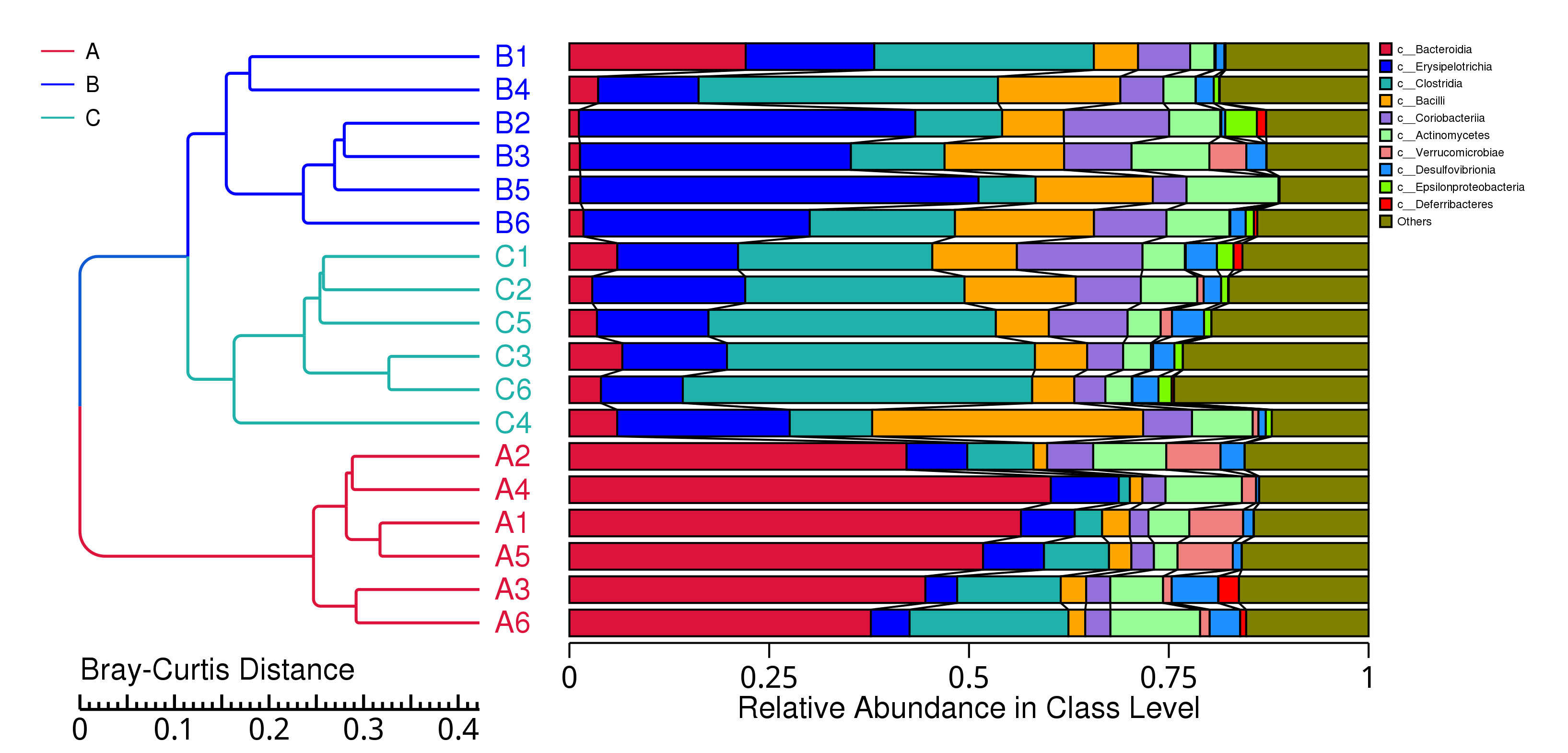
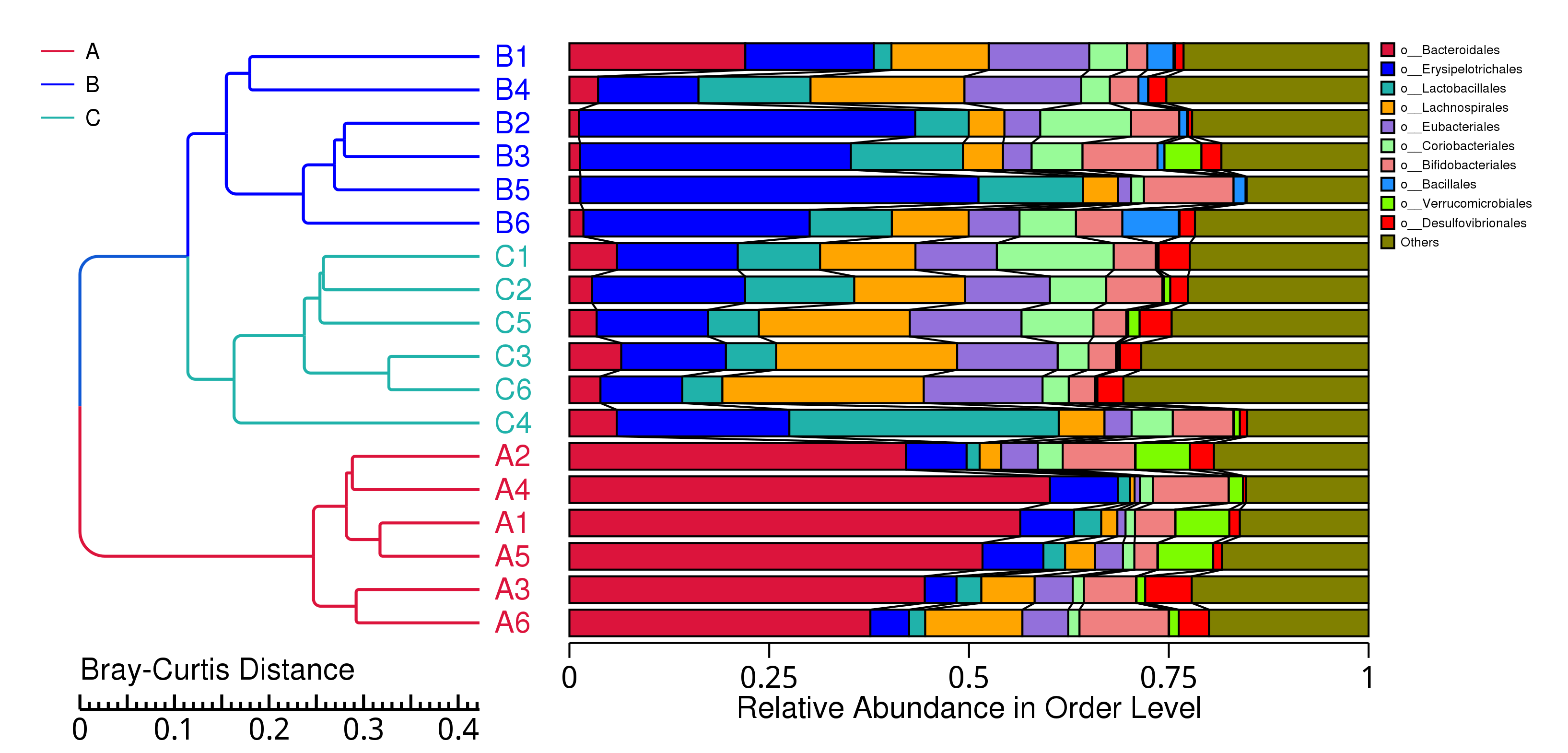
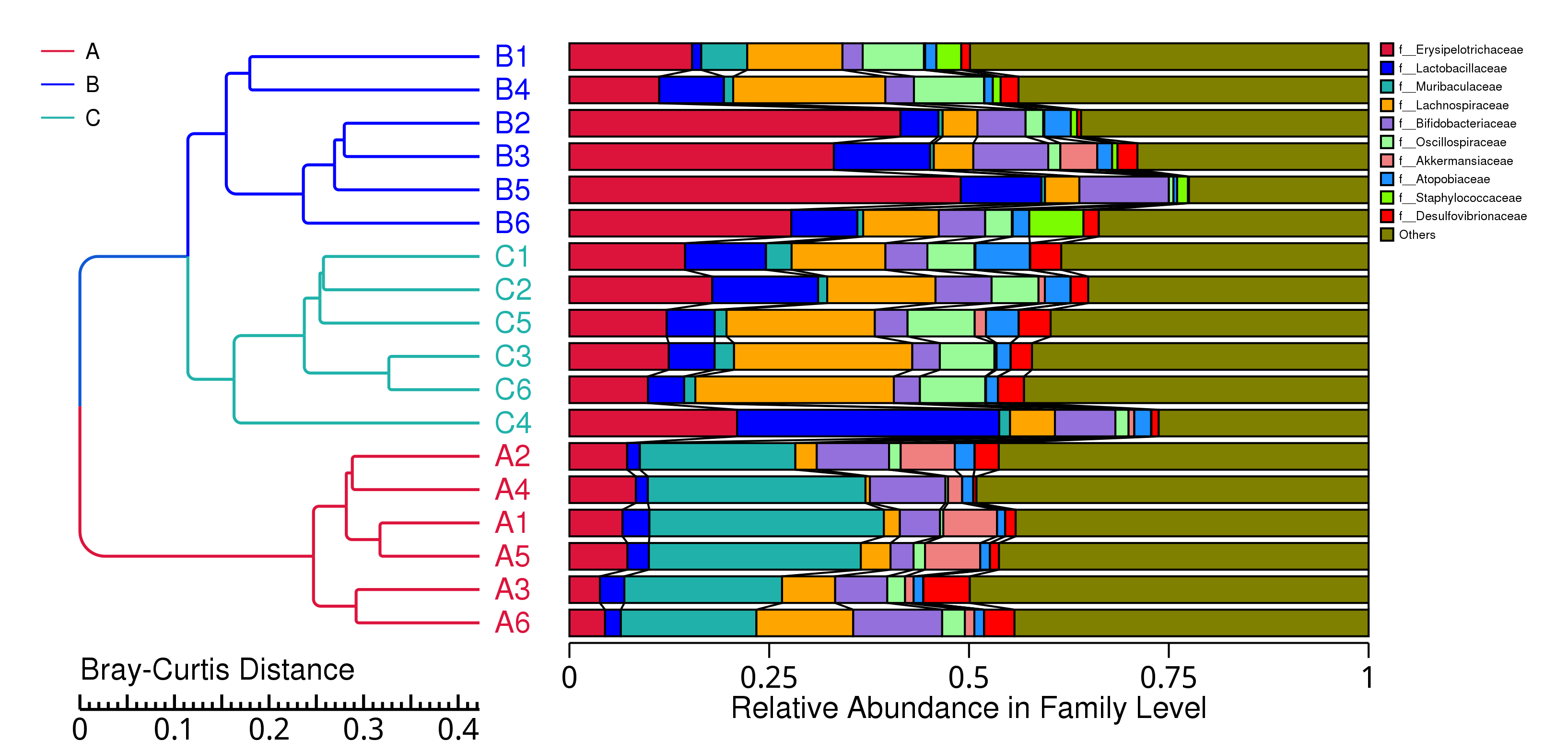
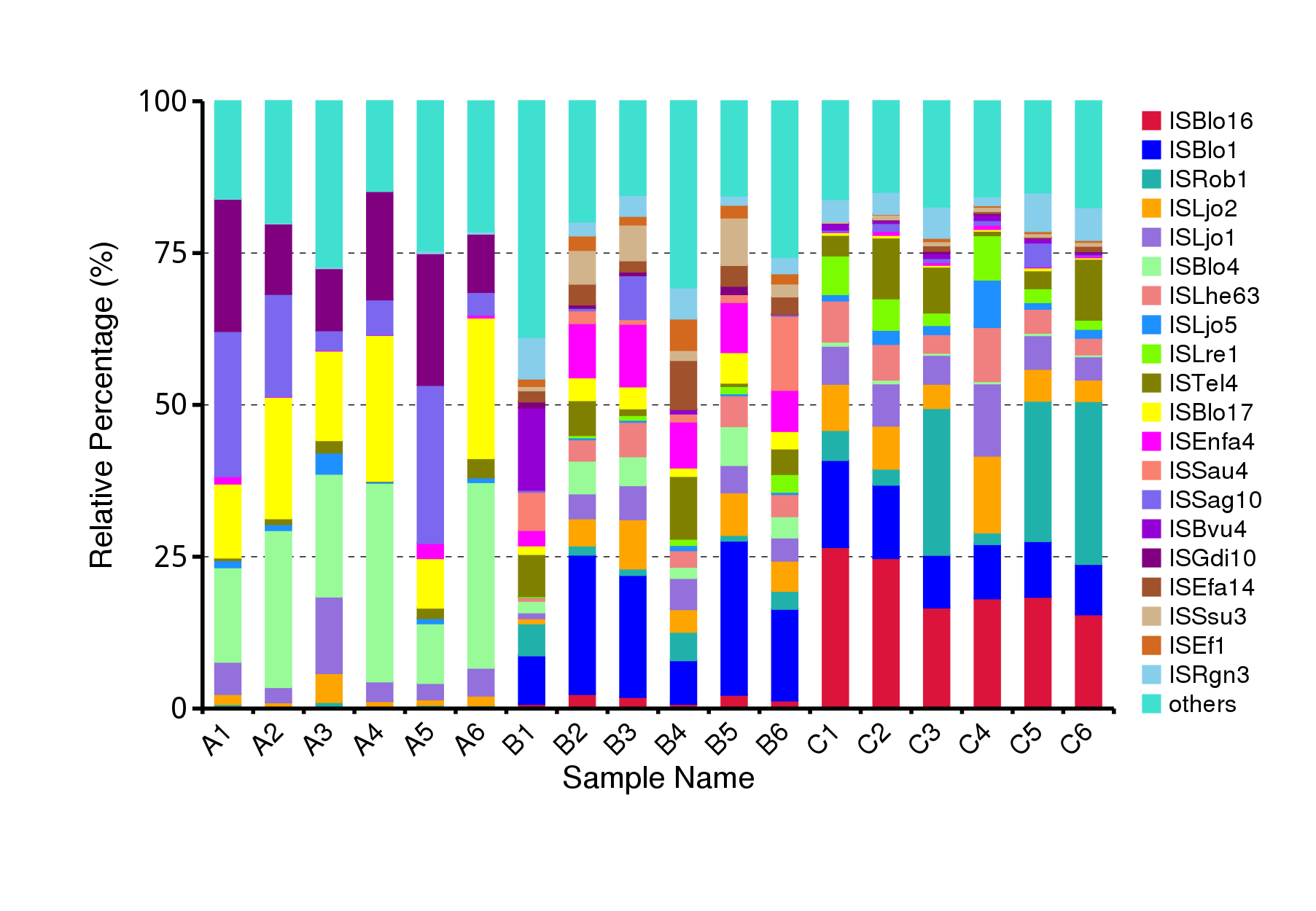
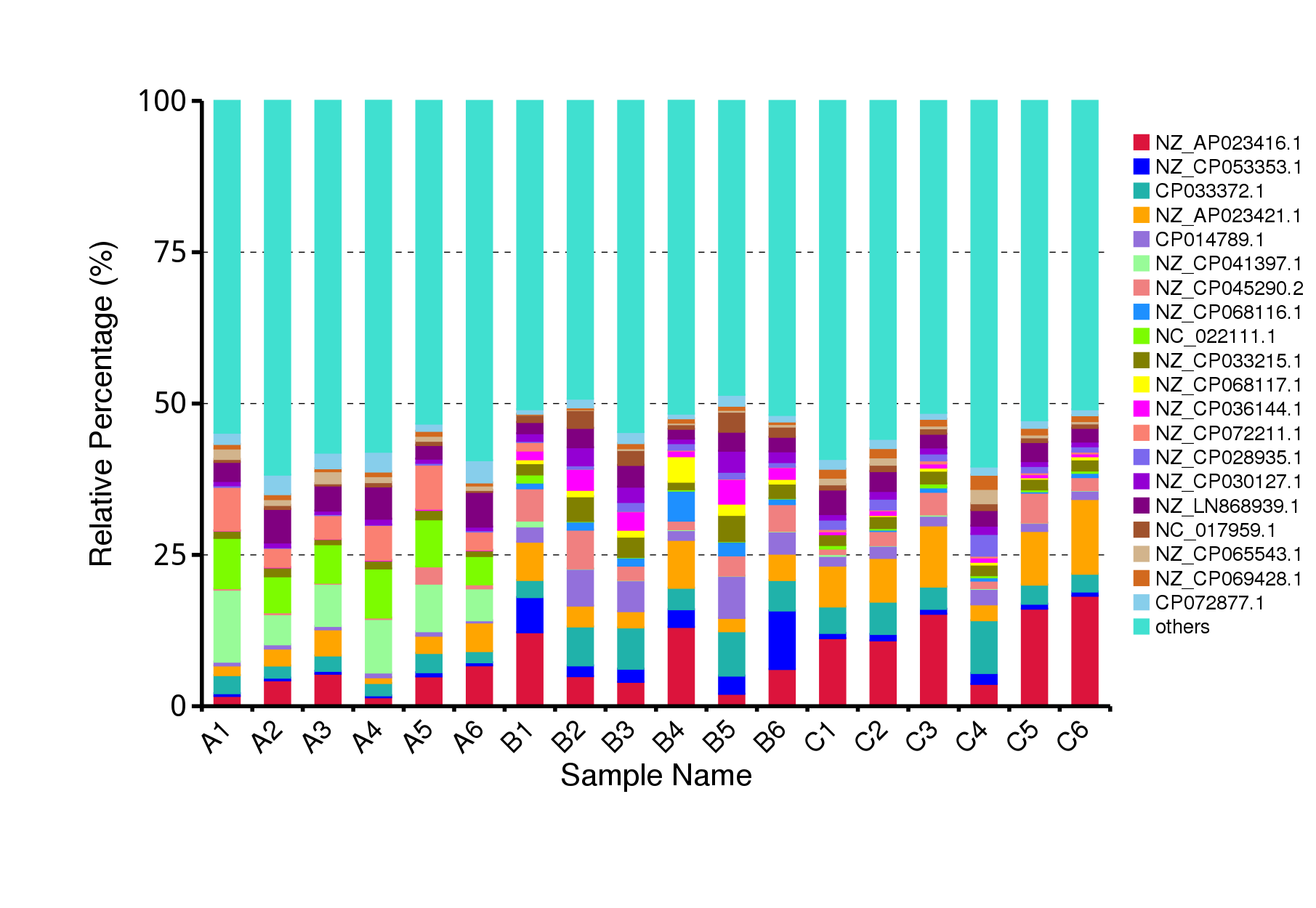
从不同分类层级的相对丰度表出发,选取出在各样品(组)中的最大相对丰度排名前 10 的物种,并将其余的物种设置为 Others,绘制出各样品对应的物种注释结果在不同分类层级上的相对丰度柱形图。
图4.6 物种水平的物种相对丰度柱形图
说明:每个层级的相对丰度柱形图,横向表示样品名称,纵向表示注释到某类型的物种的相对丰度比例;各颜色区块对应的物种类别见右侧图例
结果目录:
Krona展示结果见:result/04.Annotation/MicroNR/Krona/taxonomy.krona.html
top10物种相对丰度柱形图见:result/04.Annotation/MicroNR/{Top_sample,Top_group1},包括界门纲目科属种(Kingdom、 Phylum、 Class、 Order、 Family、 Genus、Species)7个分类级别的结果
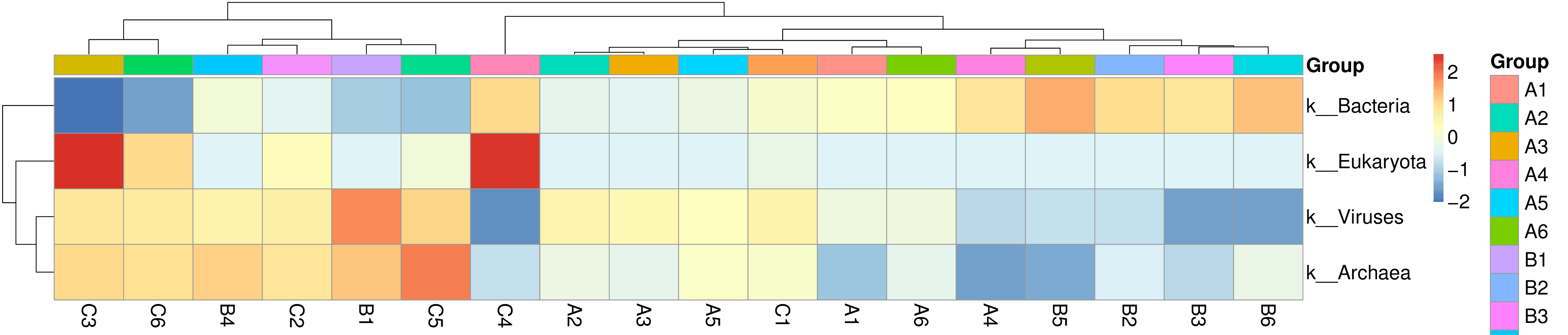
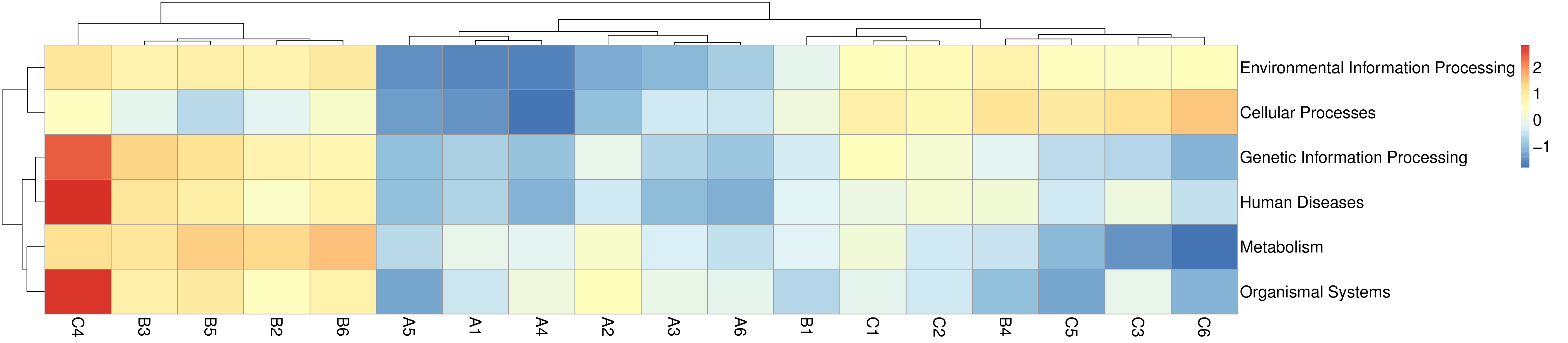
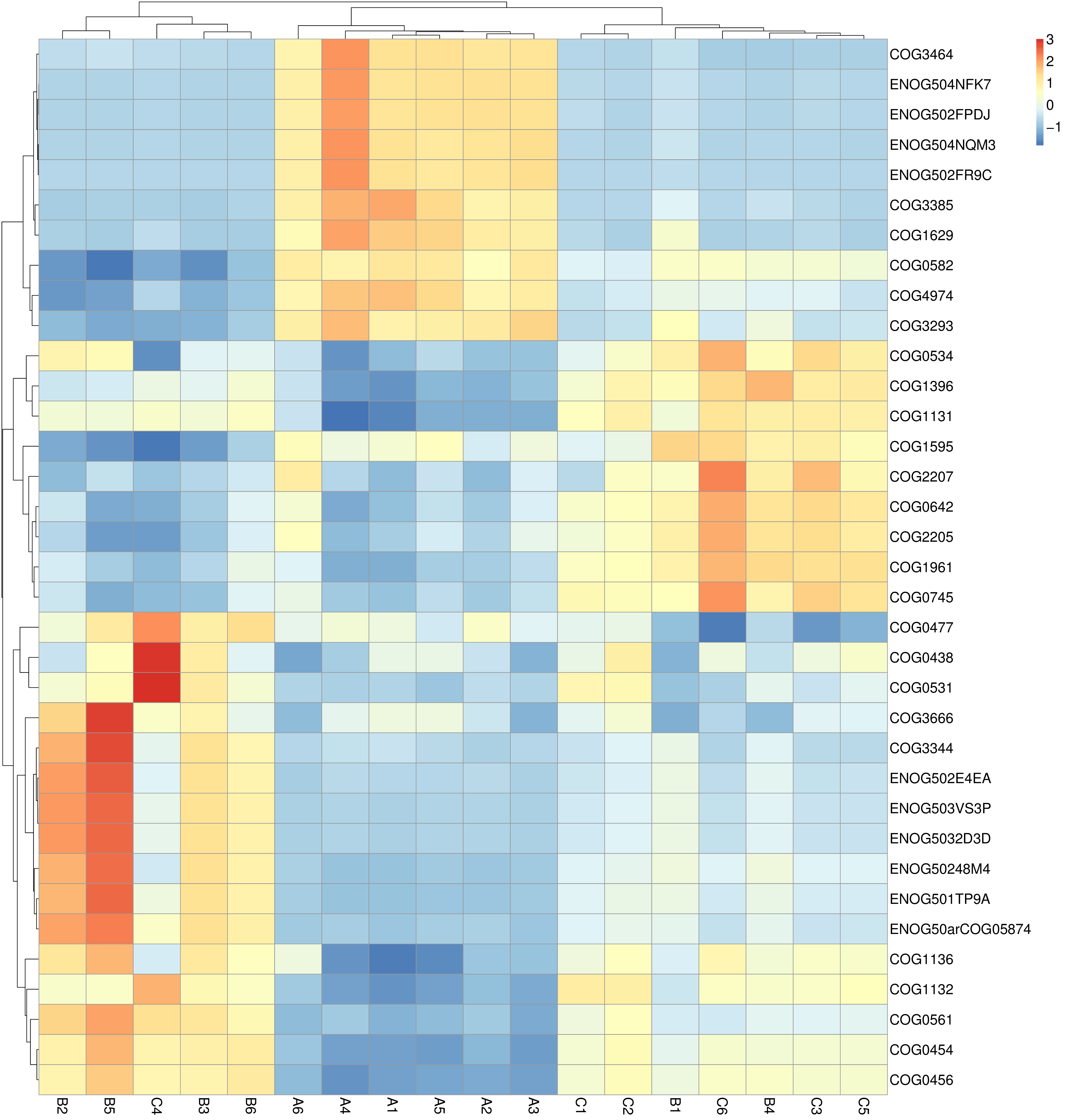
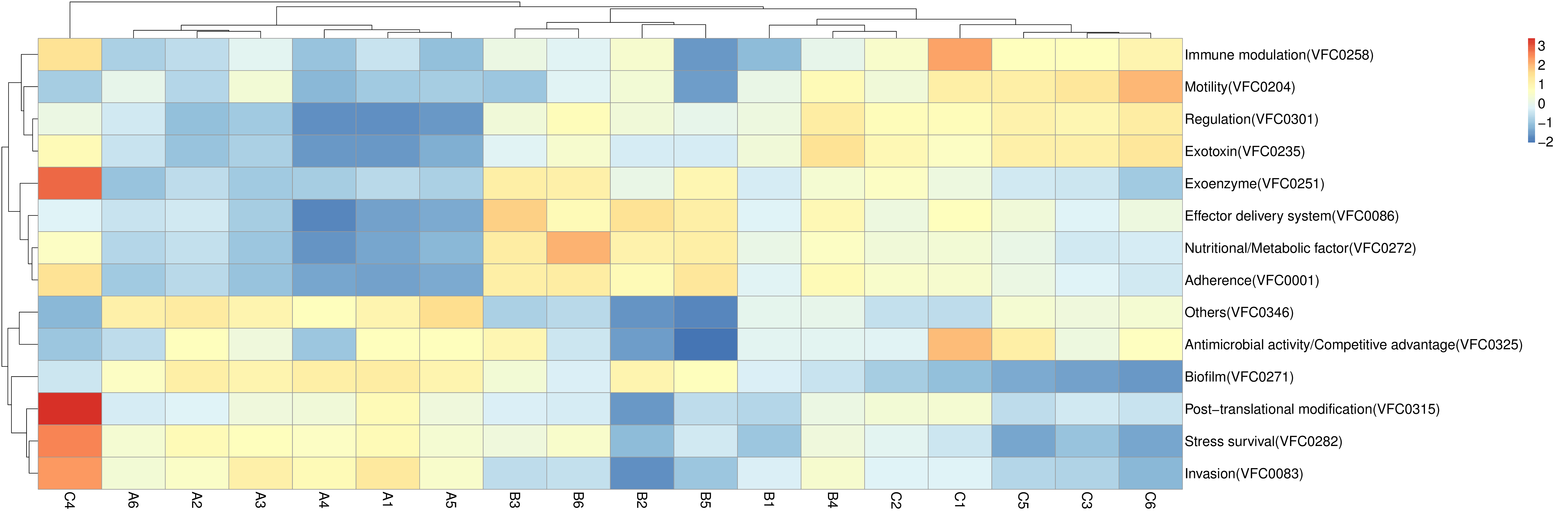
4.4.1.3 注释基因数目及相对丰度聚类分析
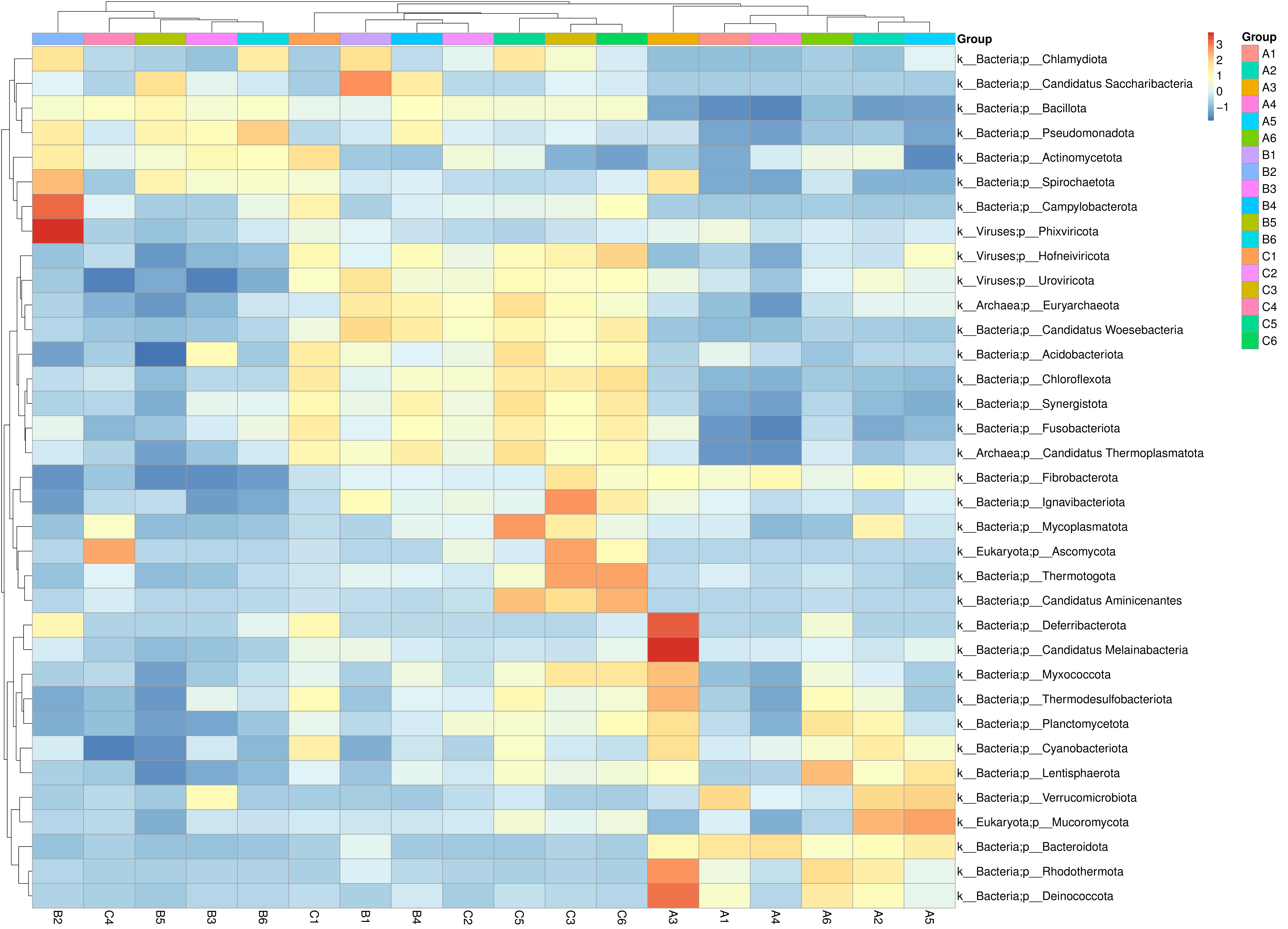
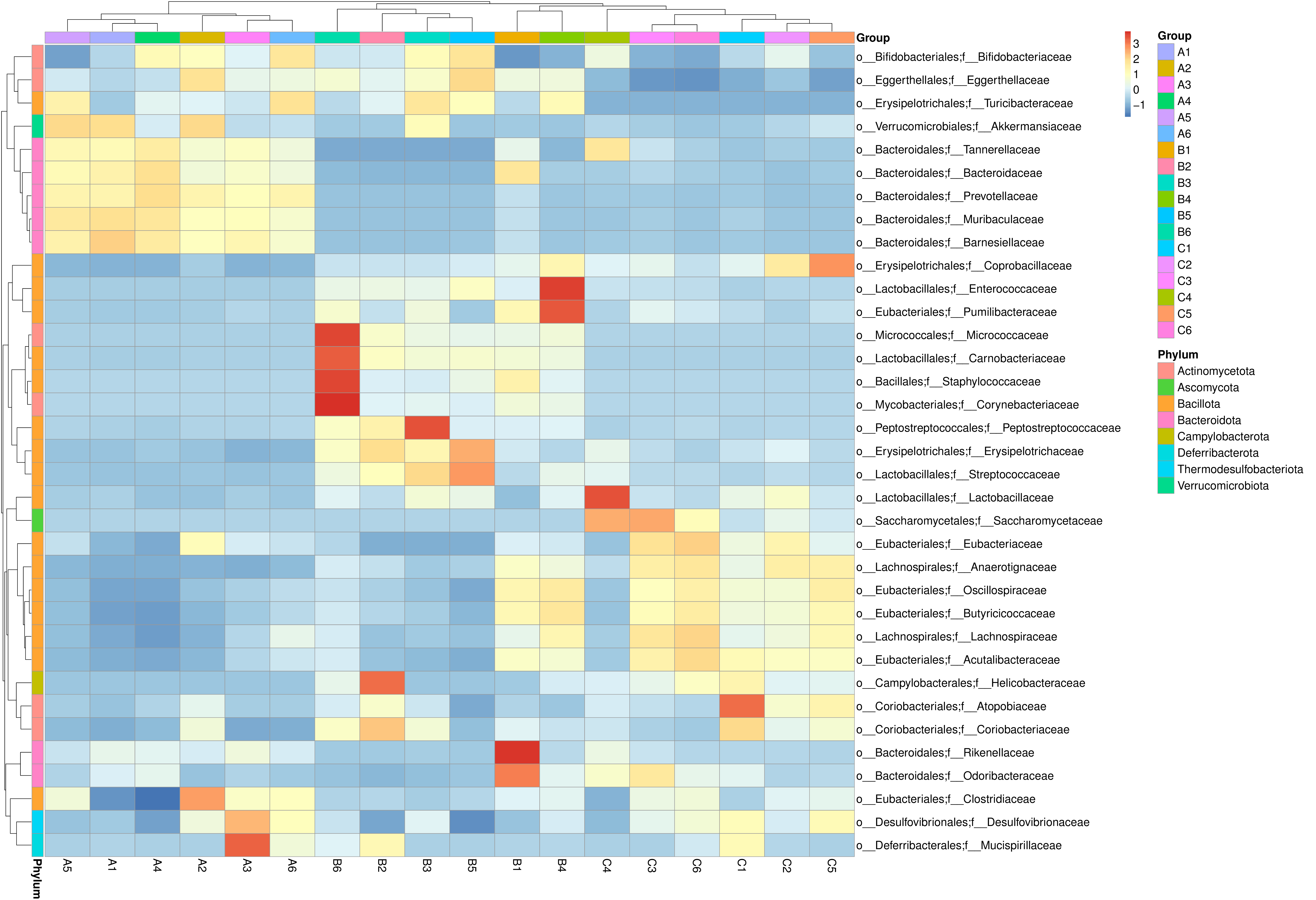
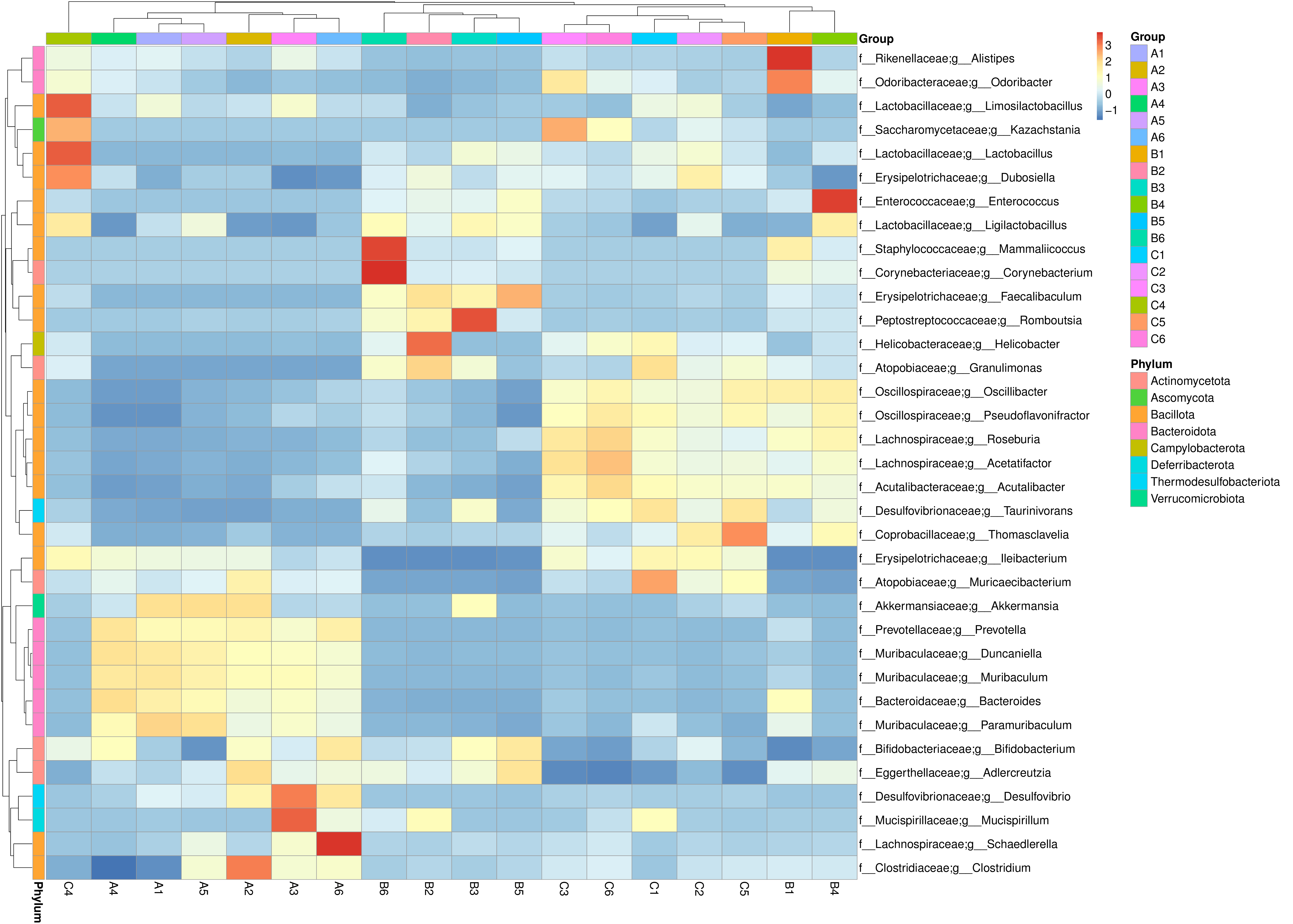
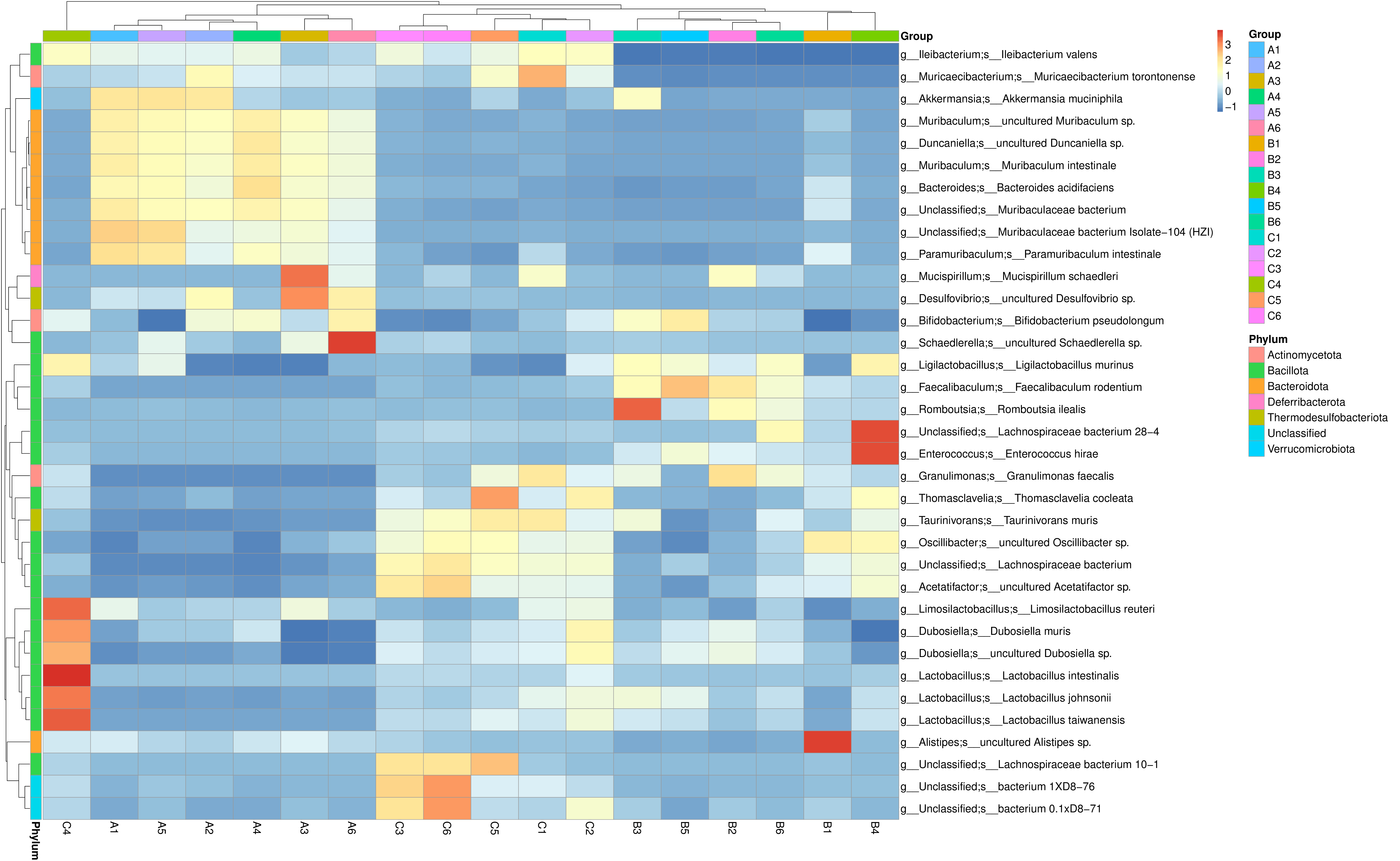
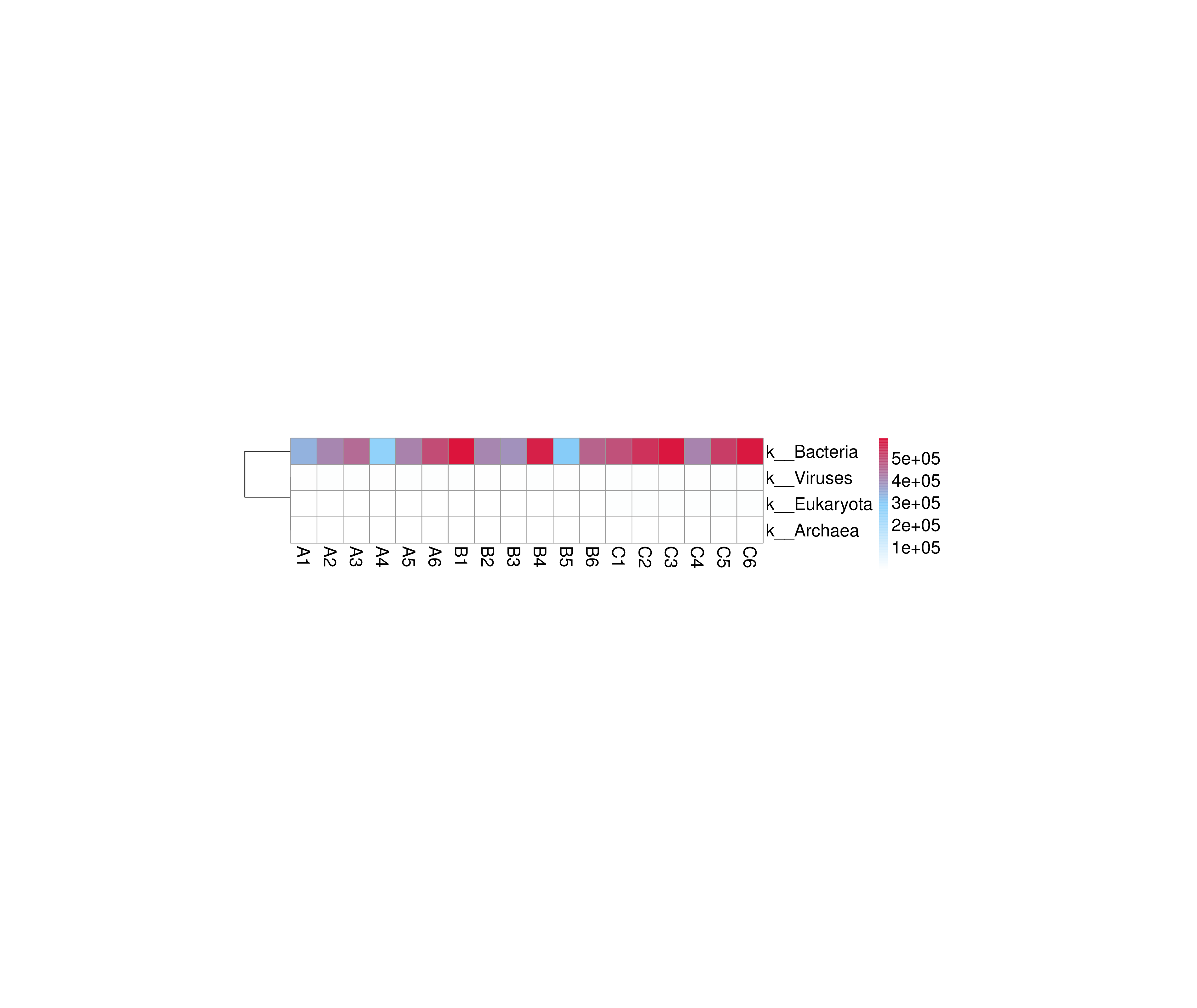
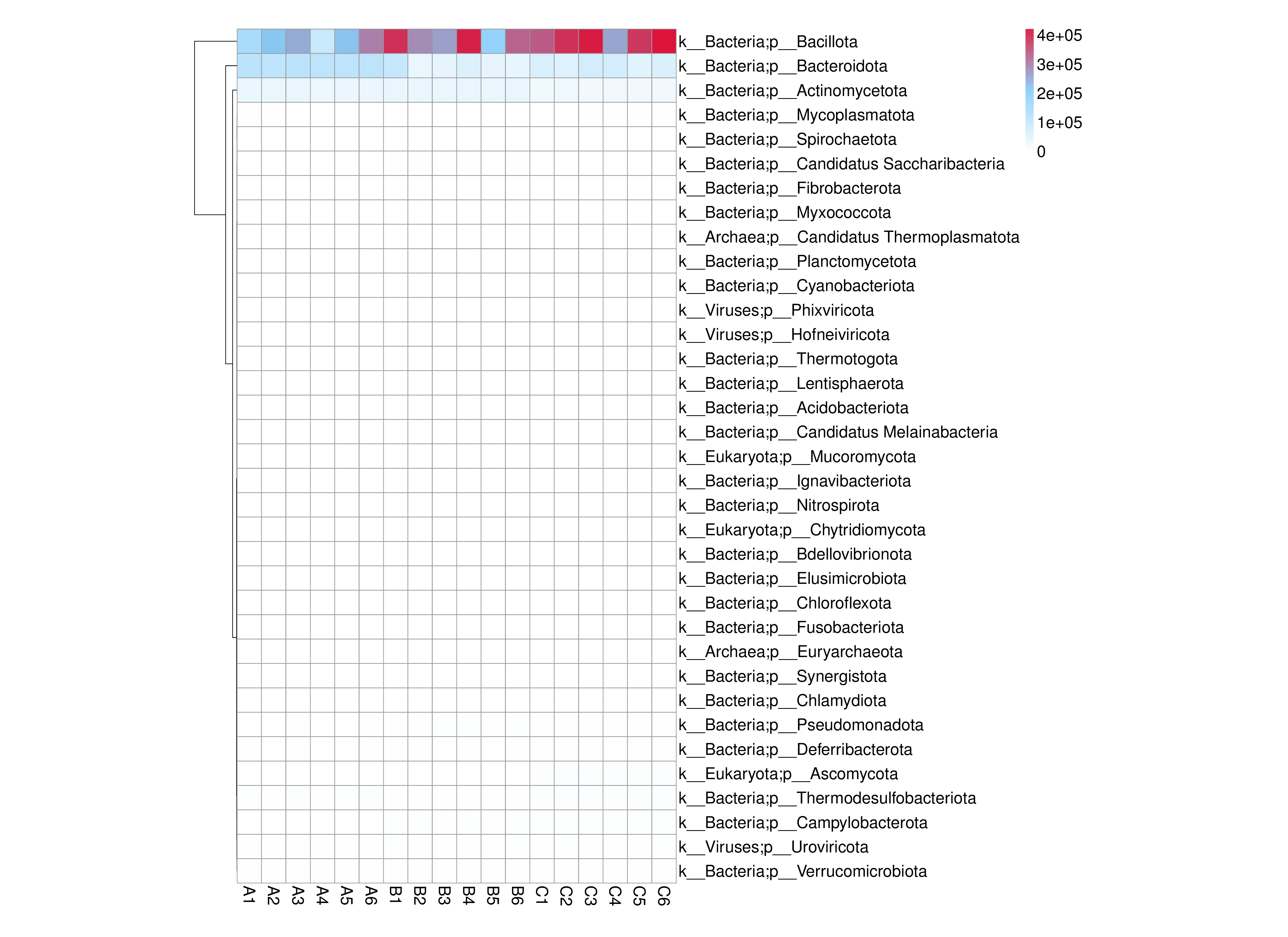
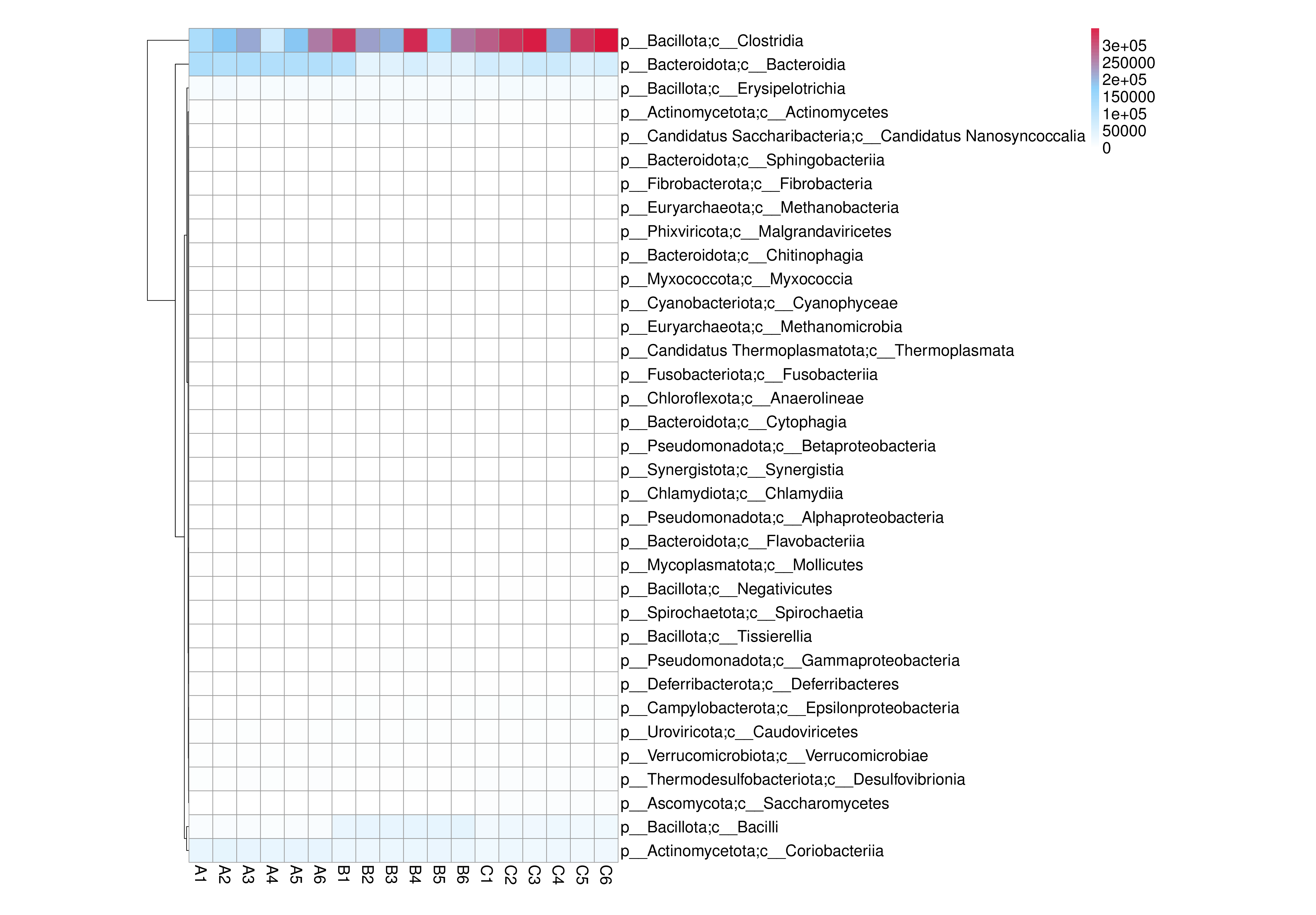
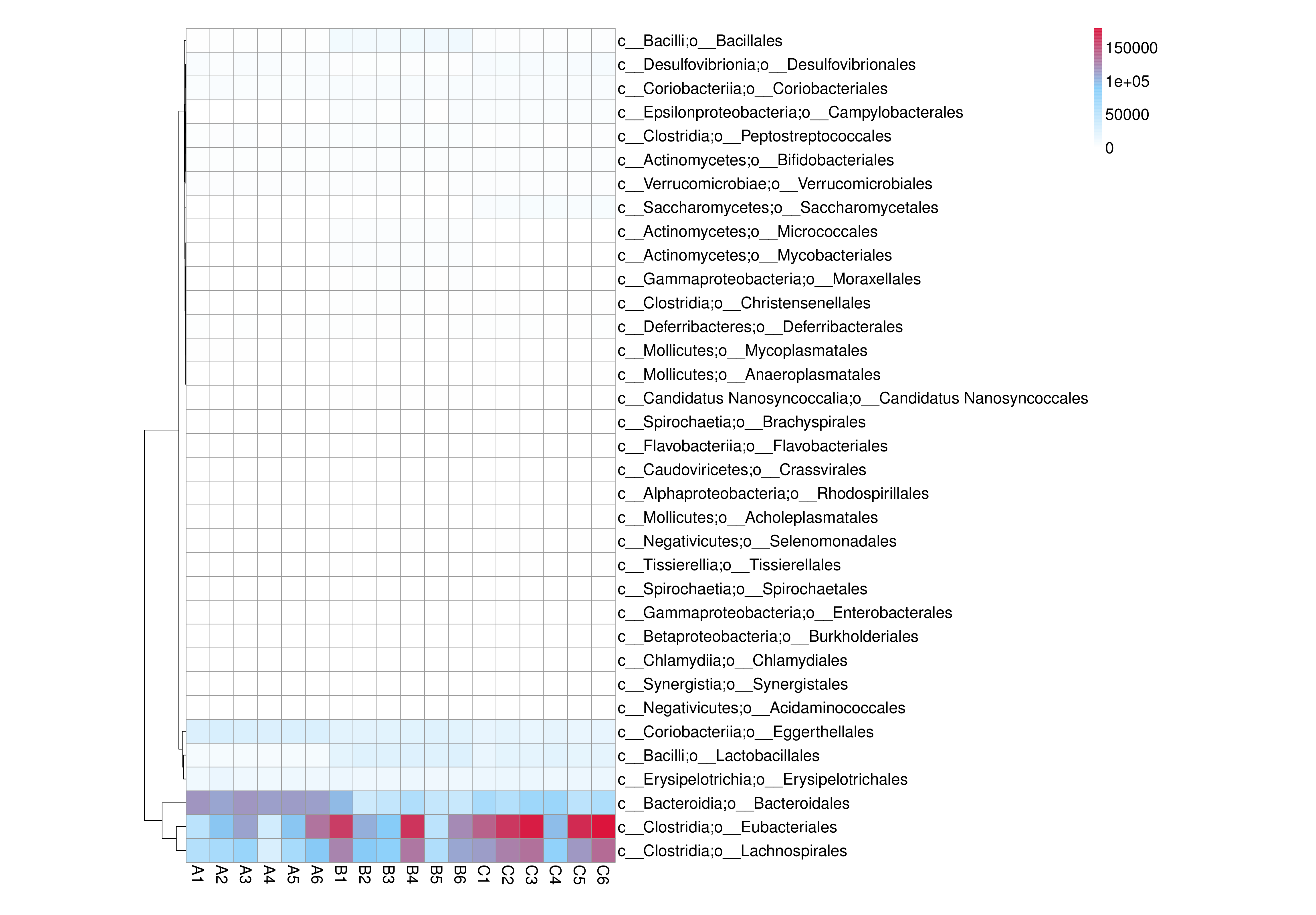
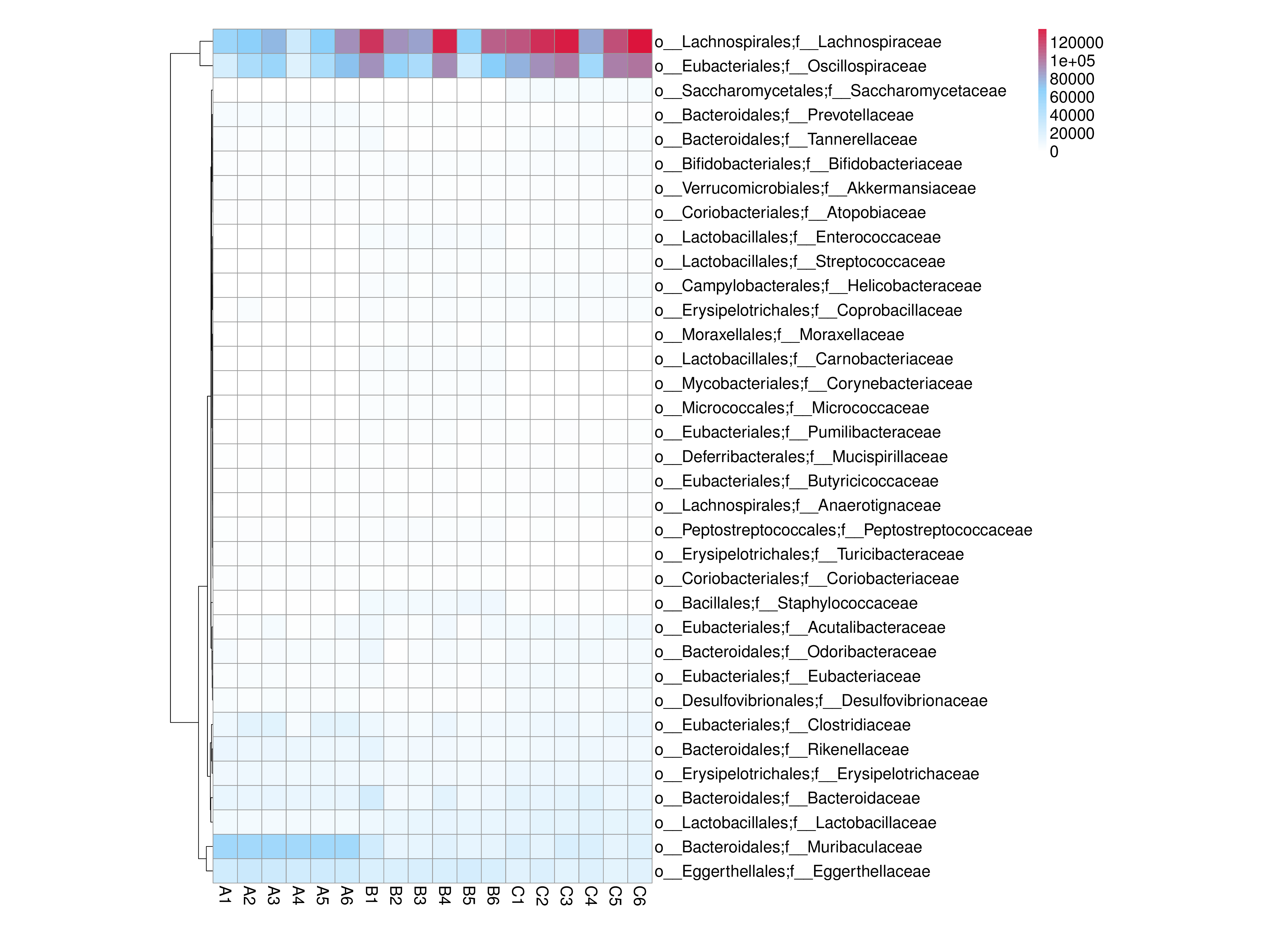
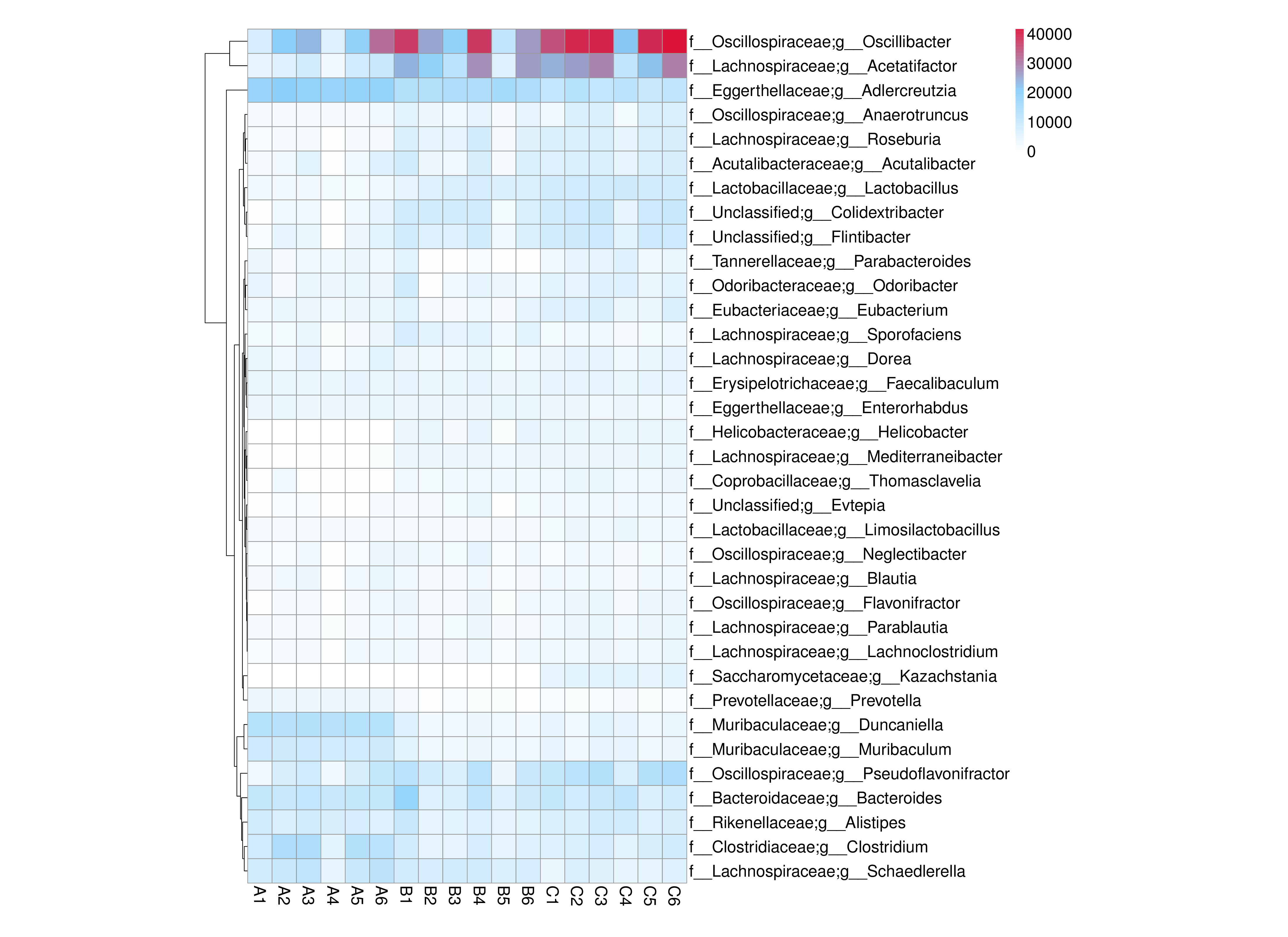
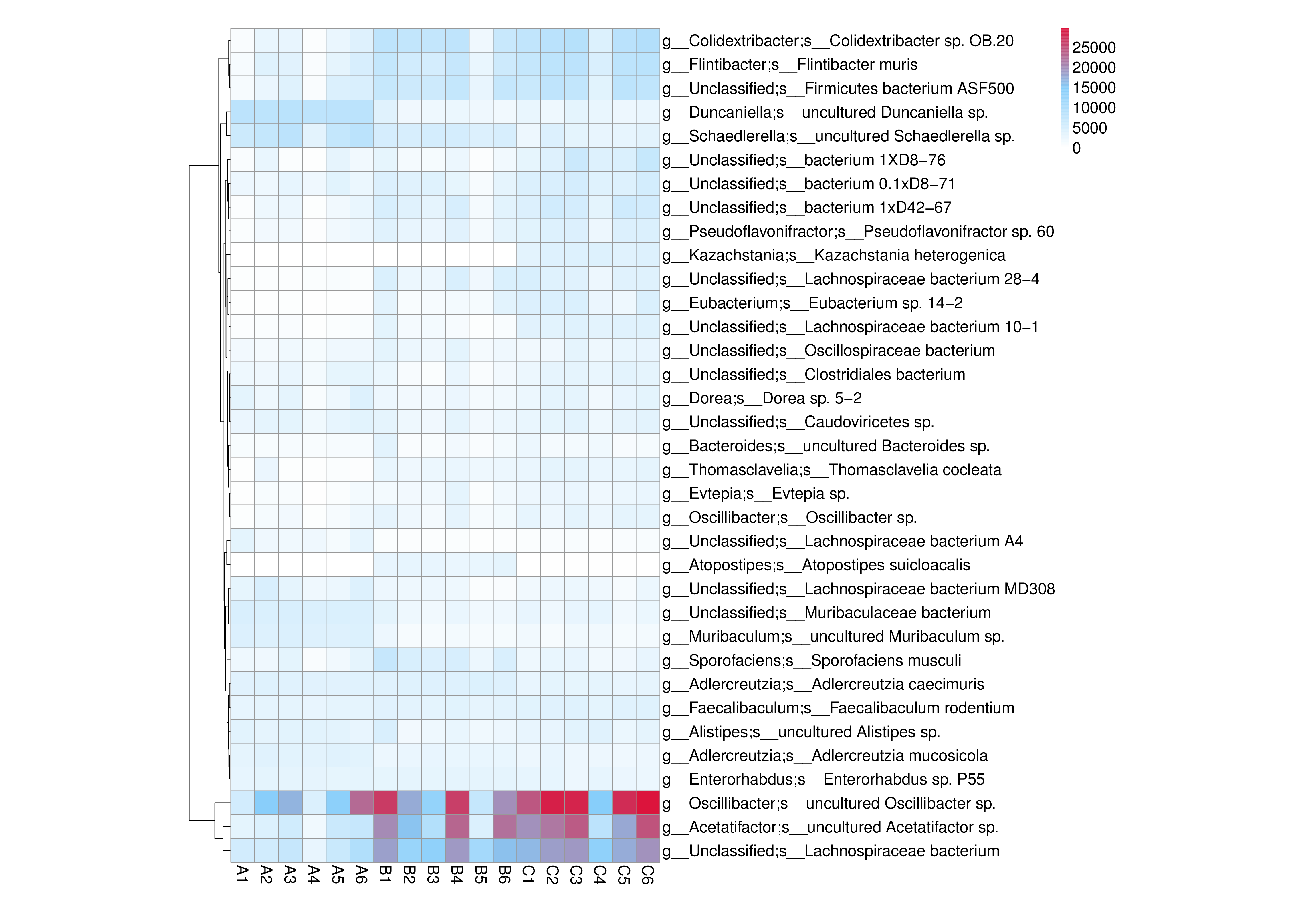
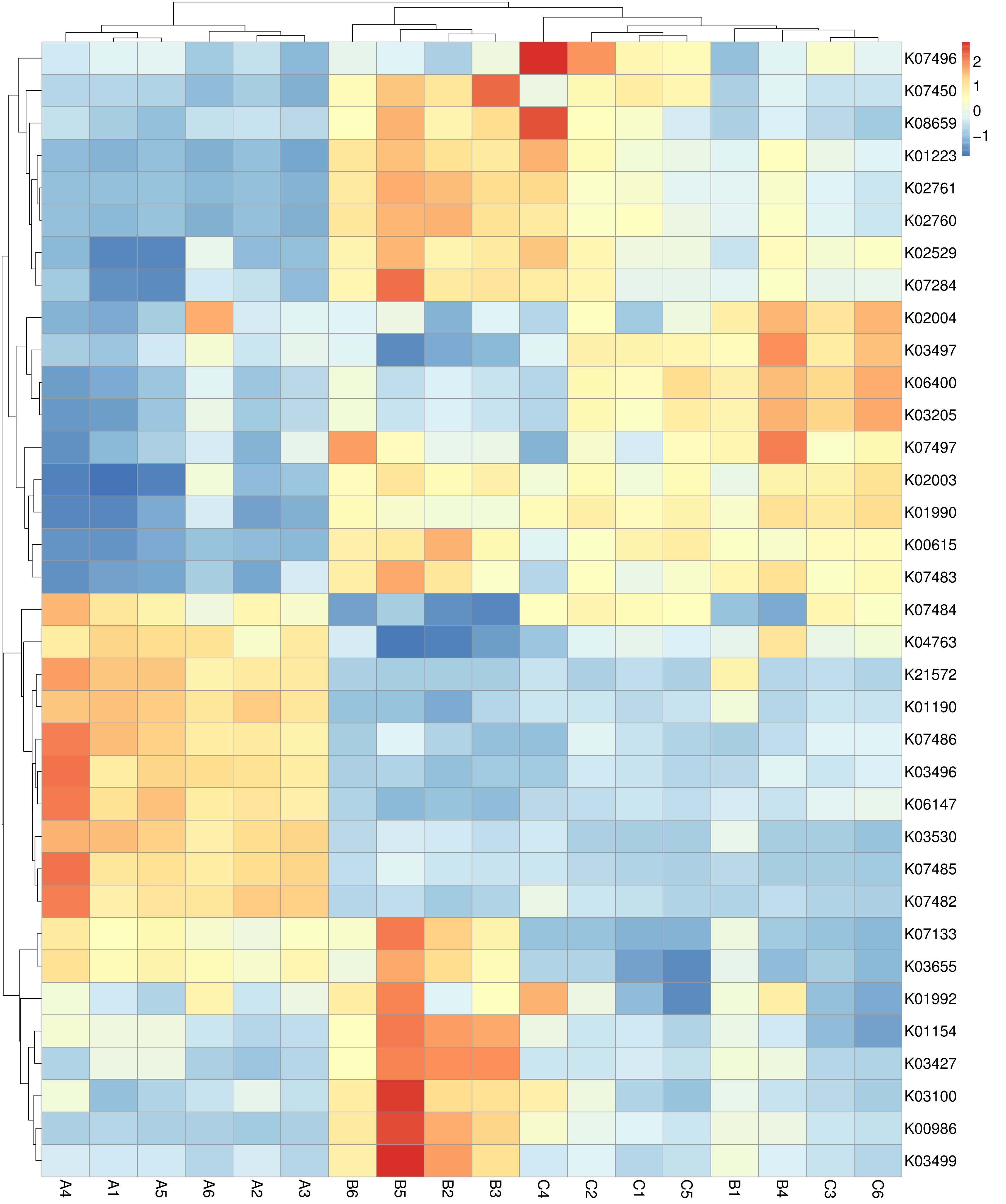
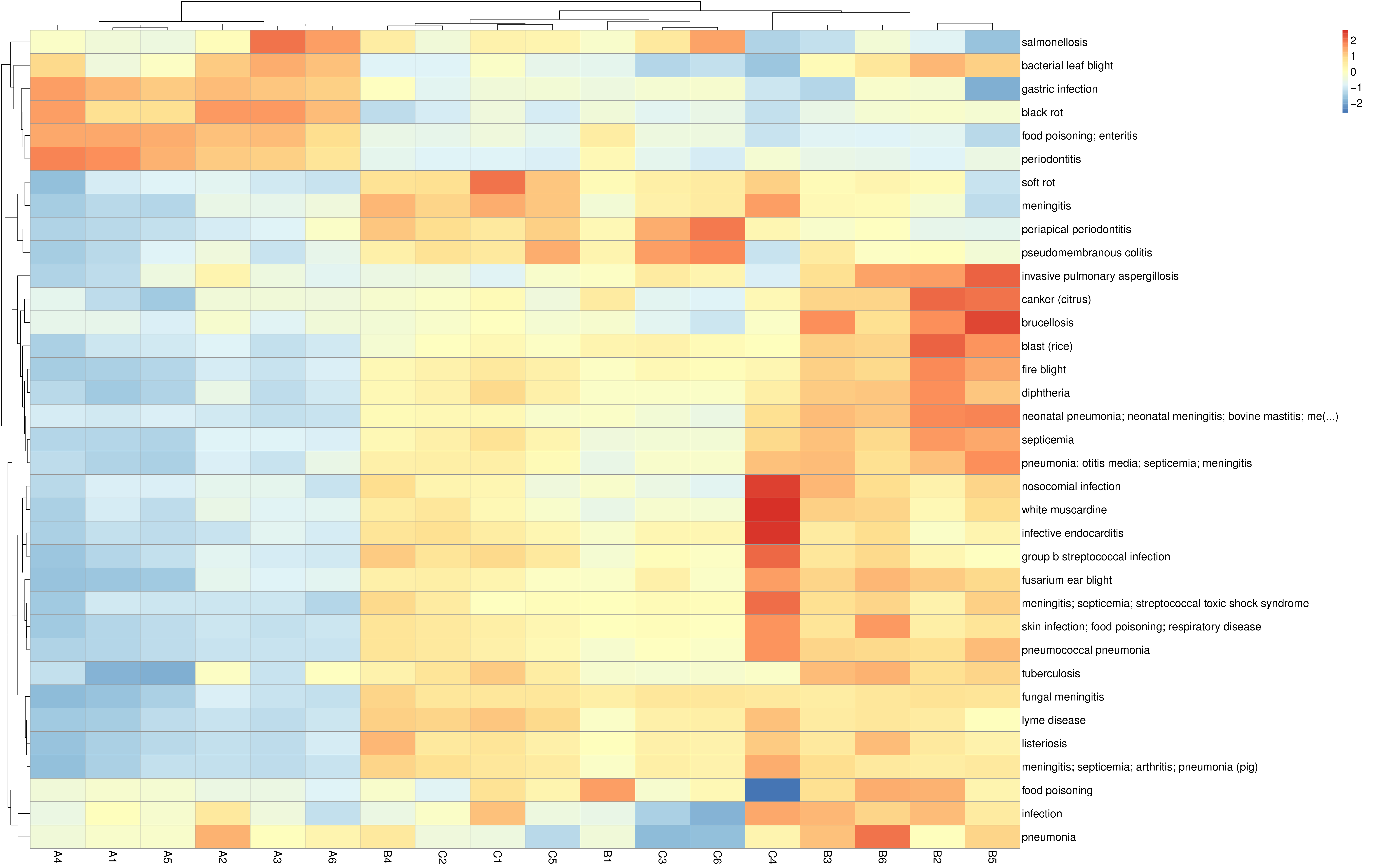
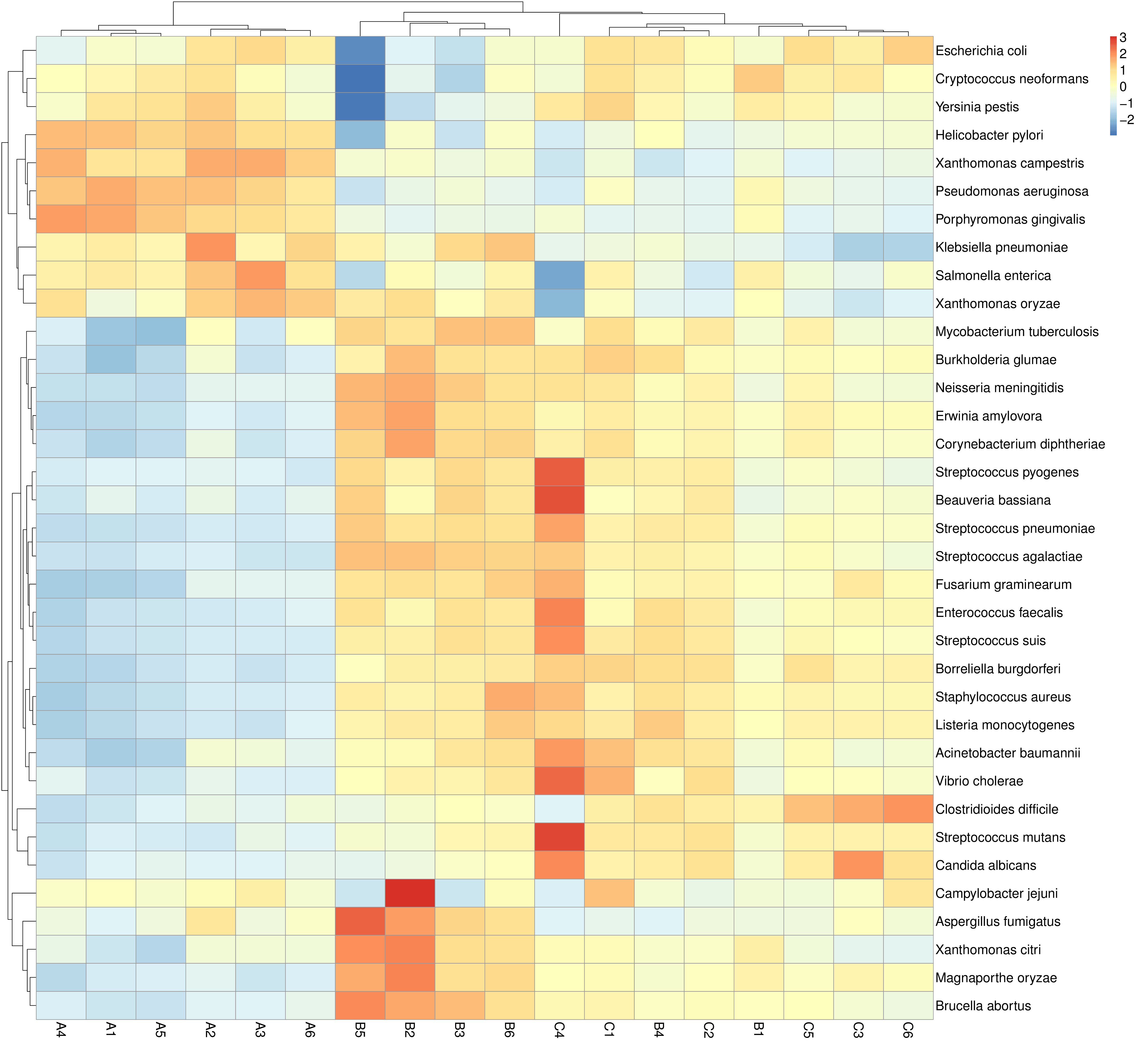
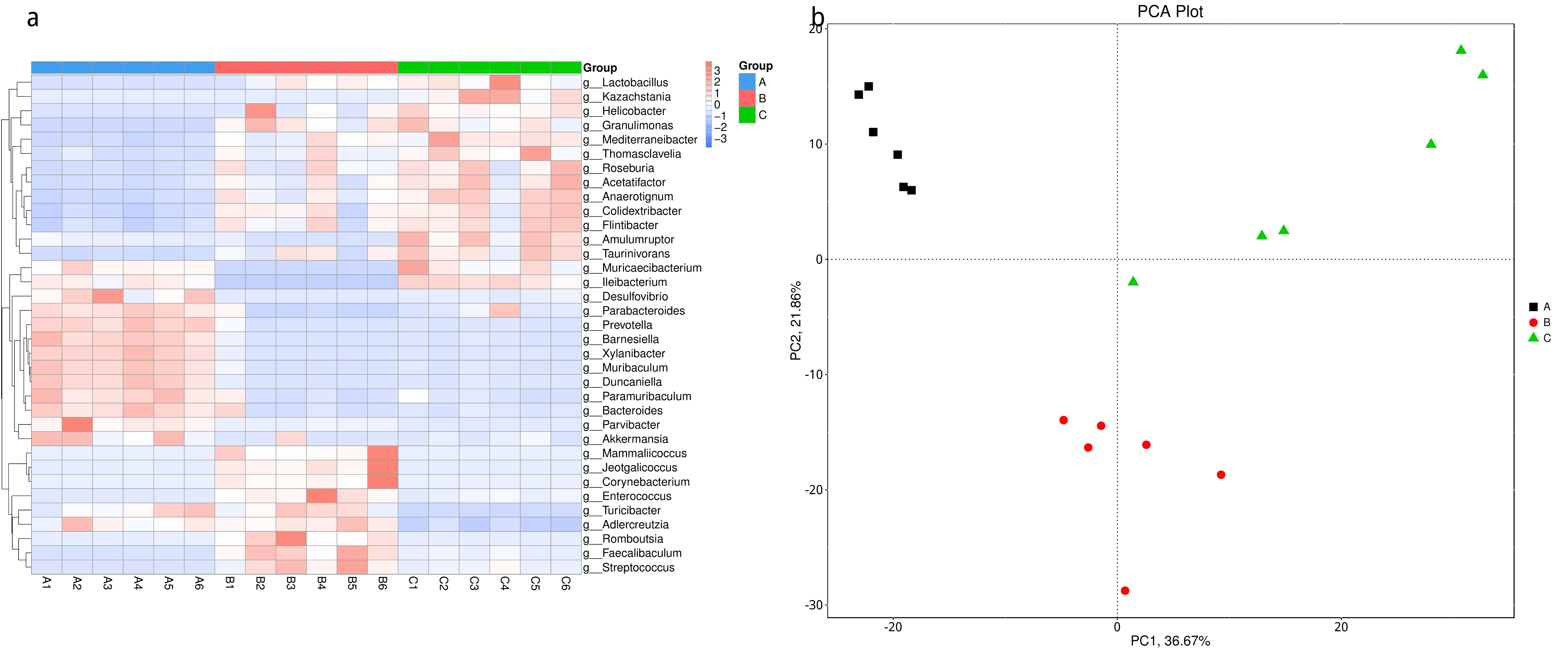
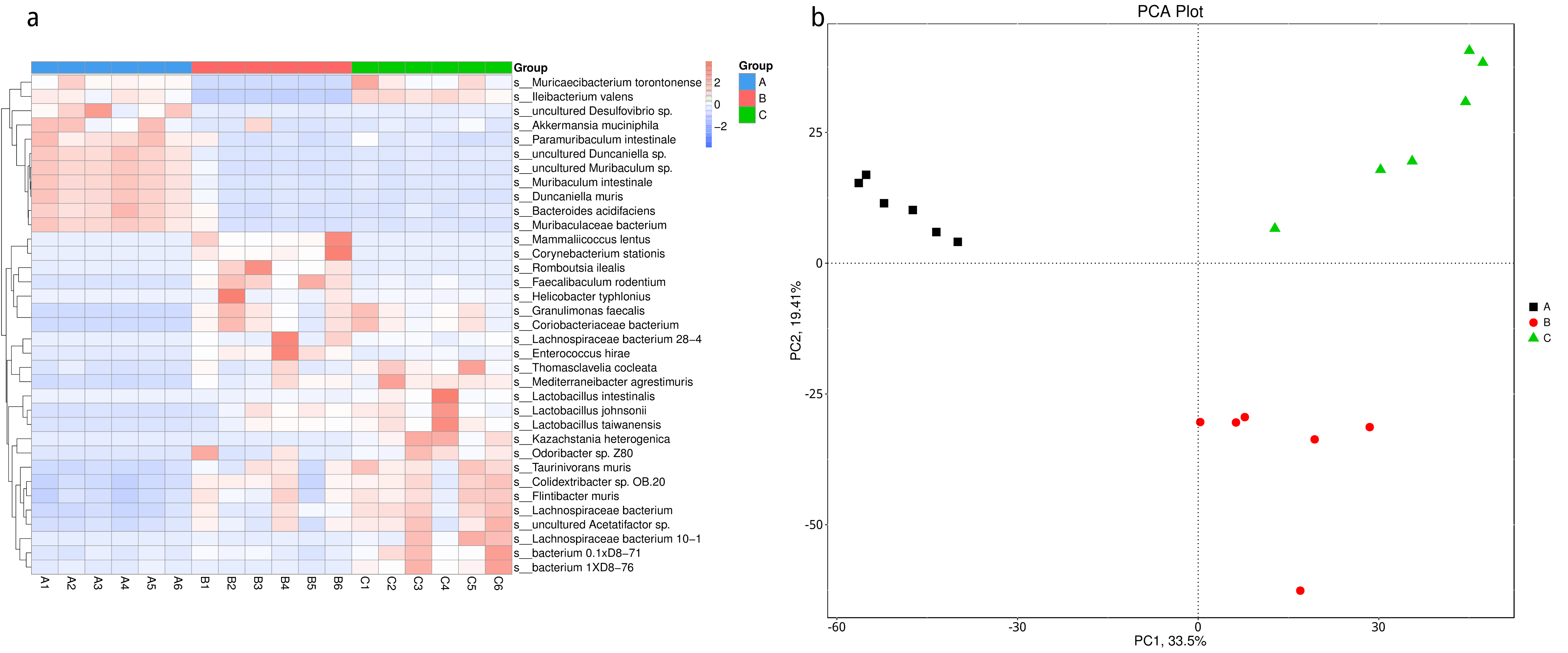
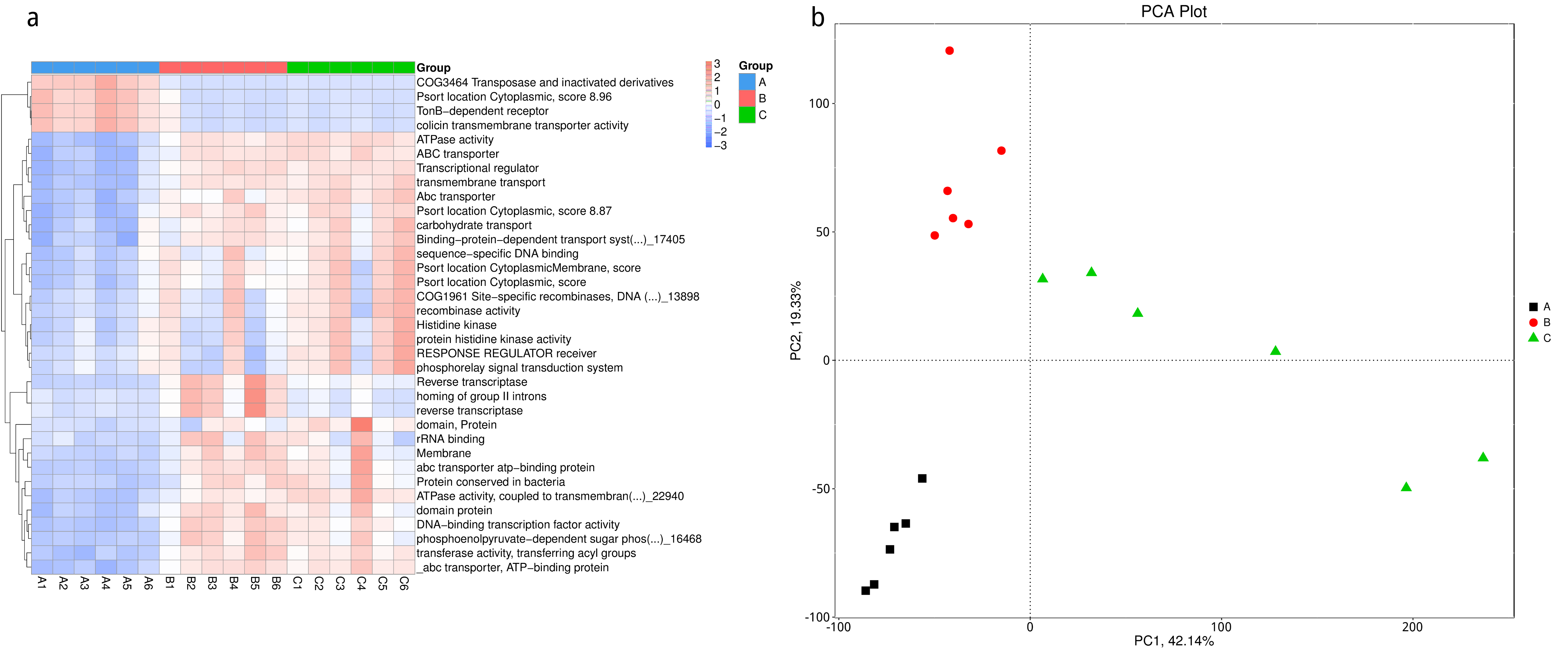
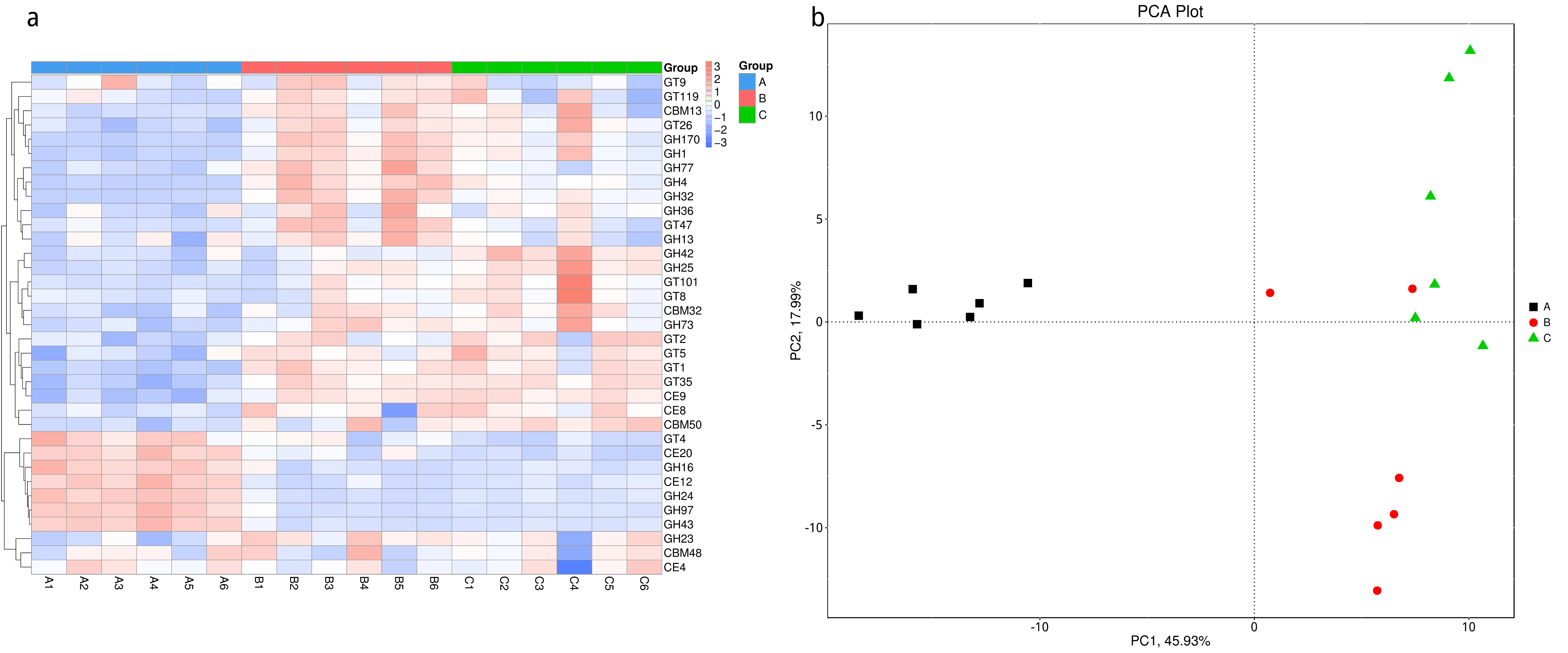
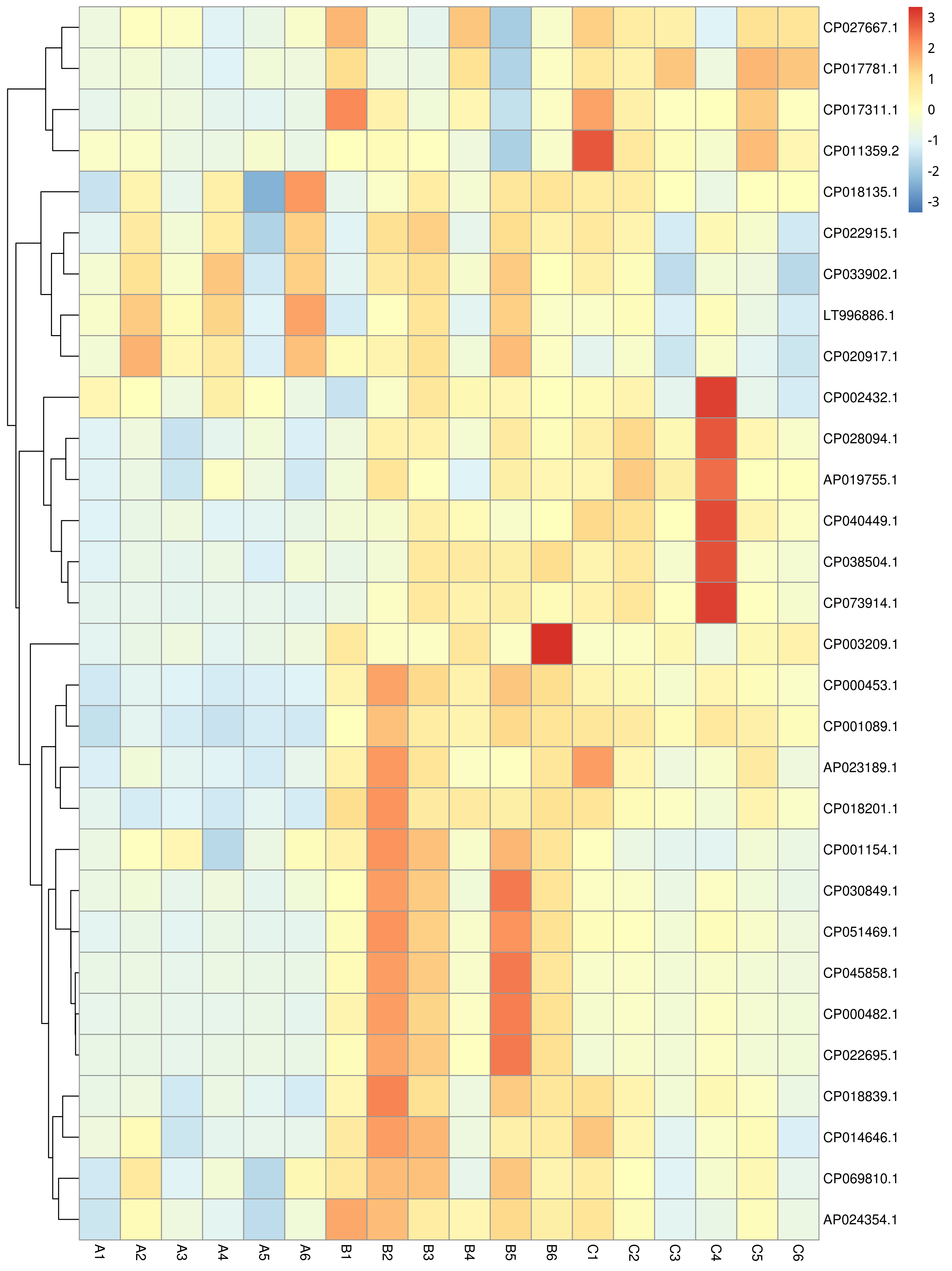
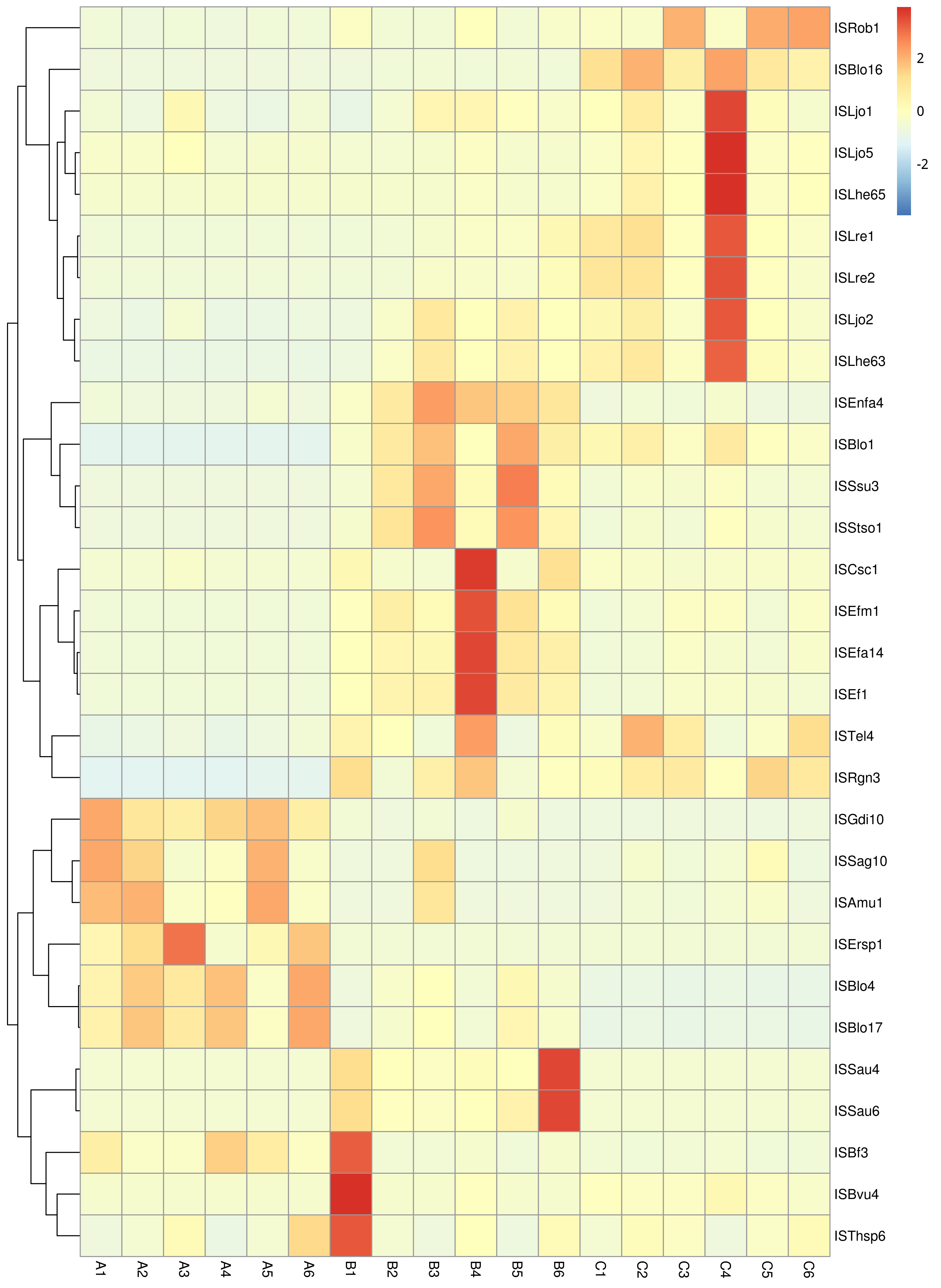
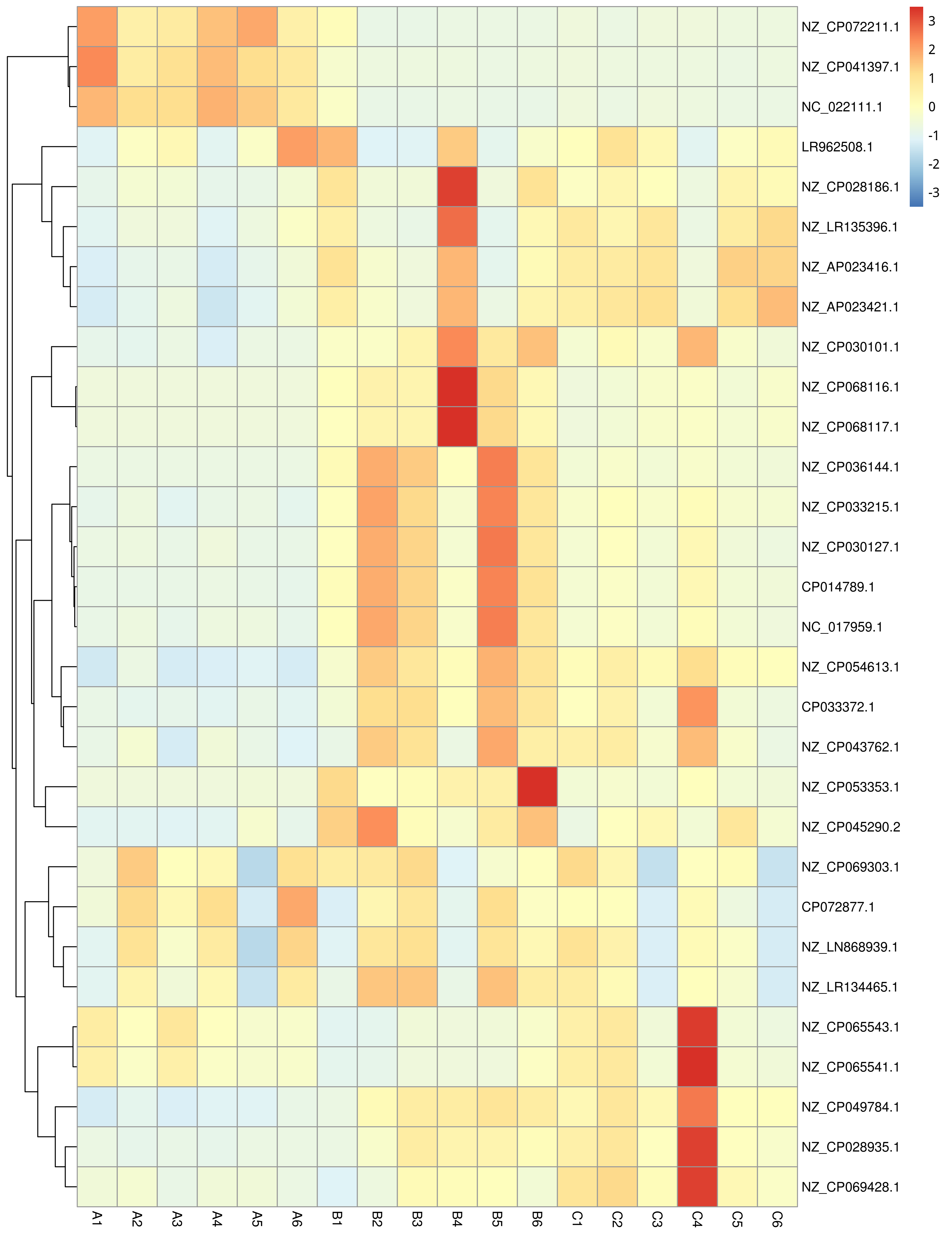
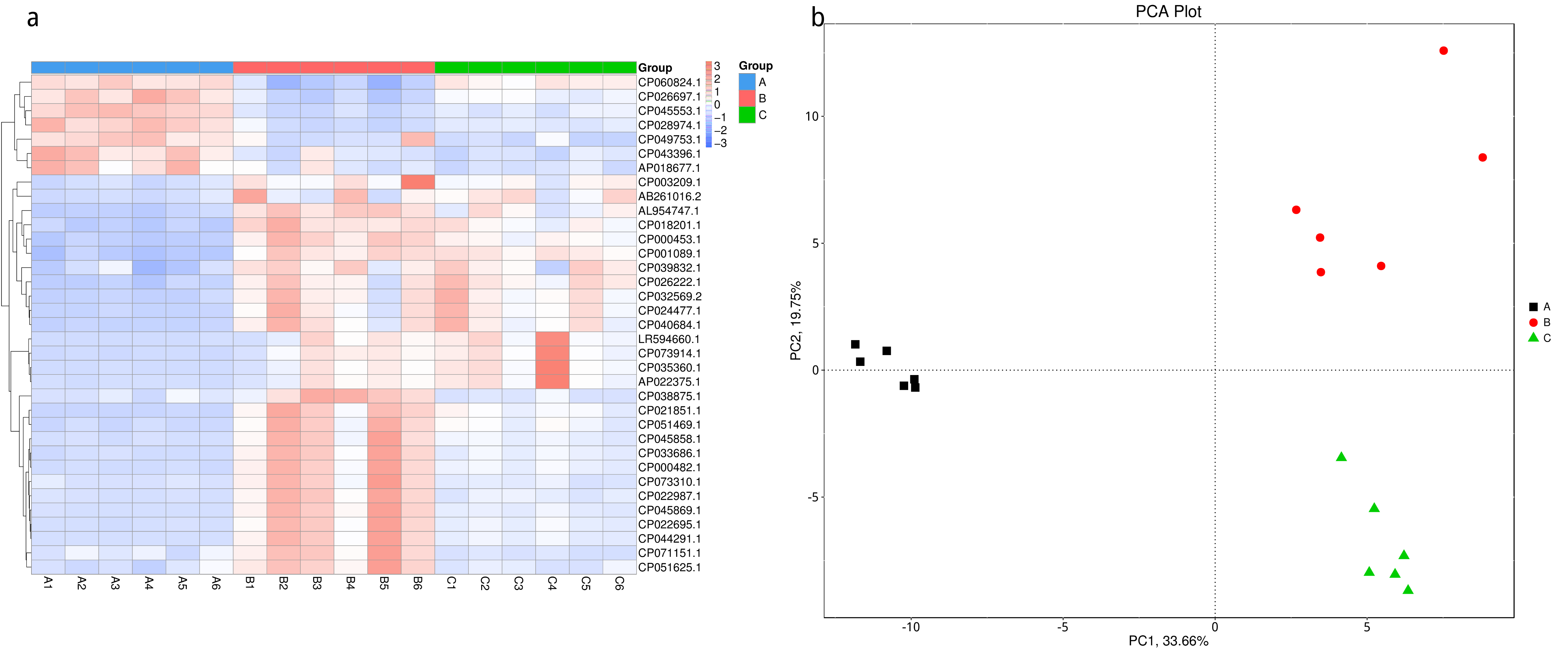
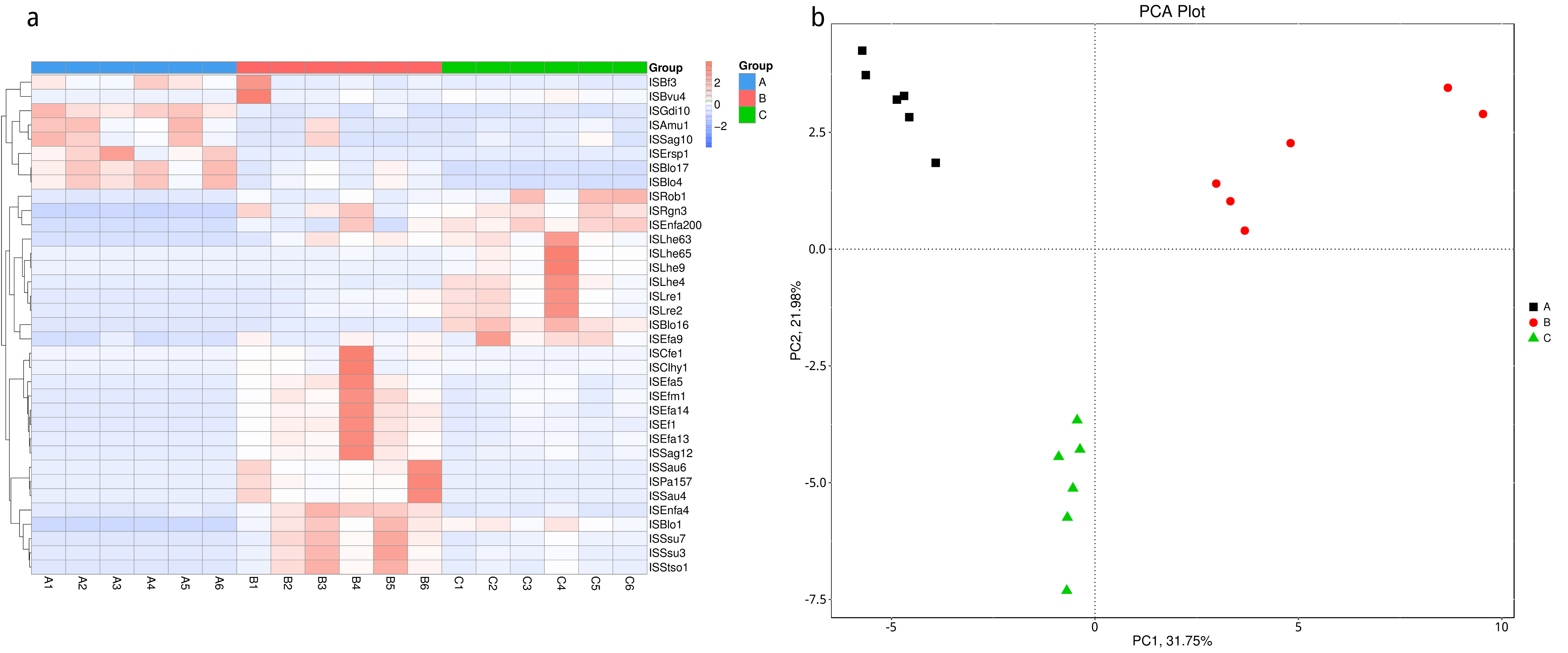
从不同分类层级的相对丰度表和基因数目表出发,选取各分类层级丰度排名前 35 的物种绘制热图,并从物种层面进行聚类,便于结果展示和信息发现,从而找出样品中聚集较多的物种,结果展示见下图:
图4.7 物种丰度聚类热图
说明:物种丰度聚类热图;横向为样品信息;纵向为物种信息;图中左侧的聚类树为物种聚类树;热图对应的值为每一行物种相对丰度经过标准化处理后得到的 Z 值;即一个样品在某个分类上的 Z 值为样品在该分类上的相对丰度和所有样品在该分类的平均相对丰度的差除以所有样品在该分类上的标准差所得到的值
图4.8 基因数目聚类热图
说明:Unigenes 注释数目统计热图;横向为样品名称;纵向为物种信息;不同颜色代表 Unigenes 数目的高低
结果目录:
物种丰度聚类热图见:result/04.Annotation/MicroNR/Heatmap_sample,包括界门纲目科属种(Kingdom、 Phylum、 Class、 Order、 Family、 Genus 、Species)7个分类级别的结果
基因数目聚类热图见:result/04.Annotation/MicroNR/Heatmap_genenums_sample,包括界门纲目科属种(Kingdom、 Phylum、 Class、 Order、 Family、 Genus 、Species)7个分类级别的结果
4.4.2 KEGG 功能蛋白注释
KEGG 数据库(Kanehisa et al., 2006; Kanehisa et al., 2017)于1995年由 Kanehisa Laboratories 推出 0.1 版,目前发展为一个综合性数据库,其中最核心的为 KEGG PATHWAY 和 KEGG ORTHOLOGY 数据库。在KEGG ORTHOLOGY数据库中,将行使相同功能的基因聚在一起,称为Ortholog Groups (KO entries),每个KO包含多个基因信息,并在一至多个pathway 中发挥作用。而在 KEGG PATHWAY 数据库中,将生物代谢通路划分为 6 类,分别为:细胞过程(Cellular Processes)、环境信息处理(Environmental Information Processing)、遗传信息处理(Genetic Information Processing)、人类疾病(Human Diseases)、新陈代谢(Metabolism)、生物体系统(Organismal Systems),其中每类又被系统分类为二、三、四层。第二层目前包括有 57个种子 pathway;第三层即为其代谢通路图;第四层为每个代谢通路图的具体注释信息。
4.4.2.1 KEGG 注释基本步骤
使用DIAMOND软件将unigene与KEGG功能数据库进行比对(Li et al., 2014; Feng et al., 2015),然后进行比对结果过滤,对于每一条序列的比对结果,选取score最高的比对结果(one HSP > 60 bits)进行后续分析(Qin et al., 2012; Li et al., 2014; Qin et al., 2014; Bäckhed et al, 2015)。
从功能注释结果及基因丰度表出发,获得各个样品在各个分类层级上的基因数目表,对于某个功能在某个样品中的基因数目,等于在注释为该功能的基因中,丰度不为 0 的基因数目。
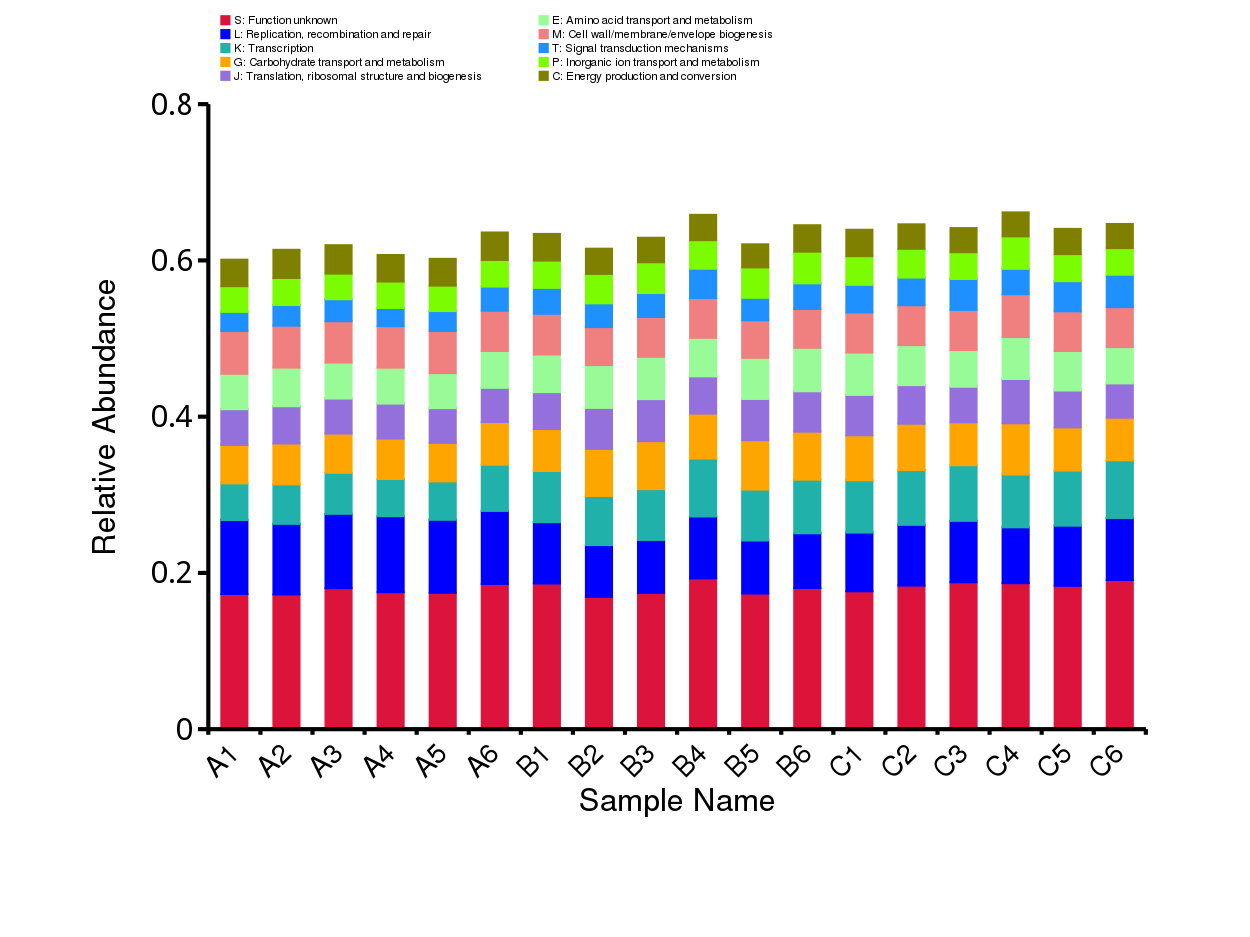
4.4.2.2 KEGG 注释基因数目统计
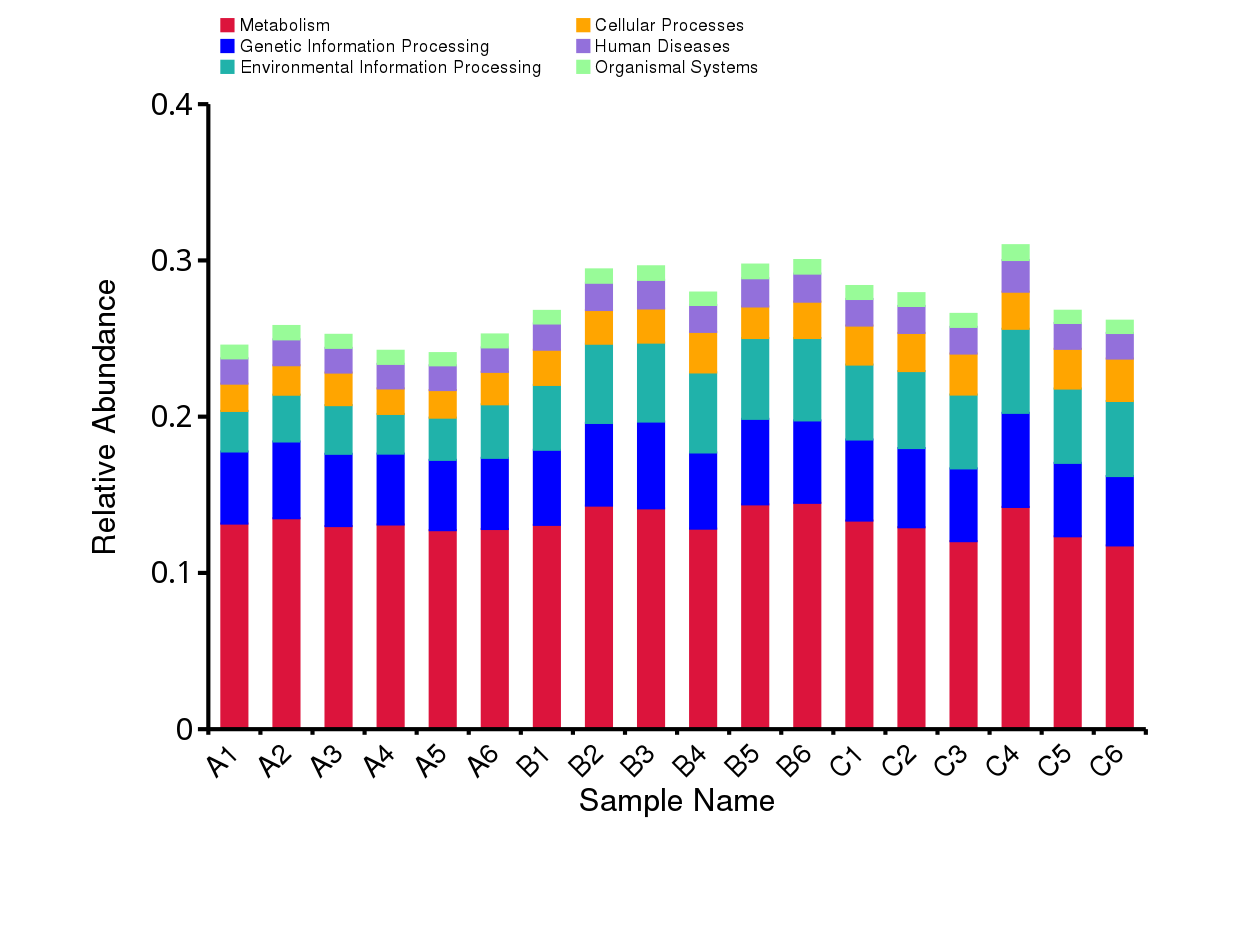
从 unigene 注释结果出发,绘制KEGG 数据库level1 层级的注释基因数目统计图,展示结果如下图所示:
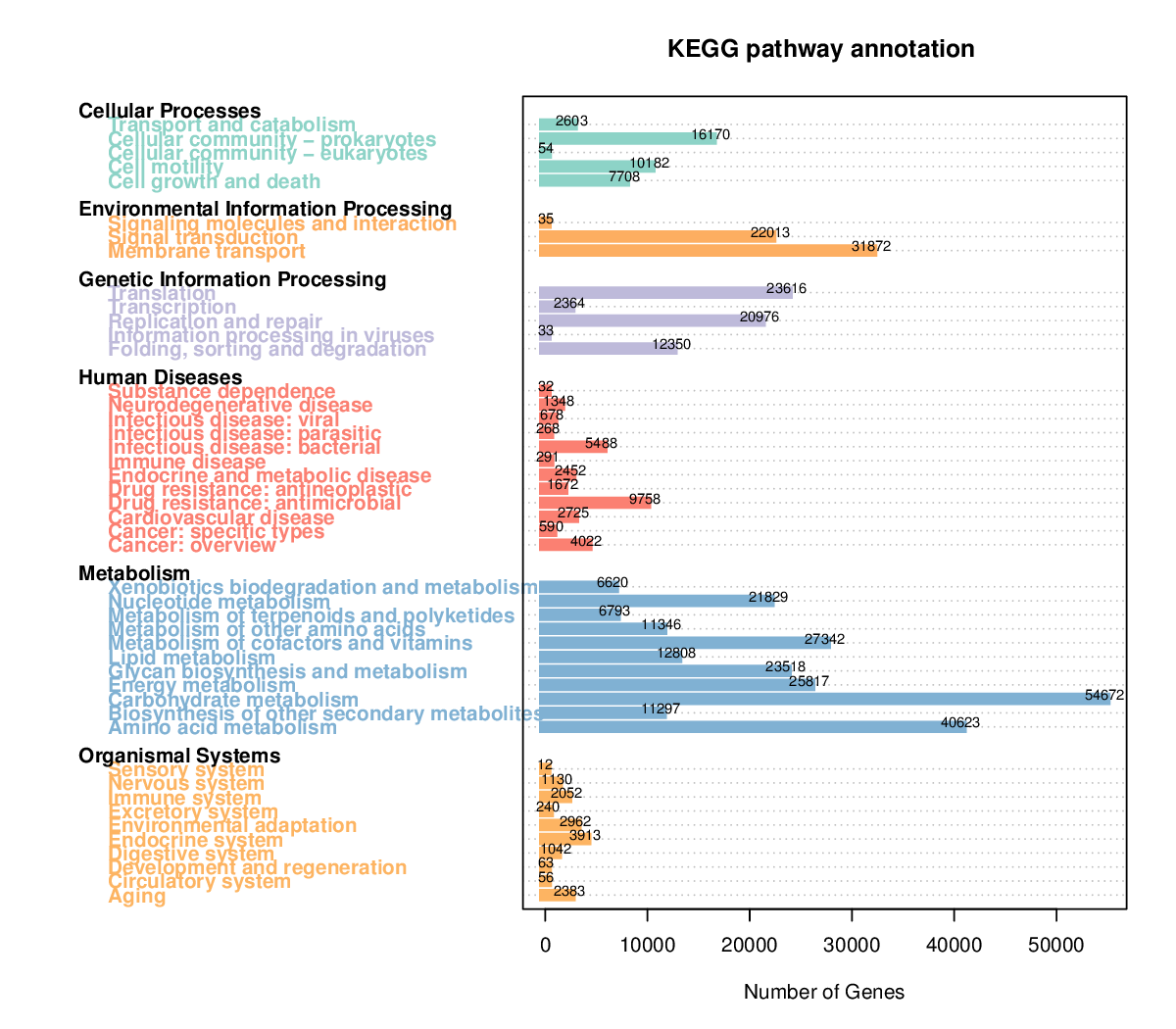
图4.9 KEGG数据库注释基因数目统计图
结果目录:
KEGG数据库注释基因数目统计图见:result/04.Annotation/KEGG/Anno/KEGG.unigenes.num.{pdf,png}
4.4.2.3 KEGG 相对丰度概况
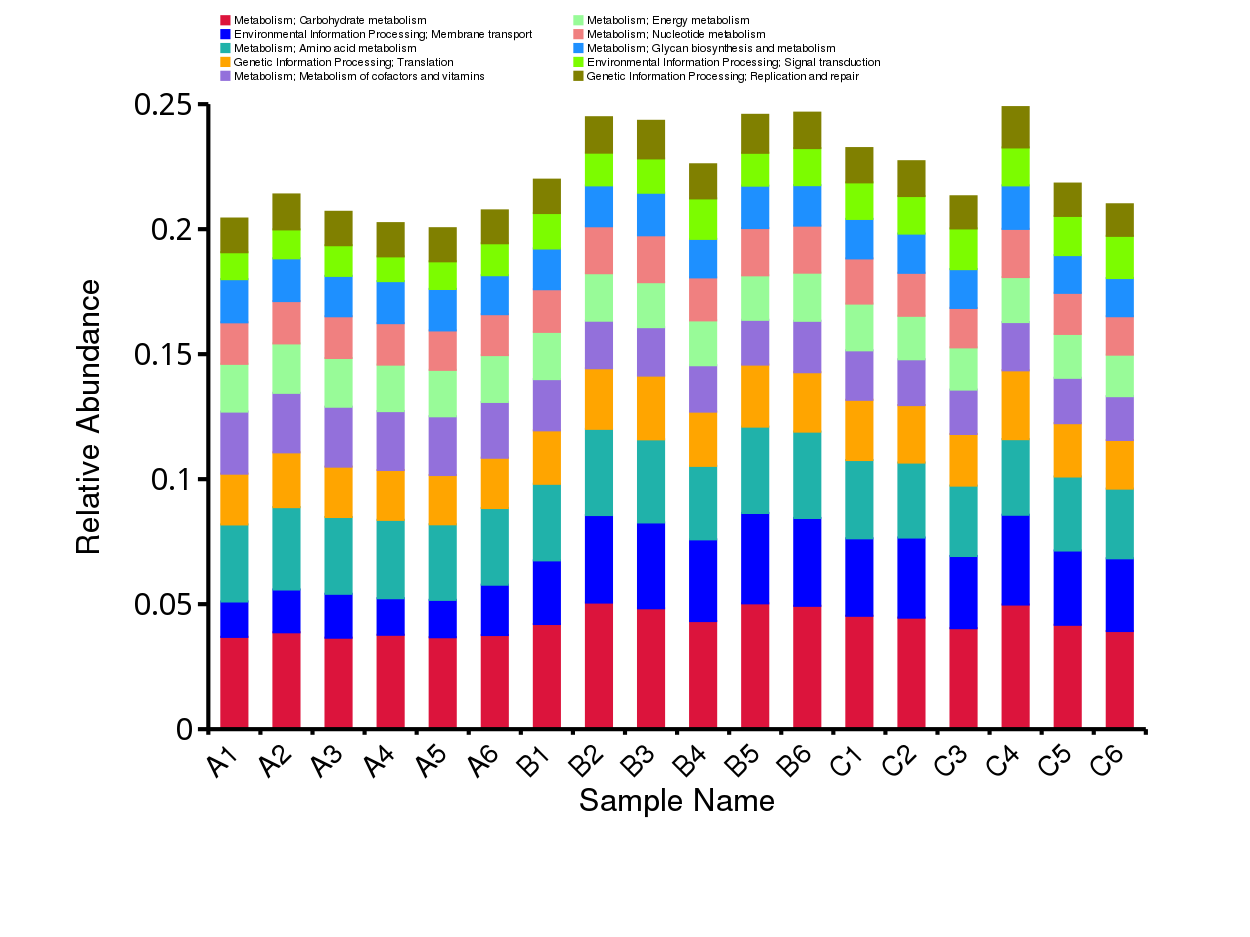
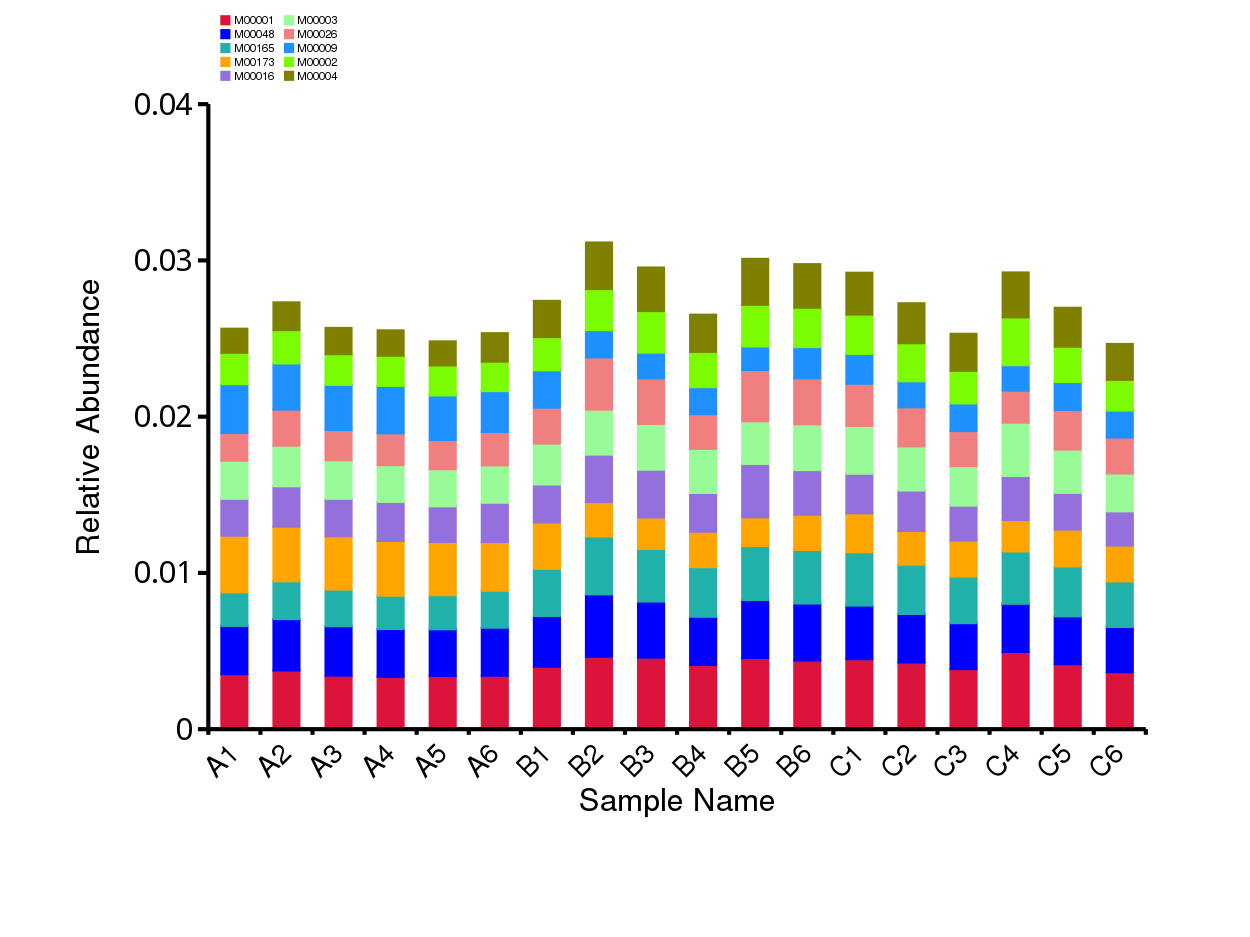
根据KEGG数据库的注释结果,绘制了样品(组)在KEGG数据库中对应层级上的相对丰度统计图。以各样品在KEGG数据库中level1层级上的相对丰度统计图为例展示。其余层级为相对丰度前10的注释结果。
图4.10 KEGG 功能注释相对丰度柱形图
说明:每个层级的相对丰度柱形图,横向表示样品名称,纵向表示注释到某类型功能的相对比例;各颜色区块对应的功能类别见图例
结果目录:
样品 - KEGG各层级相对丰度前10的相对丰度柱形图见:result/04.Annotation/KEGG/Top_sample/*/*.top10.{svg,png}
组 - KEGG各层级相对丰度前10的相对丰度柱形图见:result/04.Annotation/KEGG/Top_group1/*/*.top10.{svg,png}
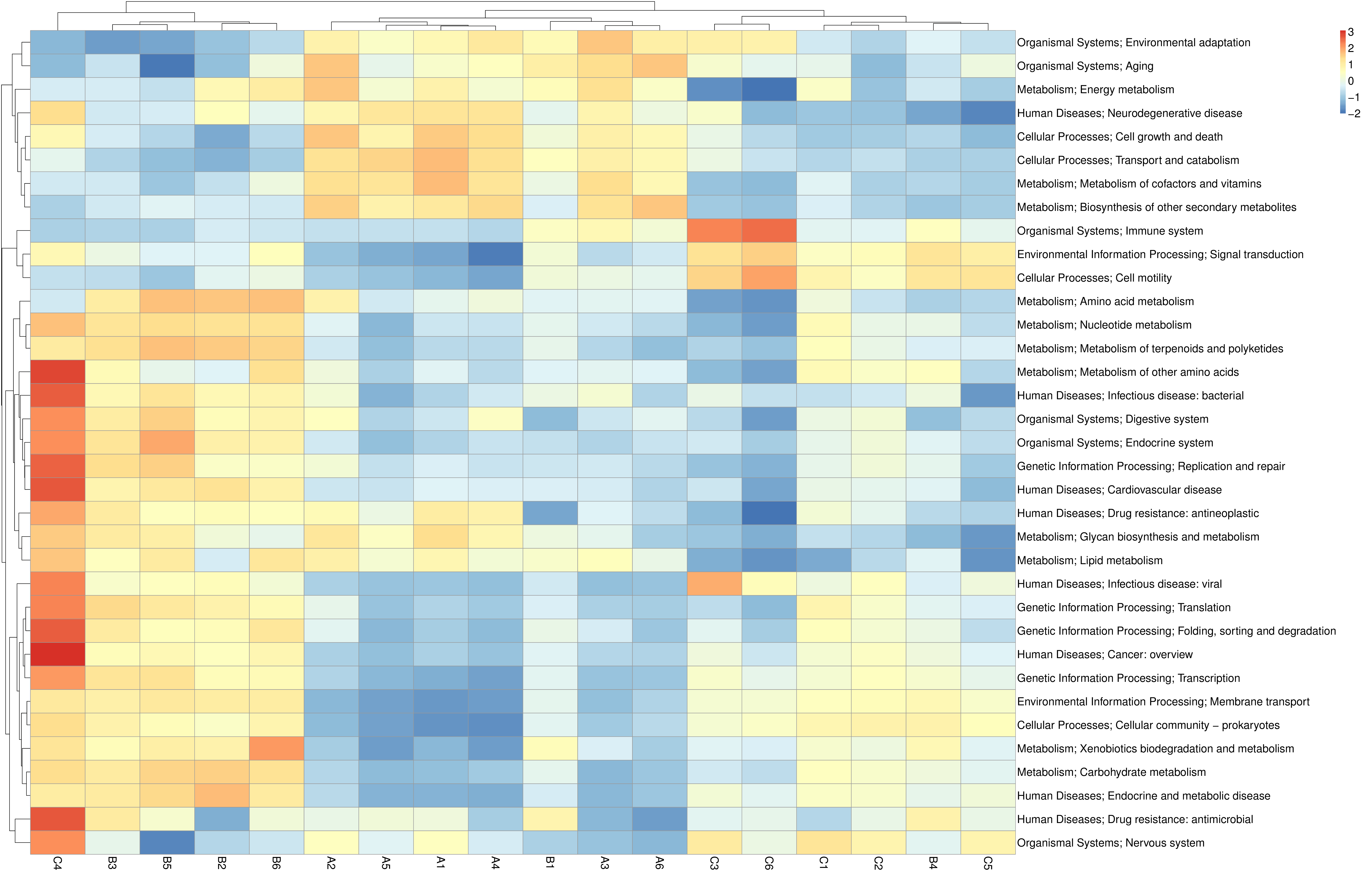
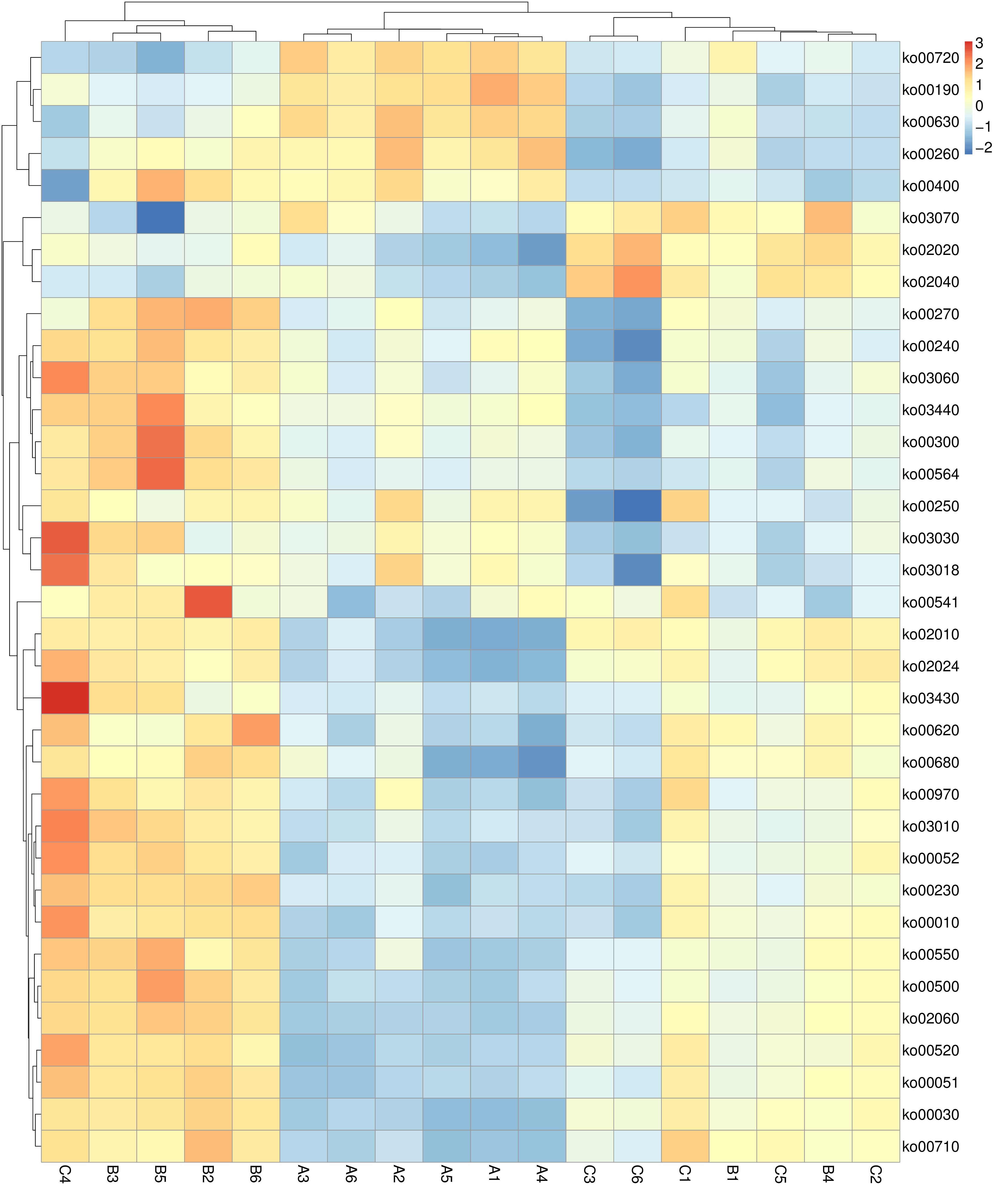
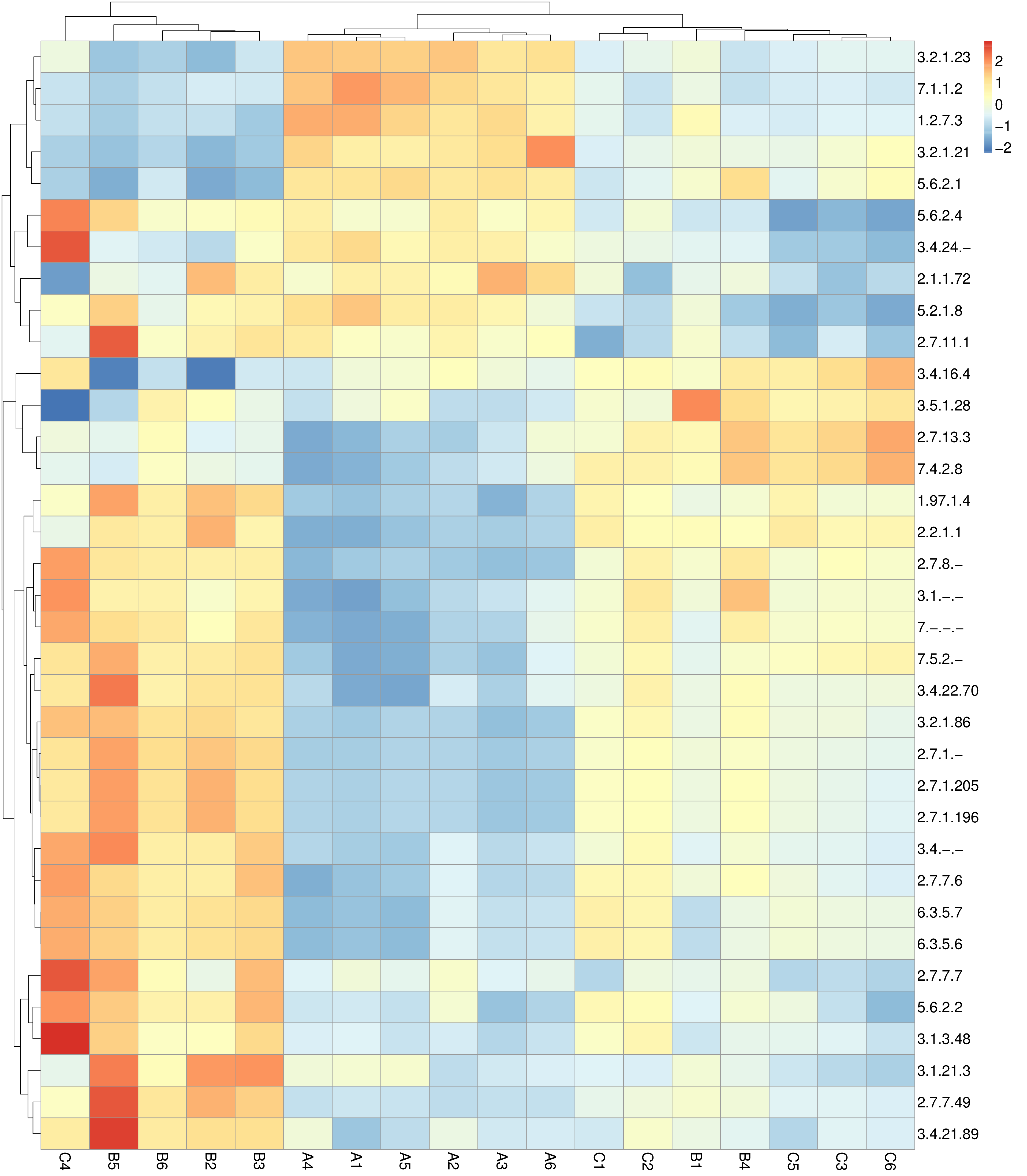
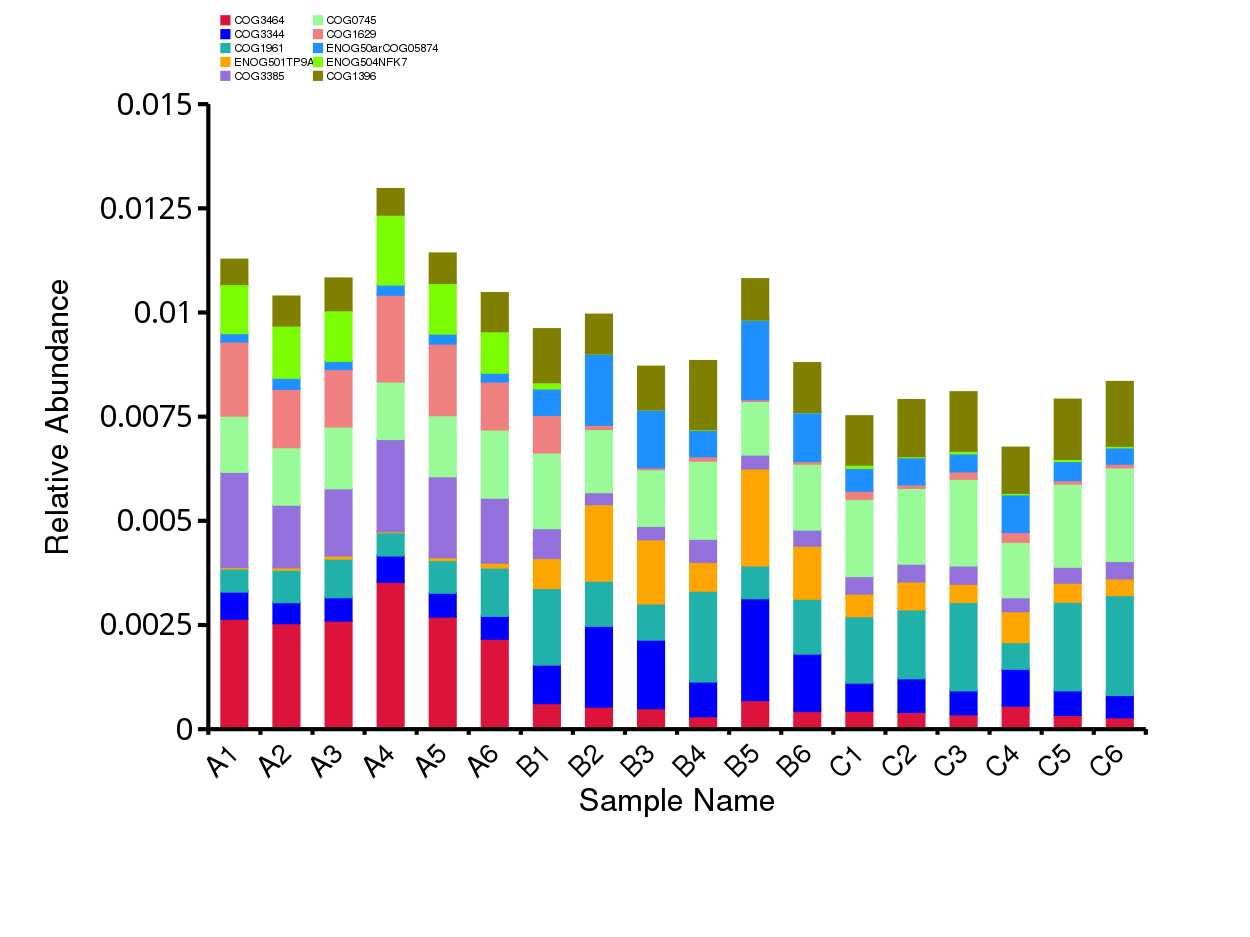
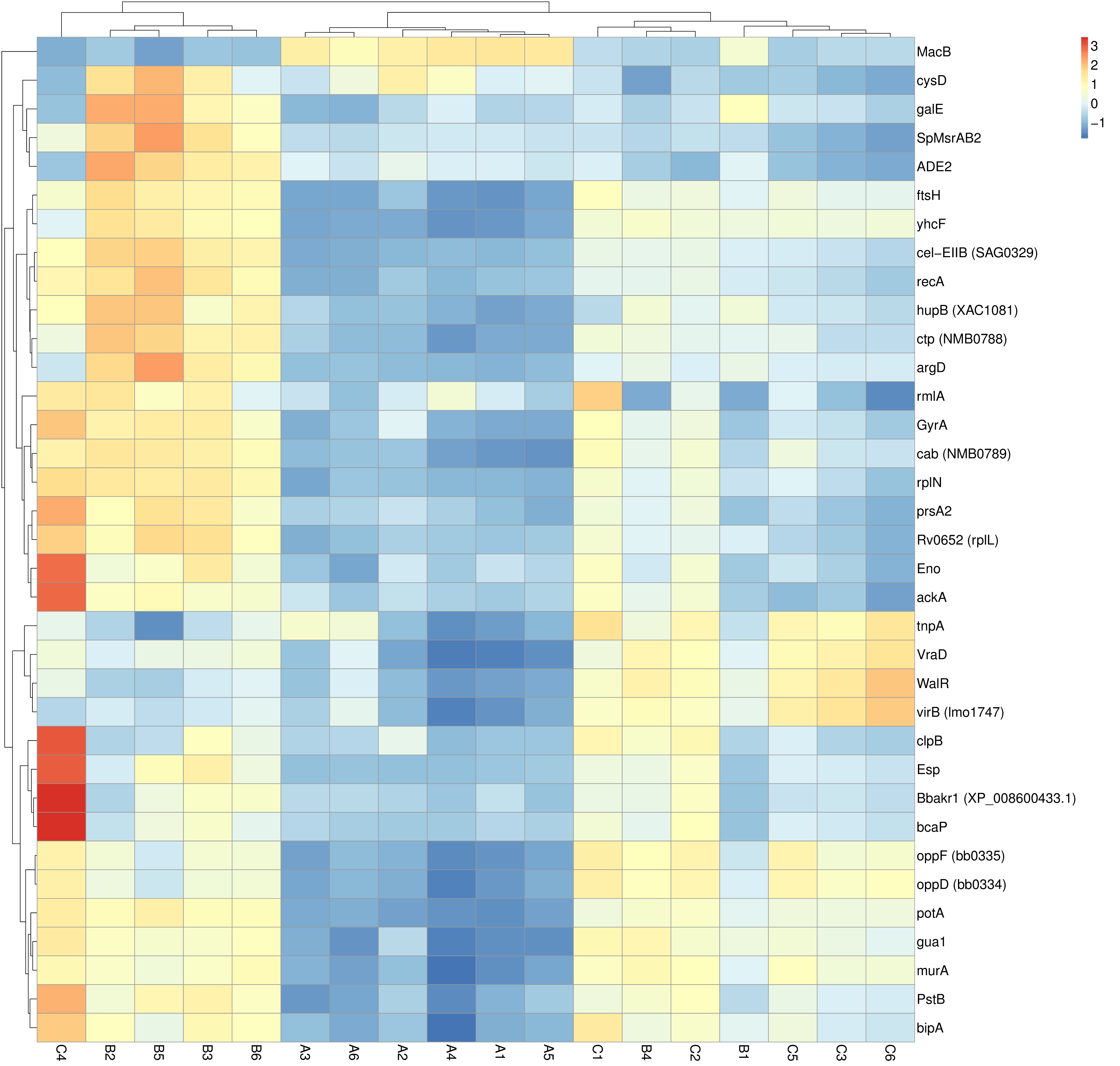
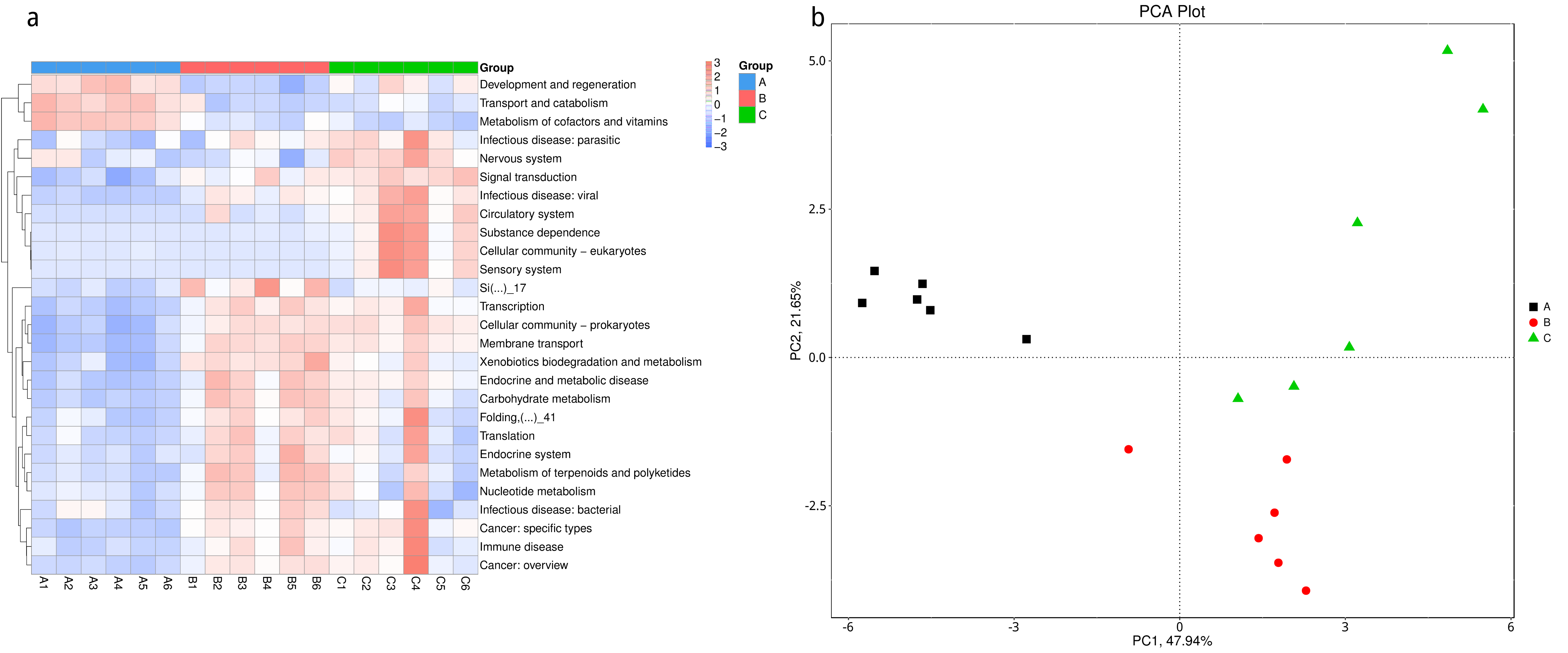
4.4.2.4 KEGG 功能相对丰度聚类分析
根据所有样品在KEGG数据库中的功能注释及丰度信息,选取丰度排名前 35 的功能及它们在每个样品中的丰度信息绘制热图,并从功能差异层面进行聚类。
图4.11 KEGG 功能丰度聚类热图
说明:横向为样品信息;纵向为KEGG注释信息;图中左侧的聚类树为功能聚类树;中间热图对应的值为每一行功能相对丰度经过标准化处理后得到的 Z 值;即一个样品在某个分类上的 Z 值为样品在该分类上的相对丰度和所有样品在该分类的平均相对丰度的差除以所有样品在该分类上的标准差所得到的值
结果目录:
KEGG丰度聚类热图见:result/04.Annotation/KEGG/Heatmap_sample/*/heatmap.sample.*.{pdf,png}
4.4.3 eggNOG 直系同源蛋白注释
EggNOG数据库是利用Smith-Waterman 比对算法对构建的基因直系同源簇(Orthologous Groups)进行功能注释,eggNOG涵盖了5,090个物种的基因,构建了约440万个Orthologous Groups。
4.4.3.1 eggNOG 注释基本步骤
使用DIAMOND软件将unigene与eggNOG功能数据库进行比对(Li et al., 2014; Feng et al., 2015),然后进行比对结果过滤,对于每一条序列的 比对结果,选取score 最高的比对结果(one HSP > 60 bits)进行后续分析(Qin et al., 2012; Li et al., 2014; Qin et al., 2014; Bäckhed et al, 2015)。
4.4.3.2 eggNOG 注释基因数目统计
从 unigene注释结果出发,绘制eggNOG数据库的level1层级注释基因数目统计图,展示结果如下图所示:
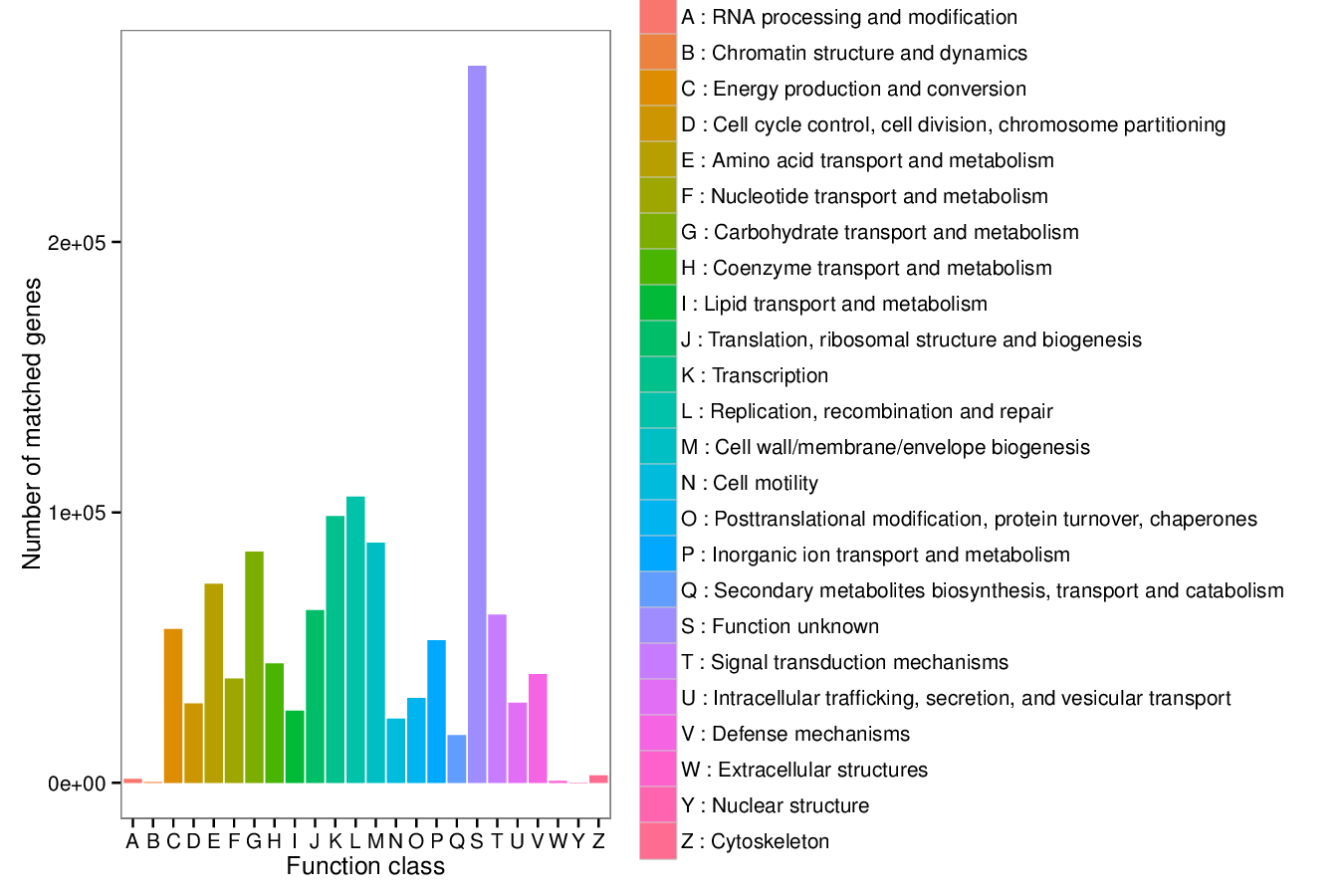
图4.12 EggNOG数据库注释基因数目统计图
结果目录:
eggNOG数据库注释基因数目统计图见:result/04.Annotation/eggNOG/Anno/eggNOG.unigenes.num.{pdf,png}
4.4.3.3 eggNOG 相对丰度概况
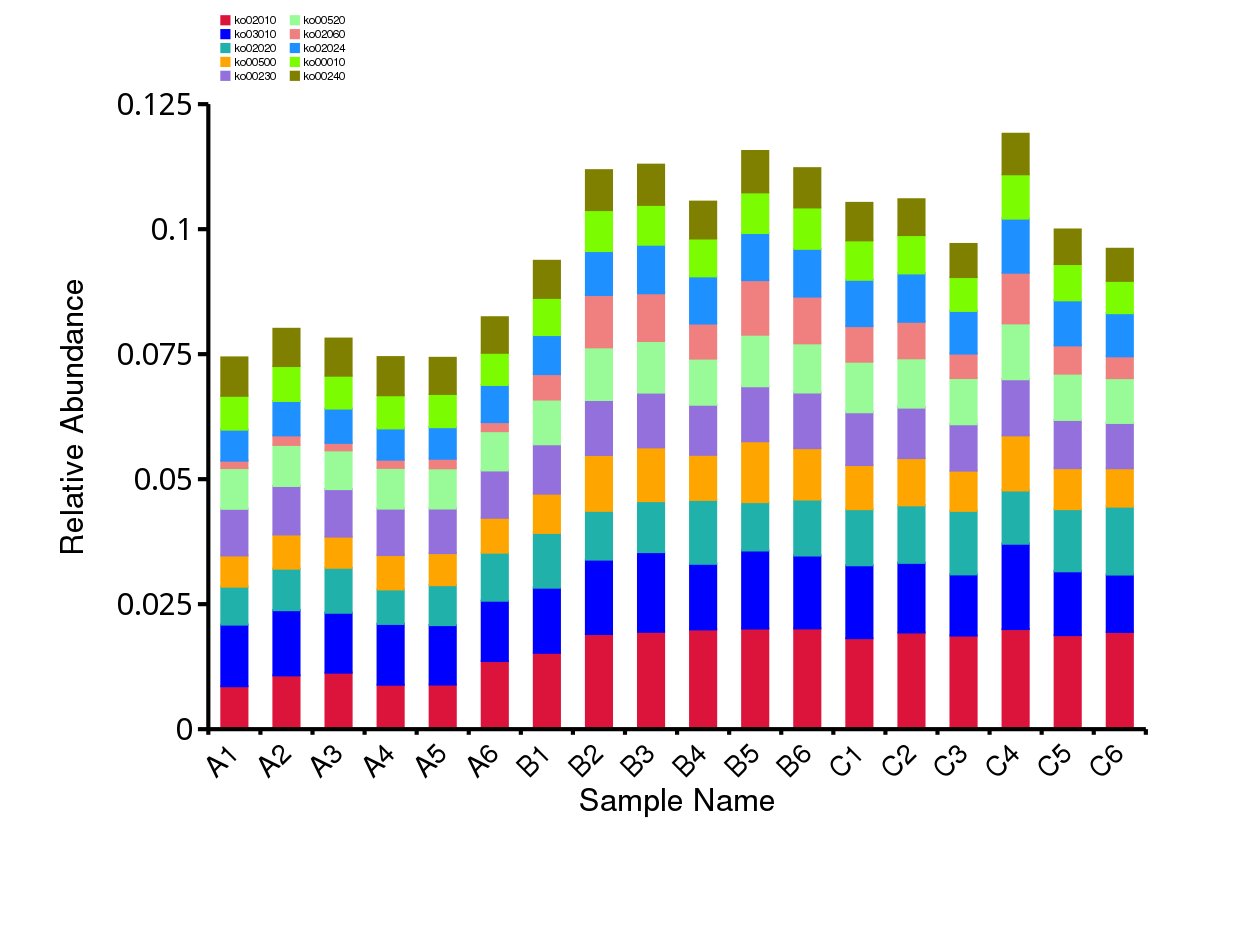
根据eggNOG数据库的注释结果,绘制了样品(组)在eggNOG数据库中对应层级上的相对丰度统计图。以各样品在eggNOG数据库中level1层级上的相对丰度统计图为例展示。其余层级为相对丰度前10的注释结果。
图4.13 eggNOG 功能注释相对丰度柱形图
说明:每个层级的相对丰度柱形图,横向表示样品名称,纵向表示注释到某类型功能的相对比例;各颜色区块对应的功能类别见图例
结果目录:
样品 - eggNOG各层级相对丰度前10的相对丰度柱形图见:result/04.Annotation/eggNOG/Top_sample/*/*.top10.{svg,png}
组 - eggNOG各层级相对丰度前10的相对丰度柱形图见:result/04.Annotation/eggNOG/Top_group1/*/*.top10.{svg,png}
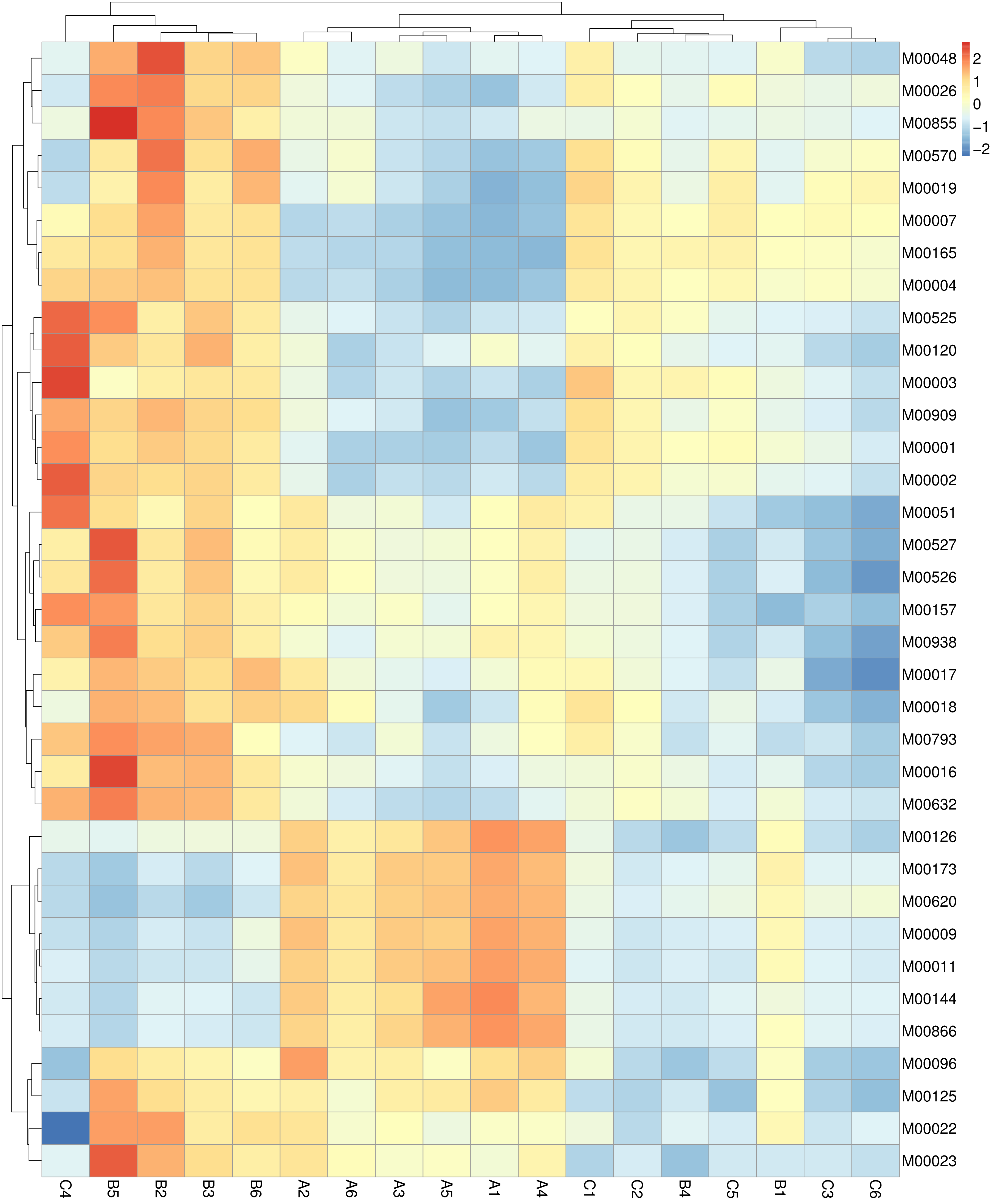
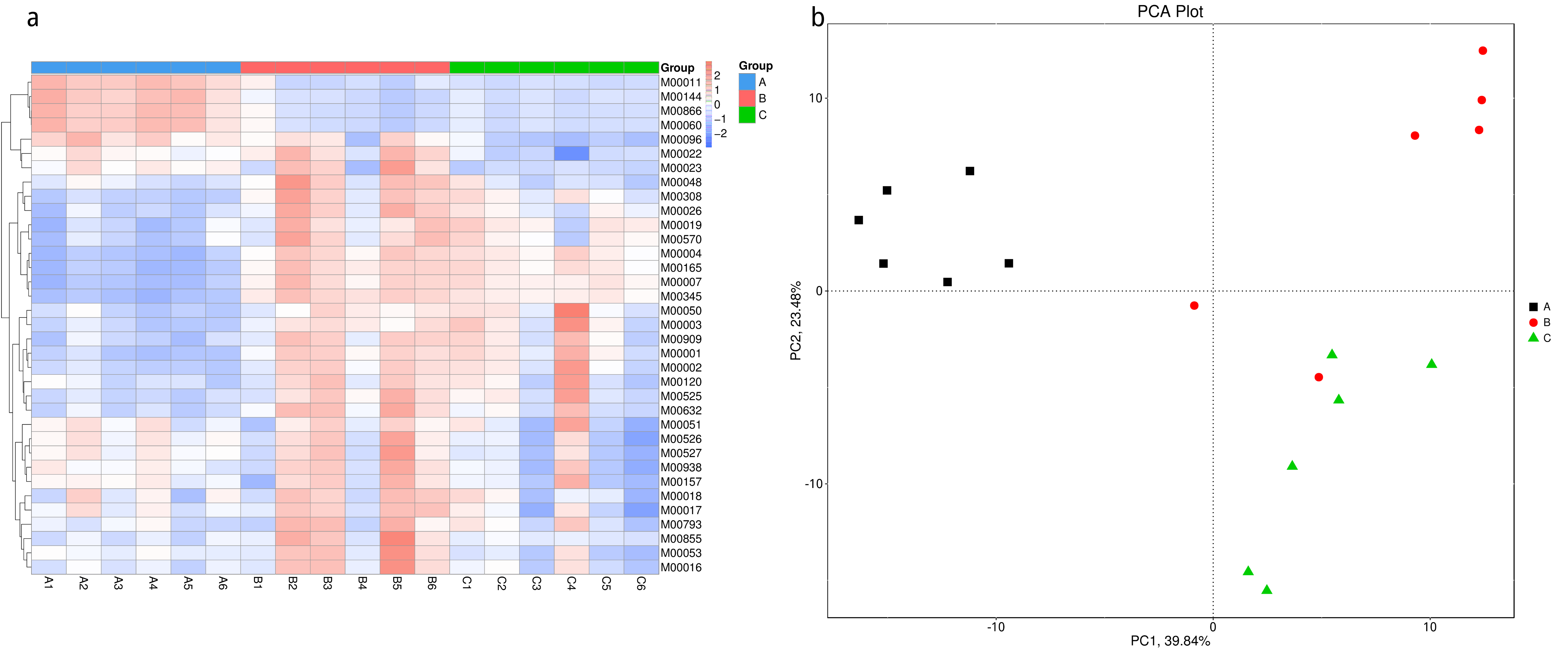
4.4.3.4 eggNOG 功能相对丰度聚类分析
根据所有样品在eggNOG数据库中的功能注释及丰度信息,选取丰度排名前 35 的功能及它们在每个样品中的丰度信息绘制热图,并从功能差异层面进行聚类。
图4.14 eggNOG 功能丰度聚类热图
说明:横向为样品信息;纵向为eggNOG注释信息;图中左侧的聚类树为功能聚类树;中间热图对应的值为每一行功能相对丰度经过标准化处理后得到的 Z 值;即一个样品在某个分类上的 Z 值为样品在该分类上的相对丰度和所有样品在该分类的平均相对丰度的差除以所有样品在该分类上的标准差所得到的值
结果目录:
eggNOG丰度聚类热图见:result/04.Annotation/eggNOG/Heatmap_sample/*/heatmap.sample.*.{pdf,png}
4.4.4 CAZy 碳水化合物活性酶注释
CAZy数据库是研究碳水化合物酶的专业级数据库,主要涵盖 6 大功能类:糖苷水解酶(Glycoside Hydrolases ,GHs),糖基转移酶(Glycosyl Transferases,GTs),多糖裂合酶(Polysaccharide Lyases,PLs),碳水化合物酯酶(Carbohydrate Esterases,CEs),辅助氧化还原酶(Auxiliary Activities , AAs)和碳水化合物结合模块(Carbohydrate-Binding Modules, CBMs)。
4.4.4.1 注释基本步骤
使用DIAMOND软件将 unigene与CAZy功能数据库进行比对(Li et al., 2014; Feng et al., 2015),然后进行比对结果过滤,对于每一条序列的比对结果,选取score 最高的比对结果(one HSP > 60 bits)进行后续分析(Qin et al., 2012; Li et al., 2014; Qin et al., 2014; Bäckhed et al, 2015)。
从功能注释结果及基因丰度表出发,获得各个样品在各个分类层级上的基因数目表,对于某个功能在某个样品中的基因数目,等于在注释为该功能的基因中,丰度不为 0 的基因数目。
4.4.4.2 CAZy 注释基因数目统计
从unigene注释结果出发,绘制CAZy数据库level1层级的注释基因数目统计图,展示结果如下图所示:
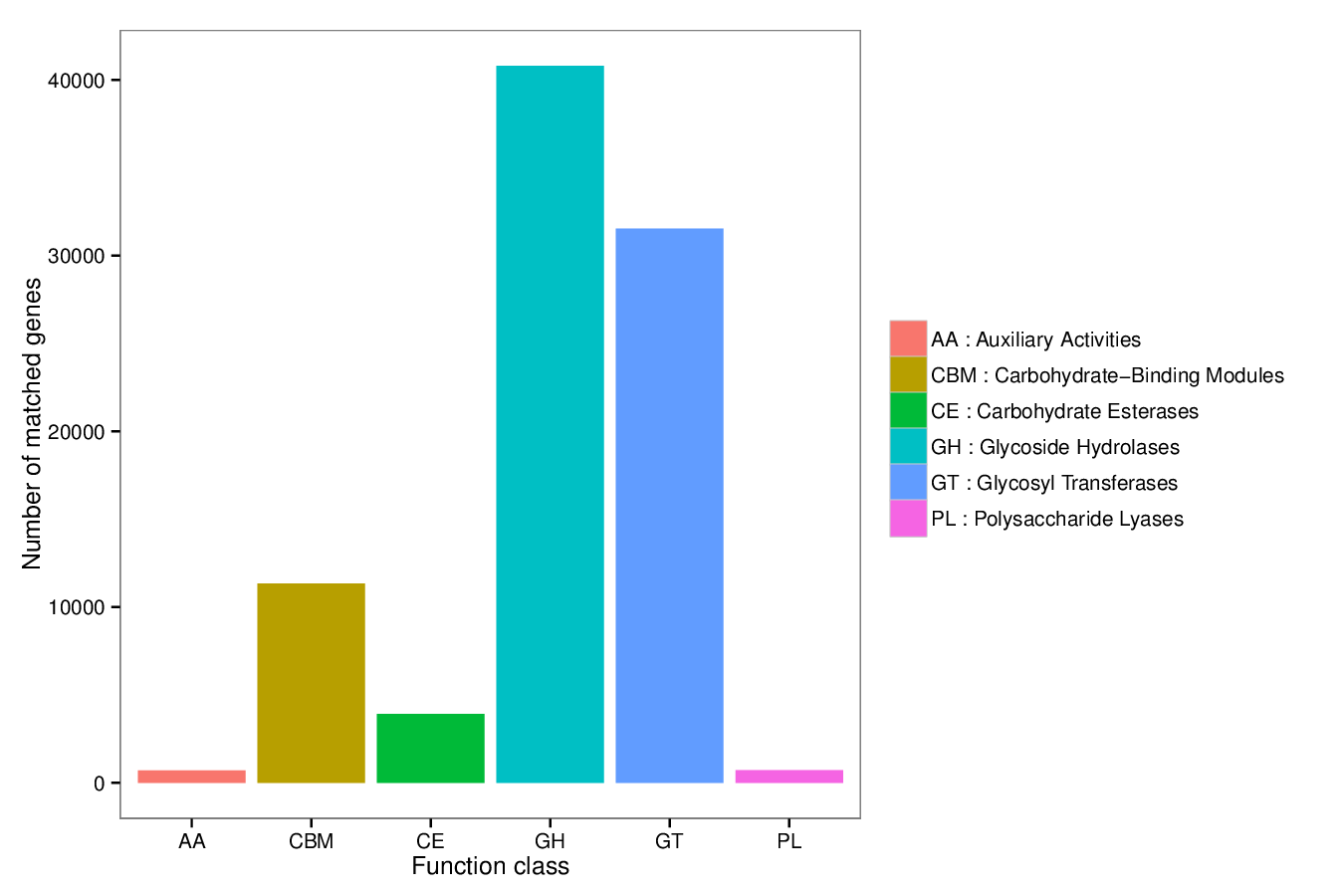
图4.15 CAZy数据库注释基因数目统计图
结果目录:
CAZy数据库注释基因数目统计图见:result/04.Annotation/CAZy/Anno/CAZy.unigenes.num.{pdf,png}
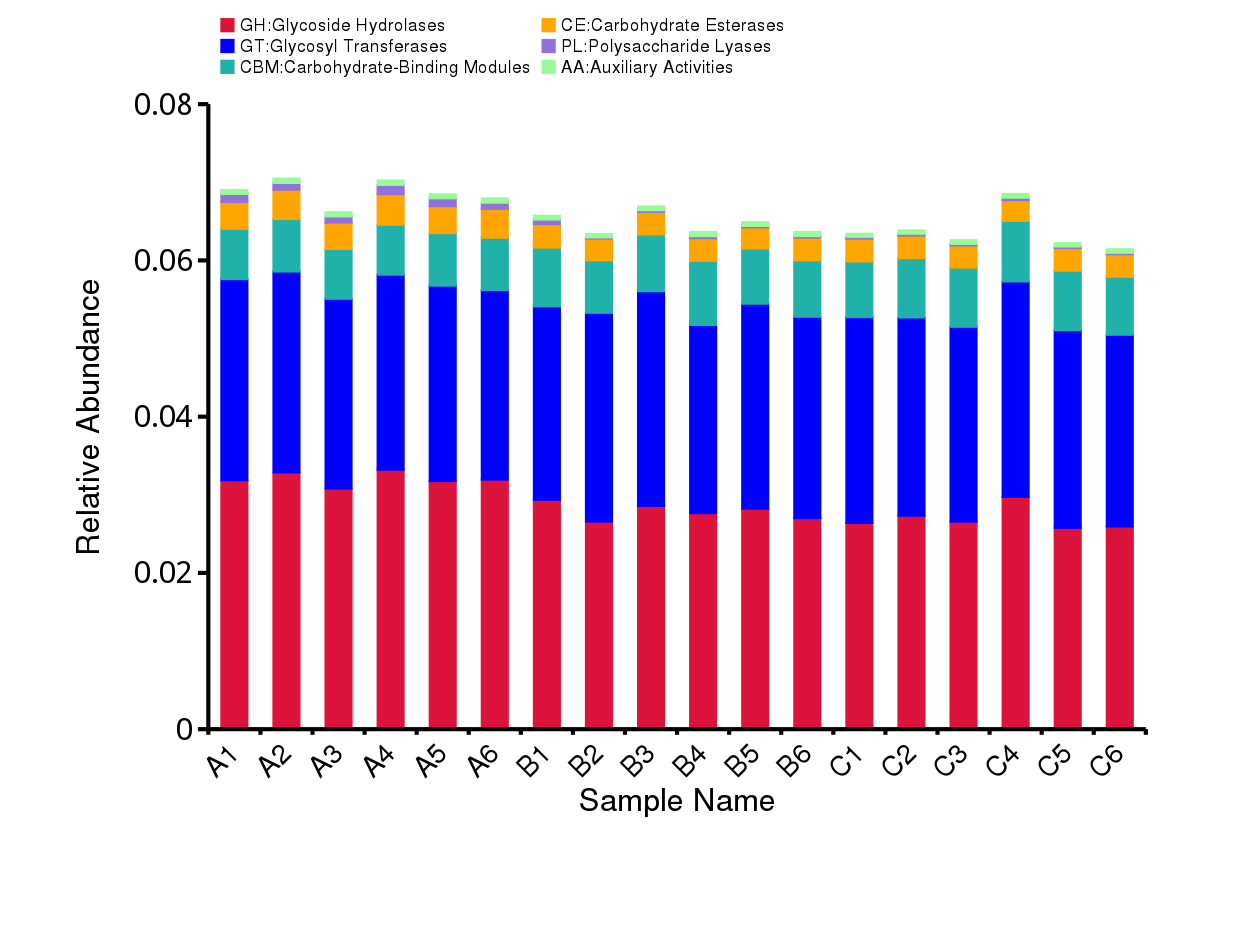
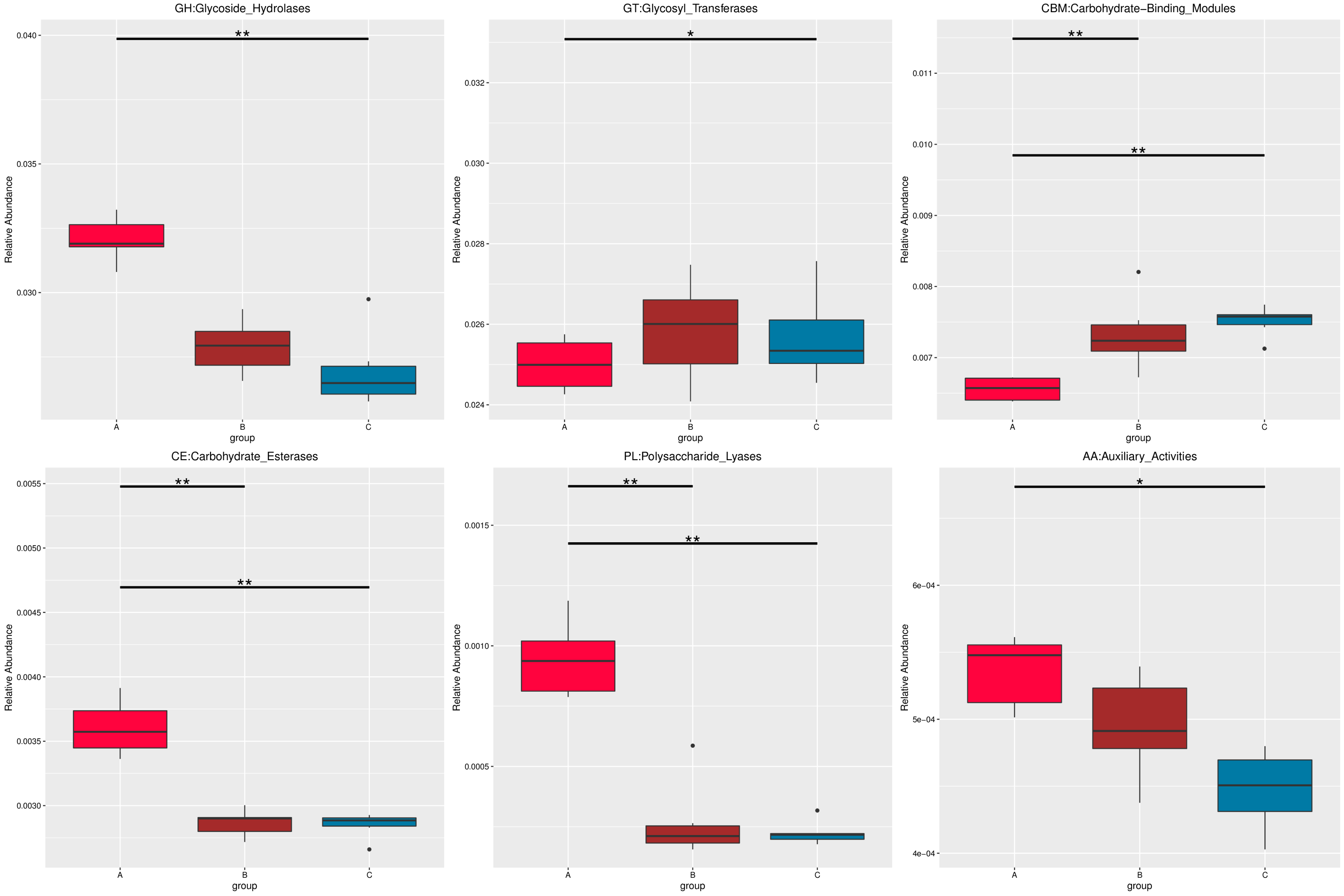
4.4.4.3 CAZy 相对丰度概况
根据CAZy数据库的注释结果,绘制了样品(组)在CAZy数据库中对应层级上的相对丰度统计图。 以各样品在CAZy数据库中level1 层级上的相对丰度统计图为例展示。其余层级为相对丰度前10的注释结果。
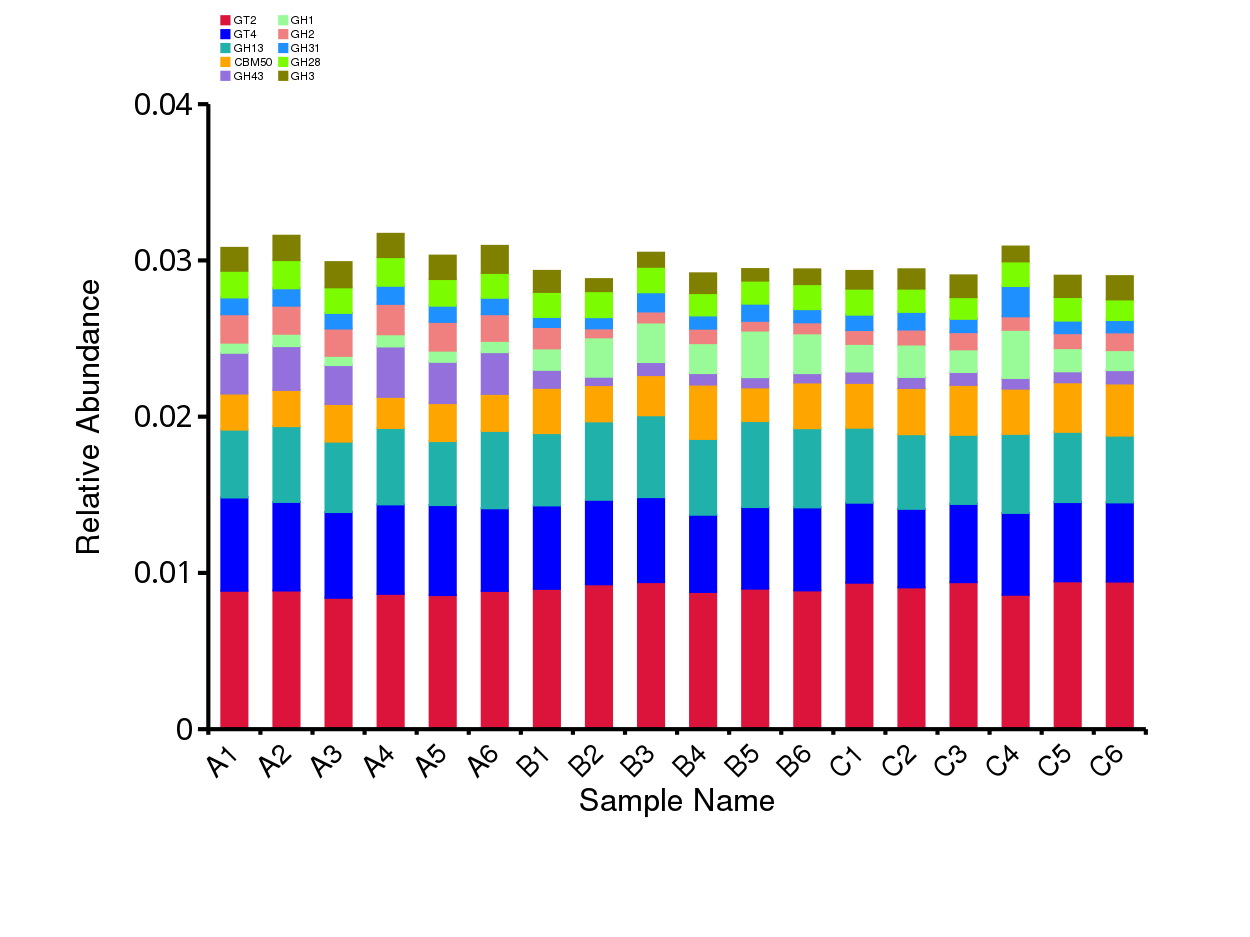
图4.16 CAZy 功能注释相对丰度柱形图
说明:每个层级的相对丰度柱形图,横向表示样品名称,纵向表示注释到某类型功能的相对比例;各颜色区块对应的功能类别见图例
结果目录:
样品 - CAZy各层级相对丰度前10的相对丰度柱形图见:result/04.Annotation/CAZy/Top_sample/*/*.top10.{svg,png}
组 - CAZy各层级相对丰度前10的相对丰度柱形图见:result/04.Annotation/CAZy/Top_group1/*/*.top10.{svg,png}
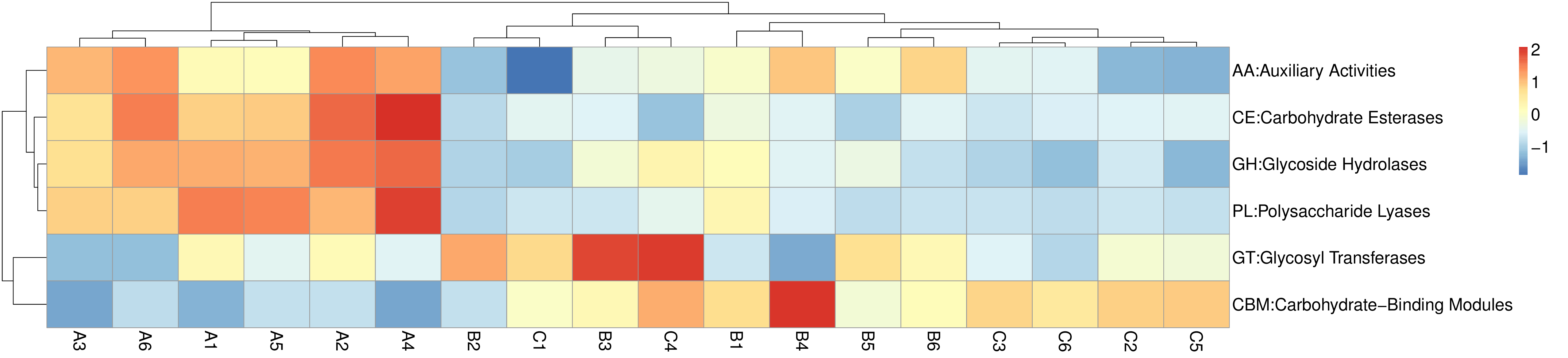
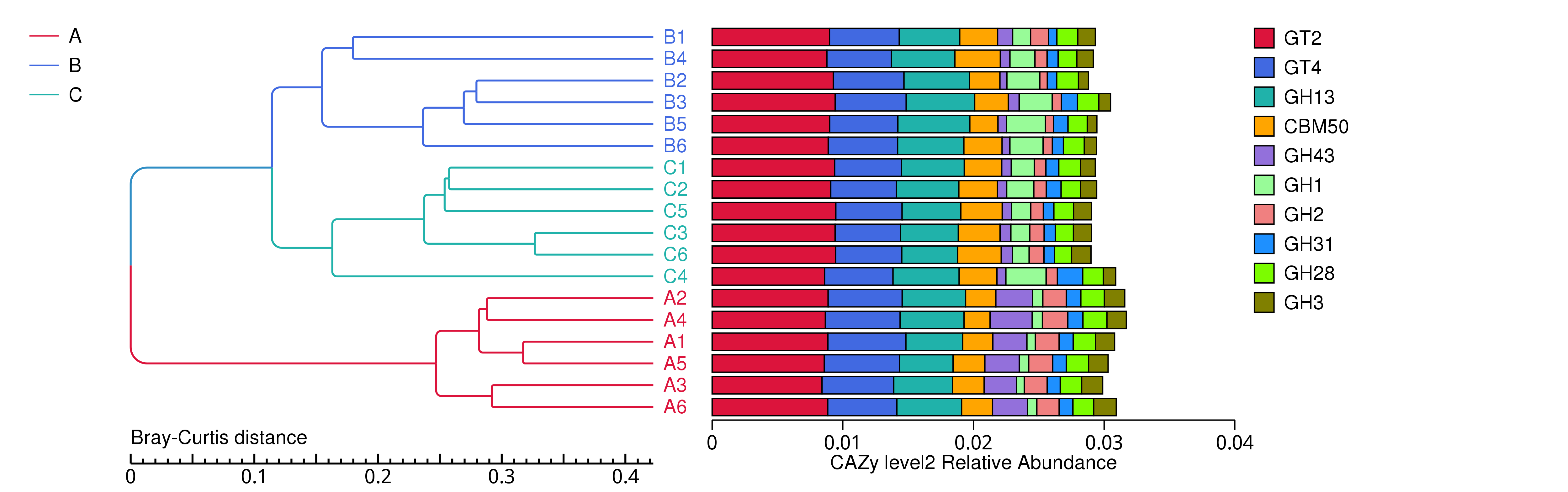
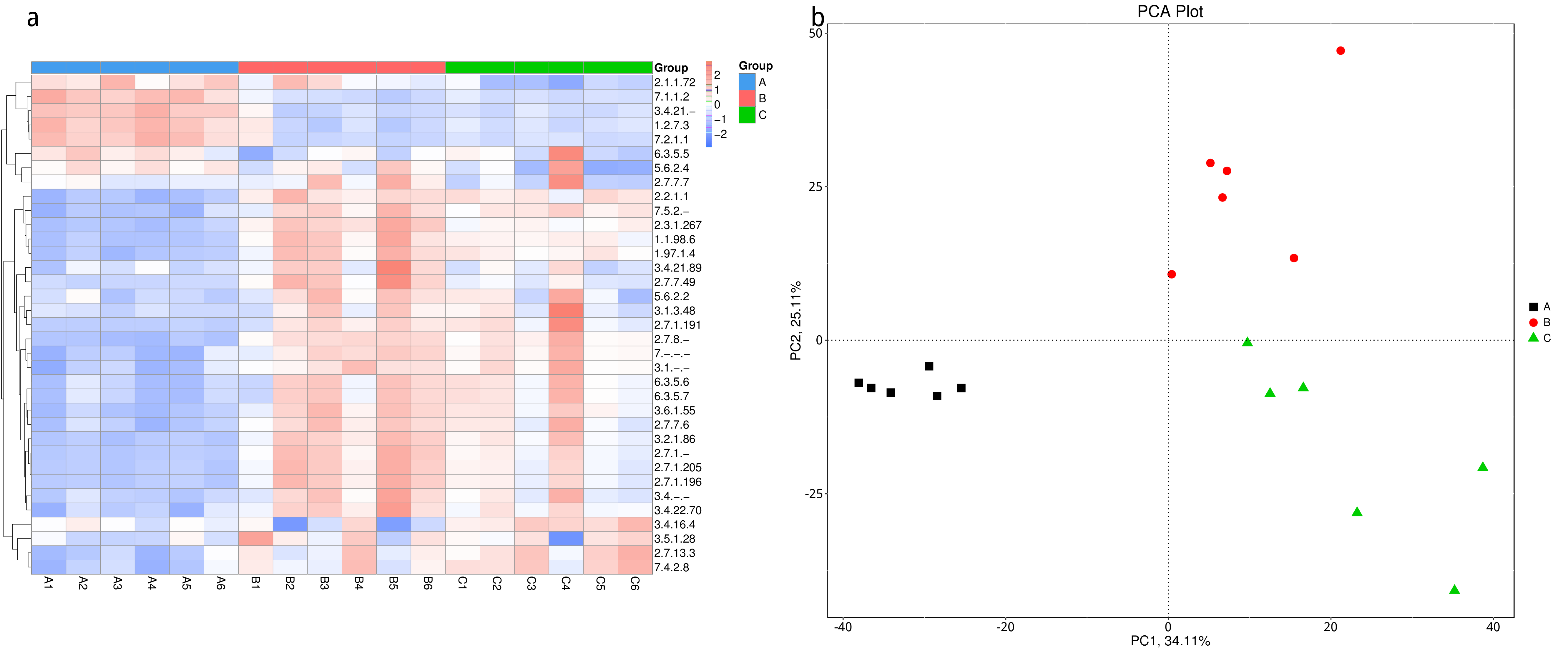
4.4.4.4 CAZy 功能相对丰度聚类分析
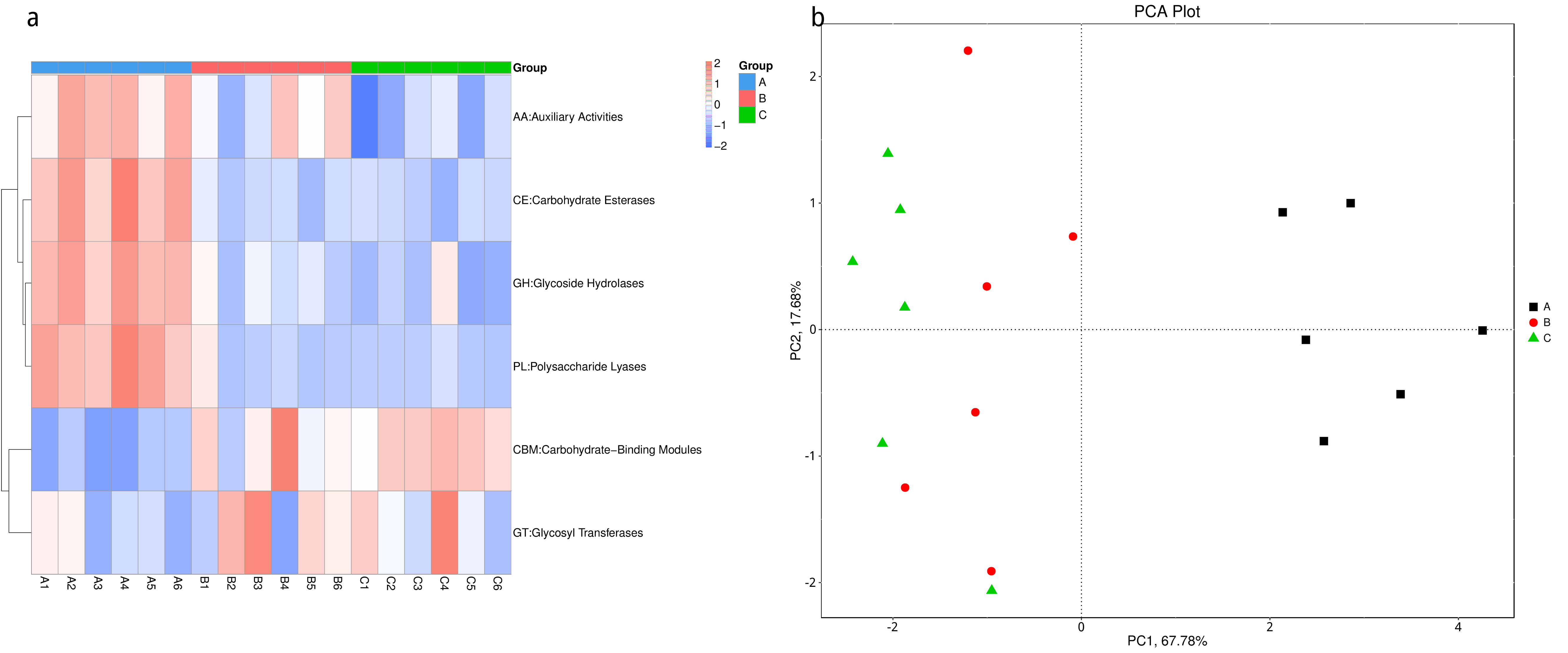
根据所有样品在CAZy数据库中的功能注释及丰度信息,选取丰度排名前 35 的功能及它们在每个样品中的丰度信息绘制热图,并从功能差异层面进行聚类。
图4.17 CAZy 功能丰度聚类热图
说明:横向为样品信息;纵向为CAZy注释信息;图中左侧的聚类树为功能聚类树;中间热图对应的值为每一行功能相对丰度经过标准化处理后得到的 Z 值;即一个样品在某个分类上的 Z 值为样品在该分类上的相对丰度和所有样品在该分类的平均相对丰度的差除以所有样品在该分类上的标准差所得到的值
结果目录:
CAZy丰度聚类热图见:result/04.Annotation/CAZy/Heatmap_sample/*/heatmap.sample.*.{pdf,png}
4.4.5 VFDB 直系同源蛋白注释
细菌致病菌毒力因子数据库VFDB(Virulence Factors of Pathogenic Bacteria),是致病细菌、衣原体和支原体的毒力因子数据库,除收录毒力基因的物种信息、基本特征描述外,还提供毒力基因功能和致病机制的详细描述。其包含26个种,共459个致病因子,24个致病岛,2,505个与毒力因子相关的基因。
4.4.5.1 VFDB 注释基本步骤
使用DIAMOND软件将unigene与VFDB功能数据库进行比对(Li et al., 2014; Feng et al., 2015),然后进行比对结果过滤,对于每一条序列的 比对结果,选取 score 最高的比对结果(one HSP > 60 bits)进行后续分析(Qin et al., 2012; Li et al., 2014; Qin et al., 2014; Bäckhed et al, 2015)。
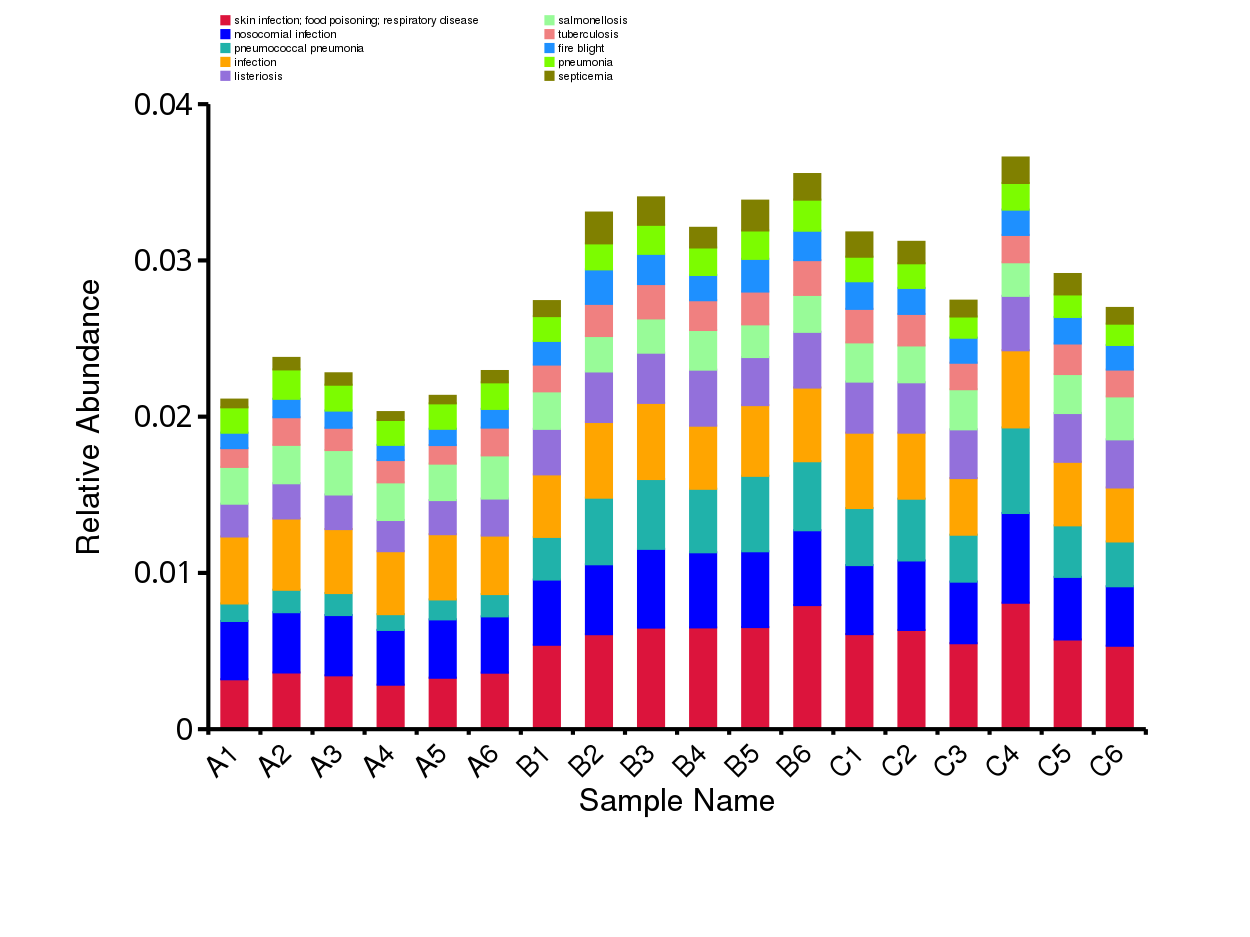
4.4.5.2 VFDB 相对丰度概况
根据VFDB数据库的注释结果,绘制了样品(组)在VFDB数据库中对应层级上的相对丰度统计图。 以各样品在VFDB数据库中level1 层级上的相对丰度统计图为例展示。其余层级为相对丰度前10的注释结果。
图4.18 VFDB 功能注释相对丰度柱形图
说明:每个层级的相对丰度柱形图,横向表示样品名称,纵向表示注释到某类型功能的相对比例;各颜色区块对应的功能类别见图例
结果目录:
样品 - VFDB各层级相对丰度前10的相对丰度柱形图见:result/04.Annotation/VFDB/Top_sample/*/*.top10.{svg.png}
组 - VFDB各层级相对丰度前10的相对丰度柱形图见 : result/04.Annotation/VFDB/Top_group1/*/*.top10.{svg.png}
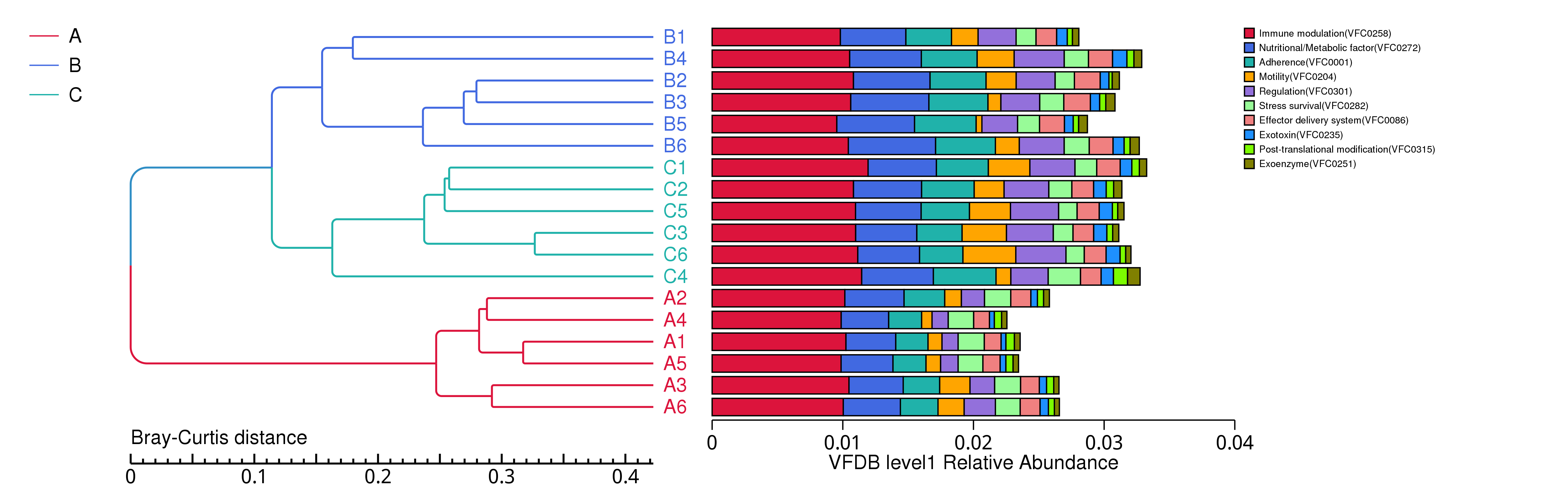
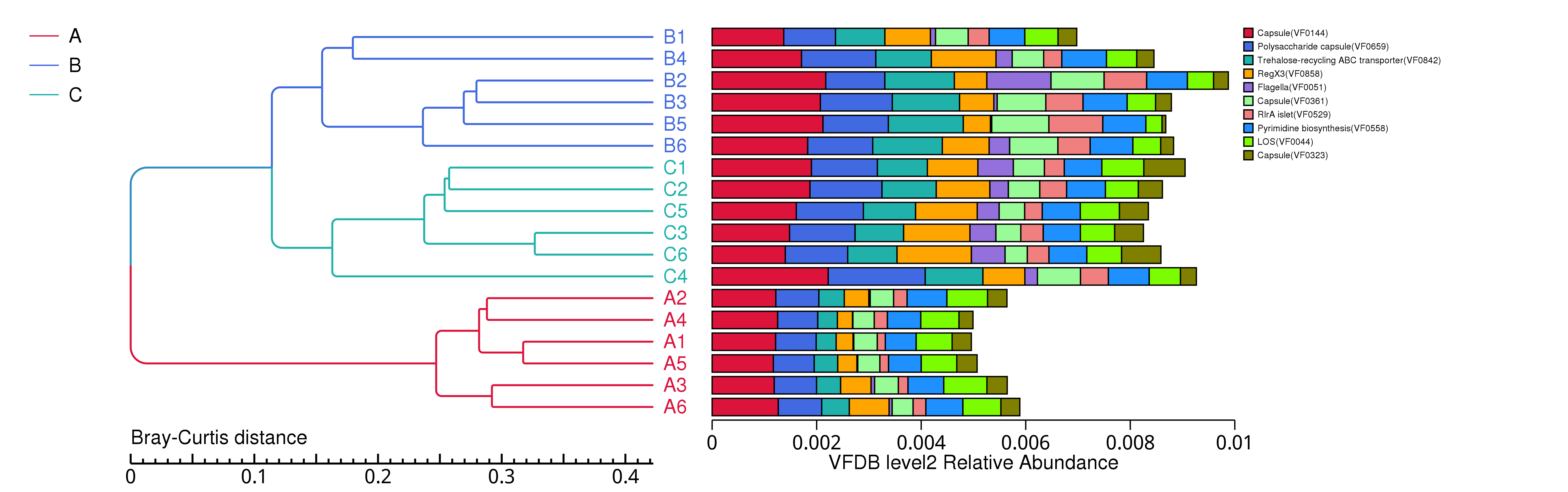
4.4.5.3 VFDB 功能相对丰度聚类分析
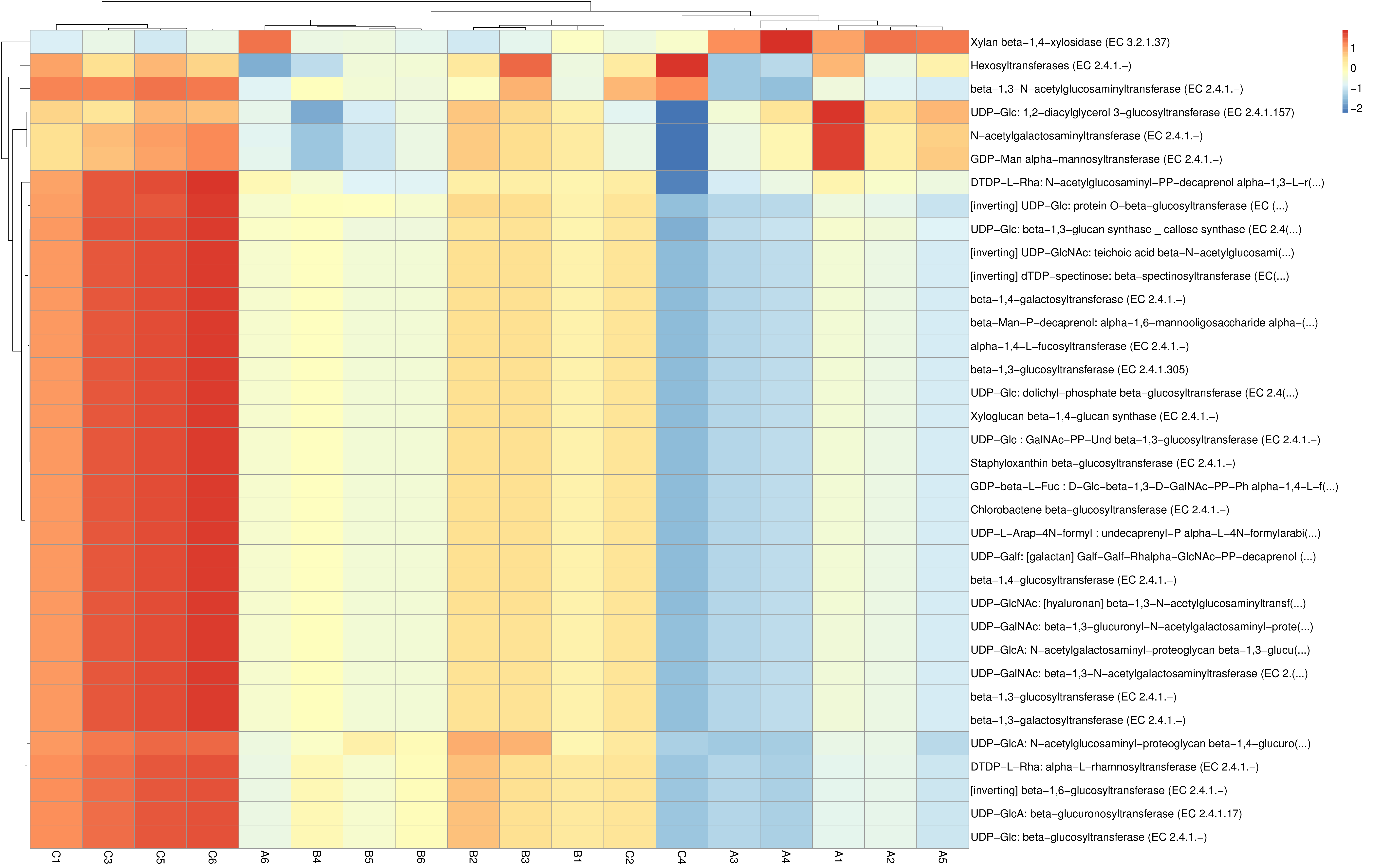
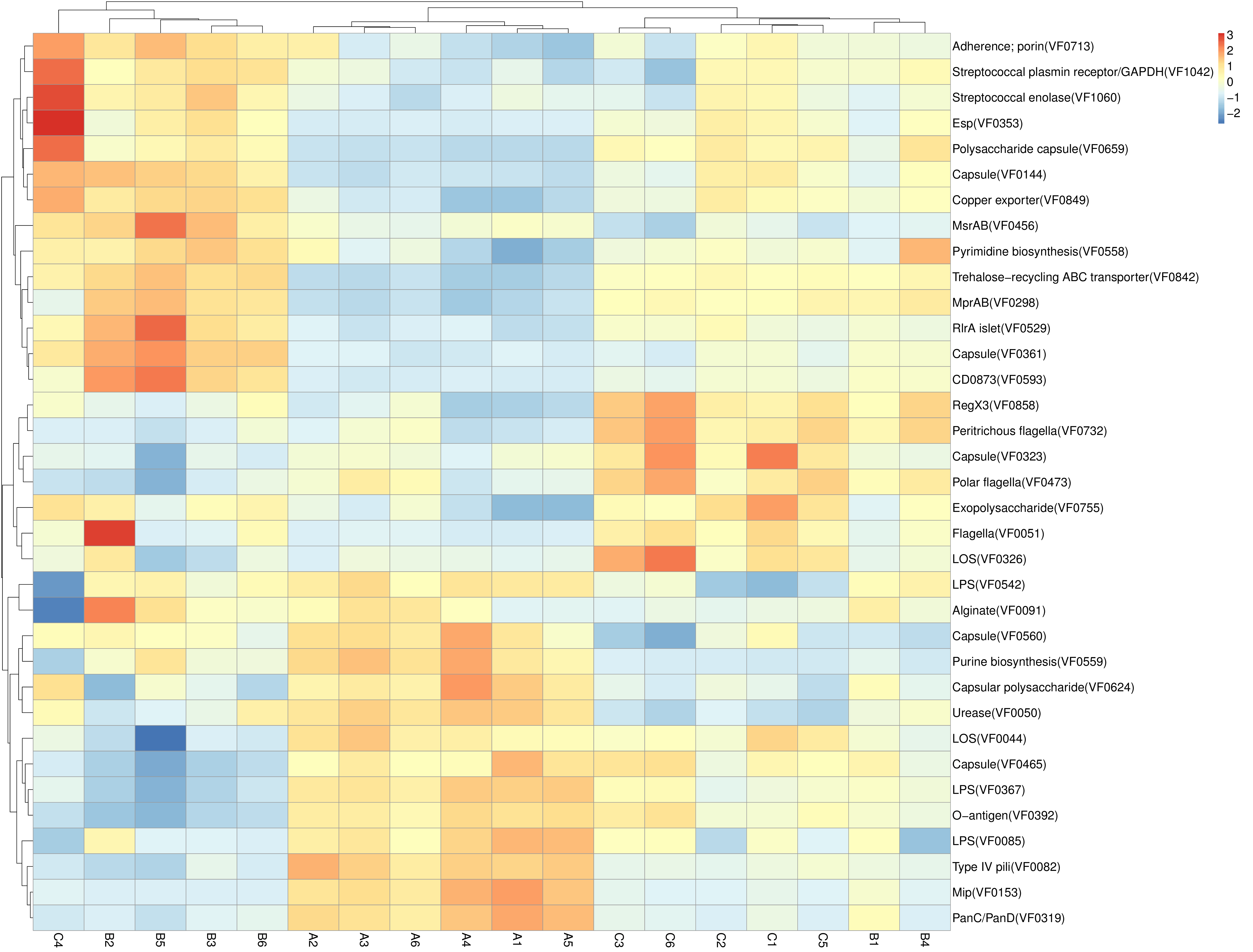
根据所有样品在VFDB数据库中的功能注释及丰度信息,选取丰度排名前 35 的功能及它们在每个样品中的丰度信息绘制热图,并从功能差异层面进行聚类。
图4.19 VFDB 功能丰度聚类热图
说明:横向为样品信息;纵向为VFDB注释信息;图中左侧的聚类树为功能聚类树;中间热图对应的值为每一行功能相对丰度经过标准化处理后得到的 Z 值;即一个样品在某个分类上的 Z 值为样品在该分类上的相对丰度和所有样品在该分类的平均相对丰度的差除以所有样品在该分类上的标准差所得到的值
结果目录:
VFDB丰度聚类热图见:result/04.Annotation/VFDB/Heatmap_sample/*/heatmap.sample.*.{pdf,png}
4.4.6 PHI 直系同源蛋白注释
PHI数据库(Pathogen-Host Interaction database)是一个专注于病原体-宿主相互作用的数据库。它收集、整理和提供了各种病原体(如细菌、病毒、真菌和寄生虫等)与宿主(包括人类和其他动植物)之间相互作用的信息。
4.4.6.1 PHI 注释基本步骤
使用DIAMOND软件将unigene与PHI功能数据库进行比对(Li et al., 2014; Feng et al., 2015),然后进行比对结果过滤,对于每一条序列的比对结果,选取 score 最高的比对结果(one HSP > 60 bits)进行后续分析(Qin et al., 2012; Li et al., 2014; Qin et al., 2014; Bäckhed et al, 2015)。
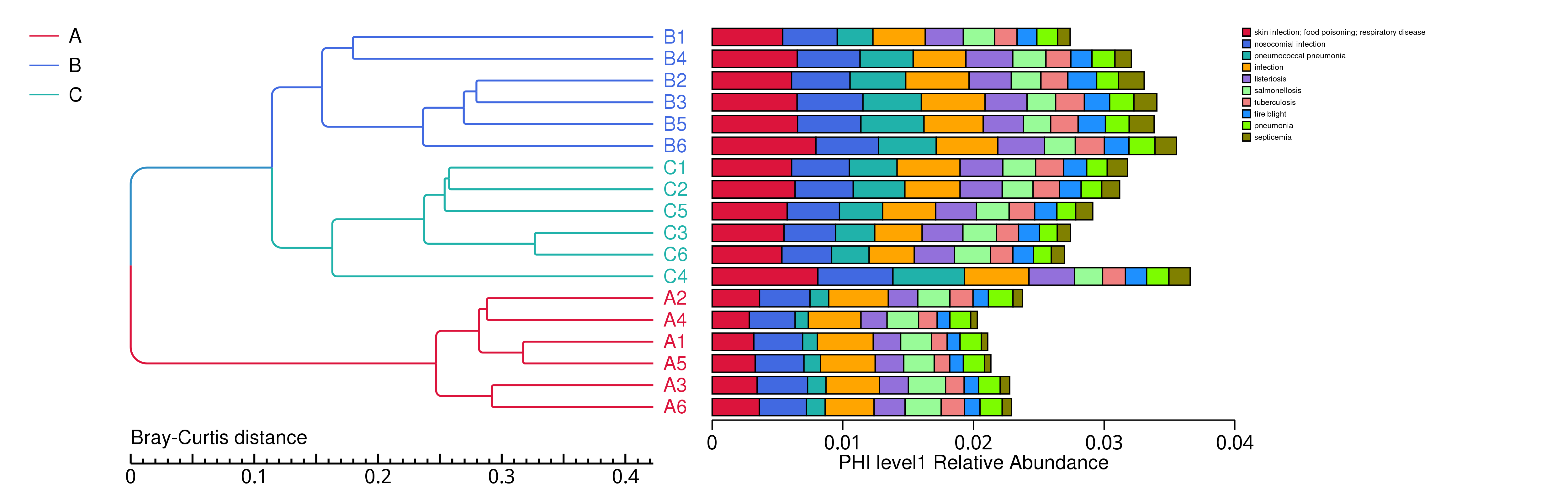
4.4.6.2 PHI 相对丰度概况
根据PHI数据库的注释结果,绘制了样品在PHI 数据库中对应层级上的相对丰度统计图。 以各样品在PHI数据库中level1 层级上的相对丰度统计图为例展示。其余层级为相对丰度前10的注释结果。
图4.20 PHI 功能注释相对丰度柱形图
说明:每个层级的相对丰度柱形图,横向表示样品名称,纵向表示注释到某类型功能的相对比例;各颜色区块对应的功能类别见图例
结果目录:
样品 - PHI各层级相对丰度前10的相对丰度柱形图 见 : result/04.Annotation/PHI/Top_sample/*/*.top10.{svg,png}
组 - PHI各层级相对丰度前10的相对丰度柱形图见 : result/04.Annotation/PHI/Top_group1/*/*.top10.{svg,png}
4.4.6.3 PHI 功能相对丰度聚类分析
根据所有样品在PHI数据库中的功能注释及丰度信息,选取丰度排名前 35 的功能及它们在每个样品中的丰度信息绘制热图,并从功能差异层面进行聚类。
图4.21 PHI 功能丰度聚类热图
说明:横向为样品信息;纵向为PHI注释信息;图中左侧的聚类树为功能聚类树;中间热图对应的值为每一行功能相对丰度经过标准化处理后得到的 Z 值;即一个样品在某个分类上的 Z 值为样品在该分类上的相对丰度和所有样品在该分类的平均相对丰度的差除以所有样品在该分类上的标准差所得到的值
结果目录:
PHI丰度聚类热图见:result/04.Annotation/PHI/Heatmap_sample/*/heatmap.sample.*.{pdf,png}
4.5 物种与功能多样性分析
4.5.1 Alpha多样性分析
4.5.1.1 Micro_NR
从物种注释 Absolute 表出发,计算得到每个样本内物种的丰富度以及多样性。该表仅展示 species 层级结果,其他层级见结果文件。
表4.4 Alpha多样性分析统计结果
| Sample | ACE | chao1 | shannon | simpson | observed_species | goods_coverage |
|---|---|---|---|---|---|---|
| A1 | 4153.16211857023 | 4192.97540983607 | 4.23652508774237 | 0.958209284387269 | 3989 | 1 |
| A2 | 4602.73575329709 | 4635.43961352657 | 4.50987271592601 | 0.959378874314839 | 4469 | 1 |
| A3 | 4788.9436452936 | 4853.04918032787 | 4.64437045044106 | 0.969292478414934 | 4659 | 1 |
| A4 | 3423.94758375198 | 3485.5303030303 | 3.96051157512467 | 0.940471347191081 | 3268 | 1 |
| A5 | 4632.57530473899 | 4642.5 | 4.56171871530976 | 0.967498440202328 | 4521 | 1 |
| A6 | 4918.94735285027 | 4962.80597014925 | 4.44206779235049 | 0.951241490383073 | 4794 | 1 |
| B1 | 5518.4225119167 | 5563.82258064516 | 4.50076093446646 | 0.935731650056453 | 5416 | 1 |
| B2 | 4764.9617400887 | 4805.17374517375 | 2.65115165076833 | 0.68023414806216 | 4625 | 1 |
| B3 | 4784.89214043213 | 4829.84435797665 | 2.90484187183182 | 0.77064545616734 | 4630 | 1 |
| B4 | 5544.13245194484 | 5570.5 | 4.39565960090137 | 0.951783196735385 | 5469 | 1 |
| B5 | 4196.11300437445 | 4298.1013215859 | 2.1159518202566 | 0.609310388100981 | 3999 | 1 |
| B6 | 5043.2503030825 | 5090.16901408451 | 3.48569723474056 | 0.838059772573625 | 4912 | 1 |
| C1 | 5168.35162106894 | 5190.424 | 4.49203323759425 | 0.96721798846755 | 5043 | 1 |
| C2 | 5270.91753977194 | 5308.20873786408 | 4.4582227679025 | 0.960715860494338 | 5151 | 1 |
| C3 | 5576.1807877487 | 5606.85534591195 | 4.73414115765239 | 0.972438698767873 | 5510 | 1 |
| C4 | 4851.77395793166 | 4884.05882352941 | 3.73204558562936 | 0.921457130317677 | 4714 | 1 |
| C5 | 5317.7939557869 | 5331.68181818182 | 4.6063846836177 | 0.968427081913359 | 5241 | 1 |
| C6 | 5614.77681166886 | 5663.54022988506 | 4.7139141440765 | 0.972334502038189 | 5520 | 1 |
显示注释
(1)ACE:估计群落中物种数目。
(2)chao1:估计群落样品中包含的物种总数,群落中低丰度物种越多,chao1指数越大。
(3)shannon:样品中的分类总数及其占比。群落多样性越高,物种分布越均匀,shannon指数越大。
(4)simpson:表征群落内物种分布的多样性和均匀度,物种均匀度越好,Simpson指数越大。
(5)observed_species:直观观测到的物种数目,指数越大观测到的物种越多。
(6)goods_coverage:覆盖度,测序覆盖度越高,指数越大。
结果目录:
Micro_NR Alpha多样性指数汇总表见:result/05.Diversity/MicroNR/AlphaDiversity/Alpha_index_table/tax_Alphaindex.*.xls
4.5.1.2 KEGG
从功能注释 Absolute 表出发,计算得到每个样本内物种的丰富度以及多样性。该表仅展示 level3 层级结果,其他层级见结果文件。
表4.5 Alpha多样性分析统计结果
| Sample | ACE | chao1 | shannon | simpson | observed_species | goods_coverage |
|---|---|---|---|---|---|---|
| A1 | 273.707444598338 | 273 | 4.77411934855941 | 0.987928684299046 | 272 | 1 |
| A2 | 285 | 285 | 4.76457945770708 | 0.987648971413218 | 285 | 1 |
| A3 | 270 | 270 | 4.77166244174991 | 0.987768810460186 | 270 | 1 |
| A4 | 269 | 269 | 4.76710073185288 | 0.987827943762566 | 269 | 1 |
| A5 | 286.622402962375 | 285.5 | 4.77100544765721 | 0.987880727814654 | 283 | 1 |
| A6 | 283.714825816905 | 285.5 | 4.75105915789526 | 0.98720532799813 | 278 | 1 |
| B1 | 303.147321428571 | 303 | 4.72245567149193 | 0.98655069095923 | 303 | 1 |
| B2 | 305.441840277778 | 312 | 4.63637994828289 | 0.984859130862253 | 302 | 1 |
| B3 | 287 | 287 | 4.63189666241211 | 0.984668674272447 | 287 | 1 |
| B4 | 306.306818181818 | 304.333333333333 | 4.65682146718501 | 0.984927662405129 | 301 | 1 |
| B5 | 295.360248447205 | 295.5 | 4.60057688981784 | 0.984162491412753 | 295 | 1 |
| B6 | 297 | 297 | 4.66394302418658 | 0.985165454799683 | 297 | 1 |
| C1 | 415.929402573907 | 415.333333333333 | 4.67284280123368 | 0.985395128698675 | 415 | 1 |
| C2 | 415.53825 | 415 | 4.67590954874842 | 0.985014290363385 | 415 | 1 |
| C3 | 415 | 415 | 4.761726454503 | 0.985823169202734 | 415 | 1 |
| C4 | 412 | 412 | 4.68908642661952 | 0.984705702675436 | 412 | 1 |
| C5 | 414.302900128103 | 414 | 4.67650949443659 | 0.98521938619125 | 414 | 1 |
| C6 | 416 | 416 | 4.71185254502252 | 0.98526606576296 | 416 | 1 |
显示注释
(1)ACE:估计群落中功能数目。
(2)chao1:估计群落样品中包含的功能总数,群落中低丰度功能越多,chao1指数越大。
(3)shannon:样品中的分类总数及其占比。群落多样性越高,功能分布越均匀,shannon指数越大。
(4)simpson:表征群落内功能分布的多样性和均匀度,功能均匀度越好,Simpson指数越大。
(5)observed_species:直观观测到的功能数目,指数越大观测到的功能越多。
(6)goods_coverage:覆盖度,测序覆盖度越高,指数越大。
结果目录:
KEGG Alpha多样性指数汇总表见:result/05.Diversity/KEGG/AlphaDiversity/Alpha_index_table/KEGG_Alphaindex.*.xls
4.5.1.3 eggNOG
从功能注释 Absolute 表出发,计算得到每个样本内物种的丰富度以及多样性。该表仅展示 og 层级结果,其他层级见结果文件。
表4.6 Alpha多样性分析统计结果
| Sample | ACE | chao1 | shannon | simpson | observed_species | goods_coverage |
|---|---|---|---|---|---|---|
| A1 | 152504.599479114 | 153133.947639359 | 10.0308450107352 | 0.999855253951031 | 149285 | 1 |
| A2 | 162663.085761941 | 163204.680626546 | 10.2334392392994 | 0.999876439458422 | 159904 | 1 |
| A3 | 169126.640172966 | 170039.179750711 | 10.3798407715437 | 0.999881247443049 | 166868 | 1 |
| A4 | 132799.509769068 | 133354.936900056 | 9.81051100719566 | 0.999829571396549 | 129080 | 1 |
| A5 | 163229.388556906 | 163546.916968647 | 10.1513746017508 | 0.999861500677522 | 160807 | 1 |
| A6 | 174509.249480247 | 175136.448888889 | 10.4439069602209 | 0.99988742958355 | 172227 | 1 |
| B1 | 216442.692036418 | 217381.022929936 | 10.6687509218202 | 0.99989923521811 | 214258 | 1 |
| B2 | 188301.105426006 | 189445.893065693 | 9.97157599261154 | 0.999834118948884 | 184526 | 1 |
| B3 | 186280.372550211 | 187579.126540881 | 10.1160966387582 | 0.999861134540014 | 182517 | 1 |
| B4 | 211885.200124527 | 212416.459049545 | 10.5720278220846 | 0.999890405077804 | 209938 | 1 |
| B5 | 167895.847107994 | 169689.026962539 | 9.73775863769876 | 0.999810109884451 | 162519 | 1 |
| B6 | 198189.338070971 | 199220.965997468 | 10.3916857730234 | 0.999882522163175 | 195954 | 1 |
| C1 | 207489.893751353 | 208037.286532422 | 10.4787461728404 | 0.999896225564693 | 202754 | 1 |
| C2 | 216463.903421993 | 217943.891014691 | 10.4786726680676 | 0.999888625691143 | 214092 | 1 |
| C3 | 221932.523445026 | 222731.370864886 | 10.6369735109214 | 0.999890338389475 | 219781 | 1 |
| C4 | 207213.636732081 | 207630.462481275 | 10.219197904323 | 0.999880367165942 | 204181 | 1 |
| C5 | 210383.156206416 | 210910.465192021 | 10.5243461558896 | 0.999889771877566 | 208377 | 1 |
| C6 | 220135.56193467 | 221209.377305463 | 10.5607272186908 | 0.999881922546549 | 217390 | 1 |
显示注释
(1)ACE:估计群落中功能数目。
(2)chao1:估计群落样品中包含的功能总数,群落中低丰度功能越多,chao1指数越大。
(3)shannon:样品中的分类总数及其占比。群落多样性越高,功能分布越均匀,shannon指数越大。
(4)simpson:表征群落内功能分布的多样性和均匀度,功能均匀度越好,Simpson指数越大。
(5)observed_species:直观观测到的功能数目,指数越大观测到的功能越多。
(6)goods_coverage:覆盖度,测序覆盖度越高,指数越大。
结果目录:
eggNOG Alpha多样性指数汇总表见:result/05.Diversity/eggNOG/AlphaDiversity/Alpha_index_table/eggNOG_Alphaindex.*.xls
4.5.1.4 CAZy
从功能注释 Absolute 表出发,计算得到每个样本内物种的丰富度以及多样性。该表仅展示 level2 层级结果,其他层级见结果文件。
表4.7 Alpha多样性分析统计结果
| Sample | ACE | chao1 | shannon | simpson | observed_species | goods_coverage |
|---|---|---|---|---|---|---|
| A1 | 315.312536254797 | 314.6 | 4.22572314998574 | 0.964337753356556 | 314 | 1 |
| A2 | 318.214953271028 | 318 | 4.2356347474775 | 0.96510466630556 | 318 | 1 |
| A3 | 326.336526077098 | 326 | 4.21433113268013 | 0.964255266260201 | 326 | 1 |
| A4 | 304.545796308954 | 304.125 | 4.21598324541129 | 0.965094789977119 | 304 | 1 |
| A5 | 323.943910989179 | 323.5 | 4.25715557512568 | 0.96563683295147 | 322 | 1 |
| A6 | 328.075435519603 | 327.6 | 4.21043990155504 | 0.963683989142133 | 327 | 1 |
| B1 | 331.376680391569 | 330.5 | 4.1759323901759 | 0.961842108822012 | 330 | 1 |
| B2 | 312.680309442895 | 313 | 3.92760303601137 | 0.956142341984569 | 310 | 1 |
| B3 | 316.413154031694 | 315.5 | 4.02230554411756 | 0.95913785458032 | 314 | 1 |
| B4 | 324.754409165509 | 325.5 | 4.08575025422213 | 0.959941700907112 | 324 | 1 |
| B5 | 301.304899930616 | 302.090909090909 | 3.93406985825386 | 0.957900540309229 | 295 | 1 |
| B6 | 314.568775686428 | 314.2 | 3.99236952338246 | 0.957977119677532 | 314 | 1 |
| C1 | 345.295207490014 | 345.142857142857 | 4.04328177272977 | 0.957837136968416 | 343 | 1 |
| C2 | 341.827769557028 | 342 | 4.07812283334349 | 0.95964309287744 | 341 | 1 |
| C3 | 355.328571128285 | 355.75 | 4.10944296584069 | 0.957804510804491 | 352 | 1 |
| C4 | 343.423950089238 | 342.428571428571 | 4.08995668833175 | 0.963238807332684 | 341 | 1 |
| C5 | 341.181461030023 | 341 | 4.06111769030604 | 0.956837981854216 | 341 | 1 |
| C6 | 353.499705739011 | 352.75 | 4.06764003512731 | 0.956146606838414 | 352 | 1 |
显示注释
(1)ACE:估计群落中功能数目。
(2)chao1:估计群落样品中包含的功能总数,群落中低丰度功能越多,chao1指数越大。
(3)shannon:样品中的分类总数及其占比。群落多样性越高,功能分布越均匀,shannon指数越大。
(4)simpson:表征群落内功能分布的多样性和均匀度,功能均匀度越好,Simpson指数越大。
(5)observed_species:直观观测到的功能数目,指数越大观测到的功能越多。
(6)goods_coverage:覆盖度,测序覆盖度越高,指数越大。
结果目录:
CAZy Alpha多样性指数汇总表见:result/05.Diversity/CAZy/AlphaDiversity/Alpha_index_table/CAZy_Alphaindex.*.xls
4.5.1.5 VFDB
从功能注释 Absolute 表出发,计算得到每个样本内物种的丰富度以及多样性。该表仅展示 level2 层级结果,其他层级见结果文件。
表4.8 Alpha多样性分析统计结果
| Sample | ACE | chao1 | shannon | simpson | observed_species | goods_coverage |
|---|---|---|---|---|---|---|
| A1 | 388.964525555637 | 388.75 | 4.60681106128574 | 0.98470464011846 | 385 | 1 |
| A2 | 397.24041730913 | 397 | 4.72389561029957 | 0.98644835192534 | 397 | 1 |
| A3 | 403.03142081448 | 403.5 | 4.75642218434215 | 0.986779414541669 | 401 | 1 |
| A4 | 359.65264472372 | 358.105263157895 | 4.50371181939169 | 0.983493528006494 | 357 | 1 |
| A5 | 400.070456412606 | 399.666666666667 | 4.66003241837505 | 0.985297833356714 | 399 | 1 |
| A6 | 428.721668468319 | 427.4 | 4.79496079870919 | 0.987081171202975 | 425 | 1 |
| B1 | 477.46007927133 | 479.111111111111 | 4.8583725112885 | 0.986956227260229 | 473 | 1 |
| B2 | 453.105265940573 | 452.769230769231 | 4.65794775277879 | 0.982700147697841 | 450 | 1 |
| B3 | 439.798688328006 | 439.666666666667 | 4.72728292467256 | 0.984065139323195 | 438 | 1 |
| B4 | 474.228619058982 | 473.75 | 4.90421579051127 | 0.986858924579309 | 473 | 1 |
| B5 | 422.860236418291 | 425.1 | 4.55318367644758 | 0.980740144991998 | 416 | 1 |
| B6 | 462.366228159147 | 464.333333333333 | 4.87039302374642 | 0.986420684981125 | 461 | 1 |
| C1 | 422.015365013354 | 426.375 | 4.7970003134788 | 0.98630623148142 | 415 | 1 |
| C2 | 442.005573189256 | 442.142857142857 | 4.8146976230964 | 0.985876990923083 | 440 | 1 |
| C3 | 438.405496200752 | 442.2 | 4.80690610314774 | 0.986319274165337 | 435 | 1 |
| C4 | 420.532976209875 | 421 | 4.71480188994615 | 0.983807665962192 | 418 | 1 |
| C5 | 415.950879765396 | 419 | 4.8090694266594 | 0.986351806659103 | 414 | 1 |
| C6 | 440.323104610956 | 443.75 | 4.78512278327314 | 0.986134841169294 | 434 | 1 |
显示注释
(1)ACE:估计群落中功能数目。
(2)chao1:估计群落样品中包含的功能总数,群落中低丰度功能越多,chao1指数越大。
(3)shannon:样品中的分类总数及其占比。群落多样性越高,功能分布越均匀,shannon指数越大。
(4)simpson:表征群落内功能分布的多样性和均匀度,功能均匀度越好,Simpson指数越大。
(5)observed_species:直观观测到的功能数目,指数越大观测到的功能越多。
(6)goods_coverage:覆盖度,测序覆盖度越高,指数越大。
结果目录:
VFDB Alpha多样性指数汇总表见:result/05.Diversity/VFDB/AlphaDiversity/Alpha_index_table/VFDB_Alphaindex.*.xls
4.5.1.6 PHI
从功能注释 Absolute 表出发,计算得到每个样本内物种的丰富度以及多样性。该表仅展示 level1 层级结果,其他层级见结果文件。
表4.9 Alpha多样性分析统计结果
| Sample | ACE | chao1 | shannon | simpson | observed_species | goods_coverage |
|---|---|---|---|---|---|---|
| A1 | 201 | 201 | 4.01759895340563 | 0.968251945852084 | 201 | 1 |
| A2 | 213.217614301625 | 212.75 | 4.0455743772716 | 0.969478833047895 | 212 | 1 |
| A3 | 210 | 210 | 4.06607335419074 | 0.96984182576026 | 210 | 1 |
| A4 | 200.206607729125 | 199.75 | 3.9989296105317 | 0.968881328985398 | 199 | 1 |
| A5 | 212 | 212 | 4.03725953724112 | 0.96873147064498 | 212 | 1 |
| A6 | 214 | 214 | 4.08325482974162 | 0.970873227472076 | 214 | 1 |
| B1 | 217.570881226054 | 216 | 4.08974464276702 | 0.969045577602434 | 215 | 1 |
| B2 | 219.744663846709 | 218.5 | 3.97011291985018 | 0.967050525570614 | 217 | 1 |
| B3 | 220.409962871287 | 222 | 3.96627329603286 | 0.965778952140164 | 217 | 1 |
| B4 | 225.79859541436 | 225.25 | 4.06400505287462 | 0.967360764709619 | 225 | 1 |
| B5 | 204.641517318228 | 205 | 3.91842811777064 | 0.964926787797584 | 204 | 1 |
| B6 | 219.624441848297 | 219.333333333333 | 3.99160103073352 | 0.964622868561648 | 219 | 1 |
| C1 | 236.365472580093 | 235.25 | 4.04084851612471 | 0.967825529822778 | 234 | 1 |
| C2 | 241.801979530165 | 242.5 | 4.03696408799607 | 0.967121068825541 | 241 | 1 |
| C3 | 241.406060606061 | 240.25 | 4.12384770413627 | 0.970215262684218 | 240 | 1 |
| C4 | 238.327971359609 | 238 | 3.94969923613933 | 0.962657889600896 | 238 | 1 |
| C5 | 238.340425531915 | 239 | 4.07136359697583 | 0.968899322601918 | 238 | 1 |
| C6 | 244.066582407443 | 246 | 4.11760993118336 | 0.970468601671707 | 243 | 1 |
显示注释
(1)ACE:估计群落中功能数目。
(2)chao1:估计群落样品中包含的功能总数,群落中低丰度功能越多,chao1指数越大。
(3)shannon:样品中的分类总数及其占比。群落多样性越高,功能分布越均匀,shannon指数越大。
(4)simpson:表征群落内功能分布的多样性和均匀度,功能均匀度越好,Simpson指数越大。
(5)observed_species:直观观测到的功能数目,指数越大观测到的功能越多。
(6)goods_coverage:覆盖度,测序覆盖度越高,指数越大。
结果目录:
PHI Alpha多样性指数汇总表见:result/05.Diversity/PHI/AlphaDiversity/Alpha_index_table/PHI_Alphaindex.*.xls
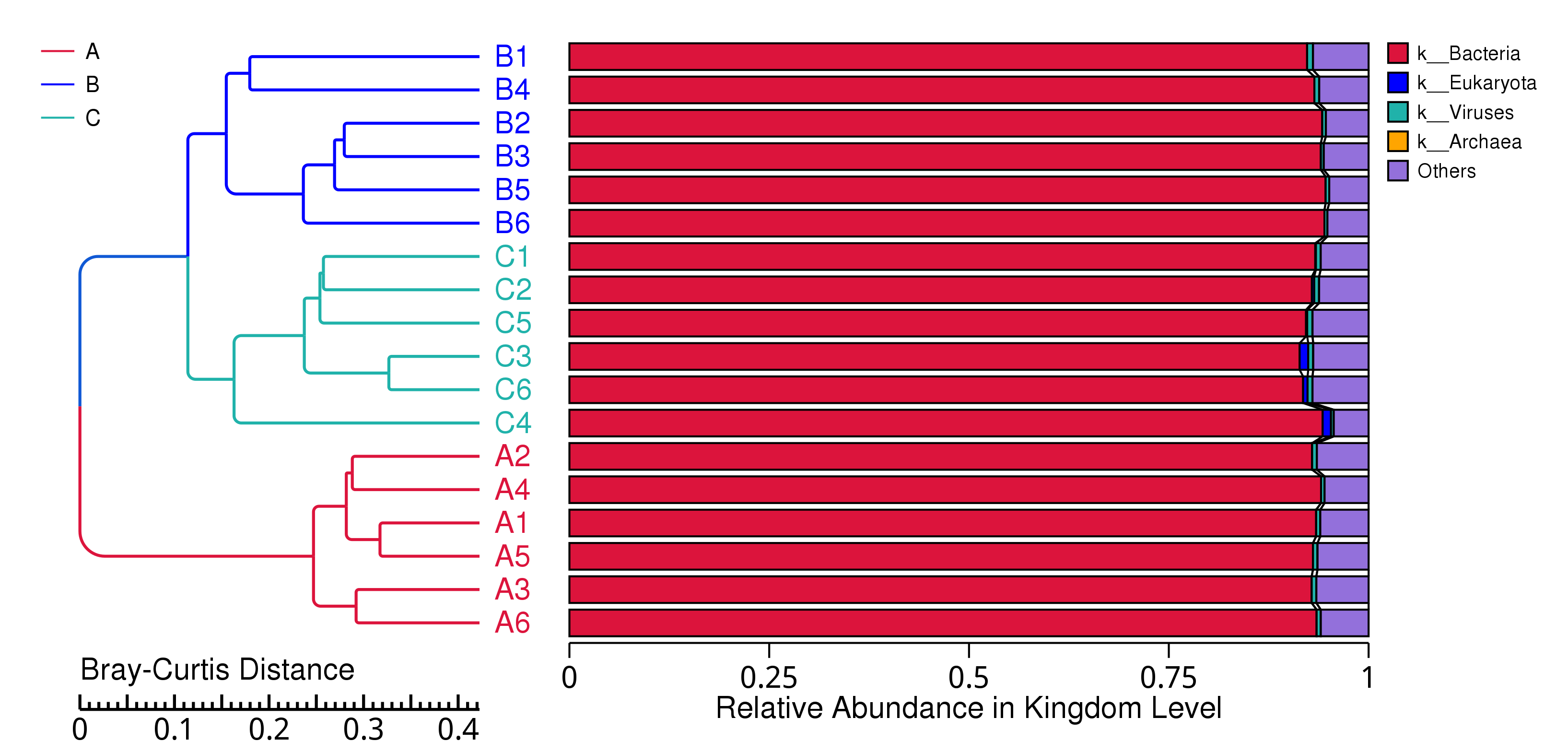
4.5.2 基于相对丰度的样品聚类分析
为了研究不同样品的相似性,还可以通过对样品进行聚类分析,构建样品的聚类树。Bray-Curtis 距离是系统聚类法中使用最普遍的一个距离指标,它主要用来刻画样品间的相近程度,它的大小是进行样品分类的主要依据。
从基因在各样品中的丰度表出发,以 Bray-Curtis 距离矩阵进行样品间聚类分析,并将聚类结果与各样品在物种或功能层级上的相对丰度整合进行展示。
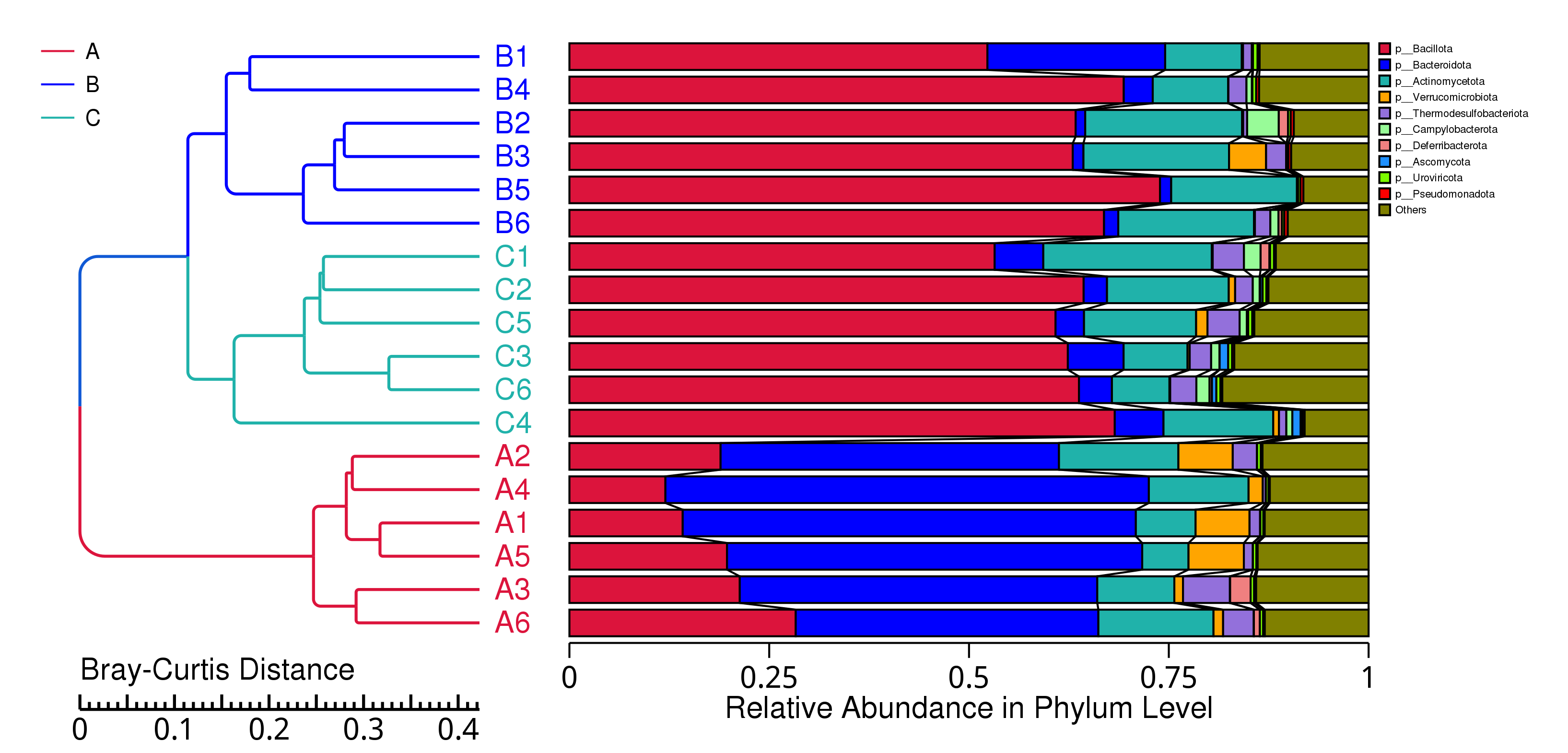
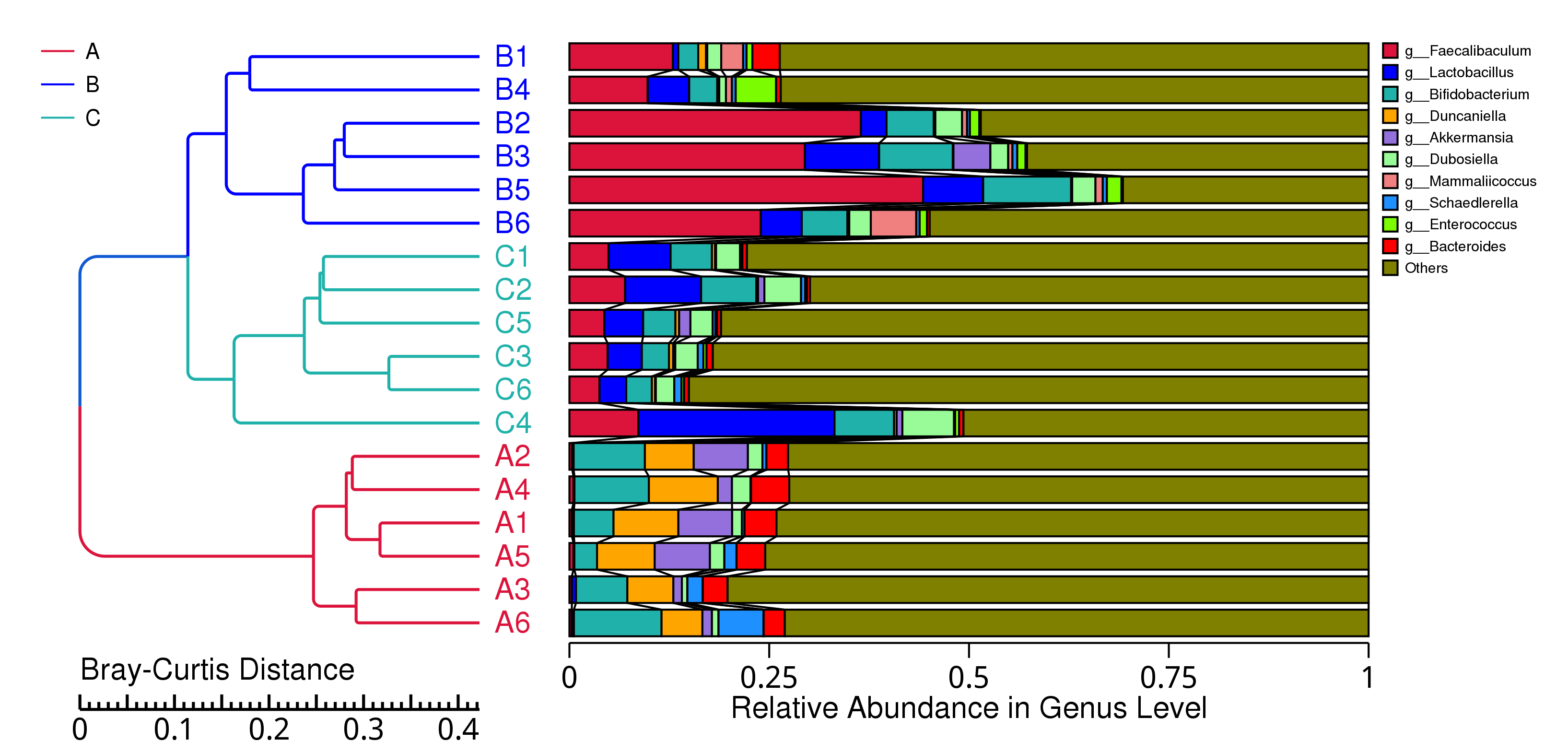
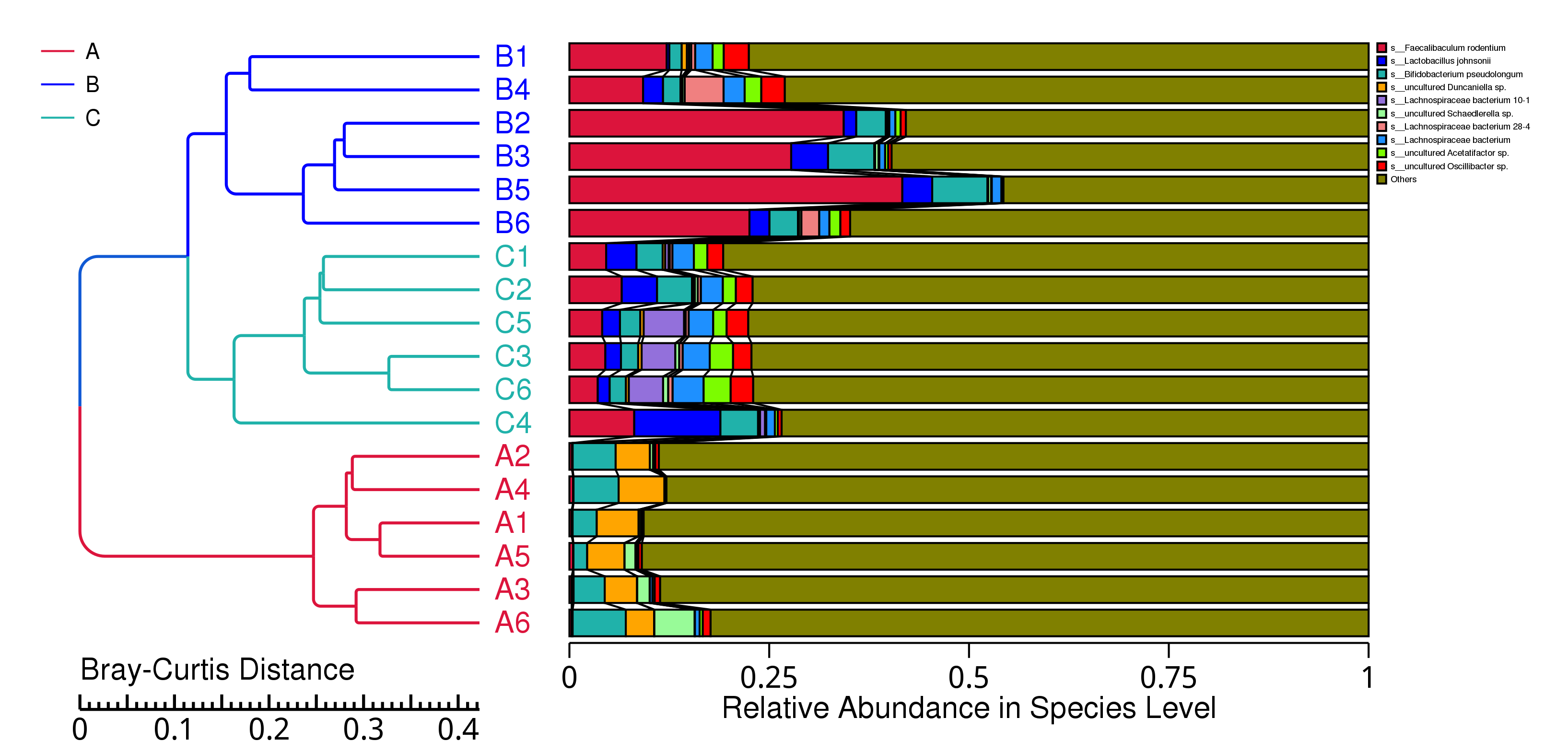
4.5.2.1 Micro_NR
图4.22 基于 Bray-Curtis 距离的Micro_NR聚类树
说明:图中,左侧是 Bray-Curtis 距离聚类树结构;右侧是各样品的物种相对丰度分布图
结果目录:
基于 Bray-Curtis 距离的聚类树见 : result/05.Diversity/MicroNR/ClusterTree_group1
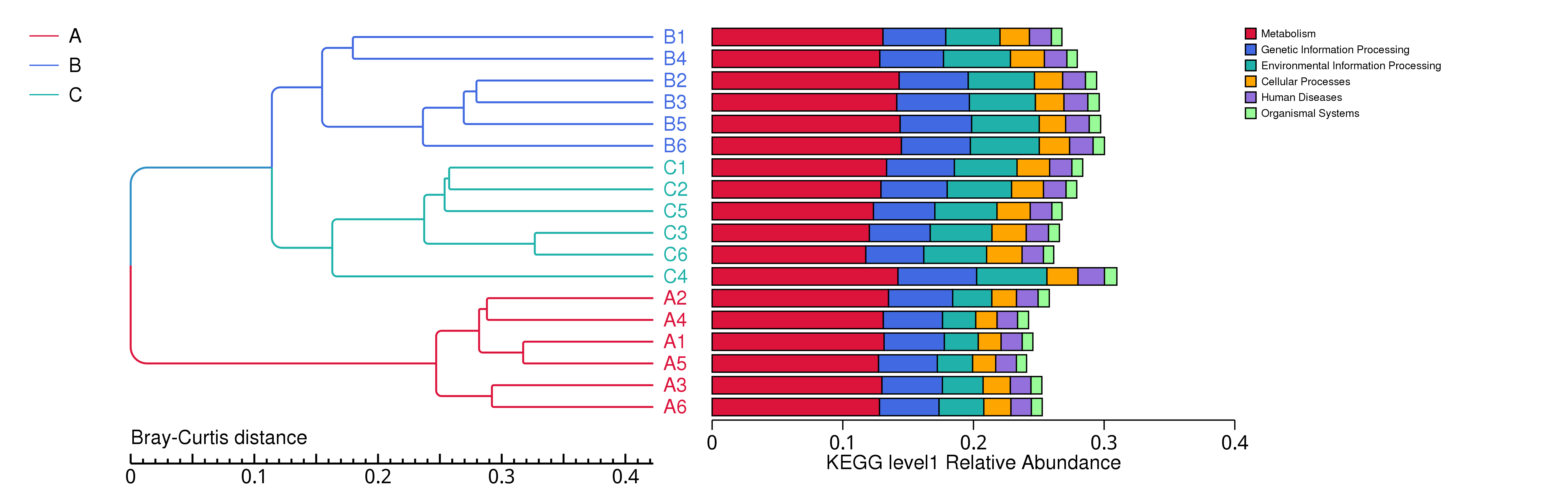
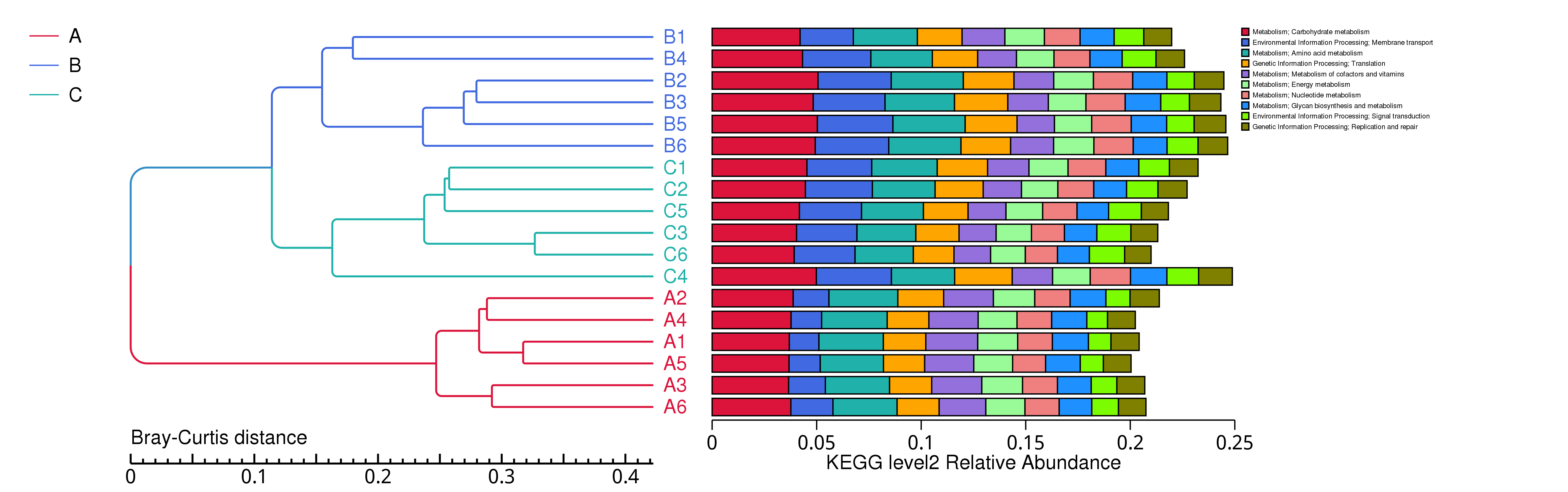
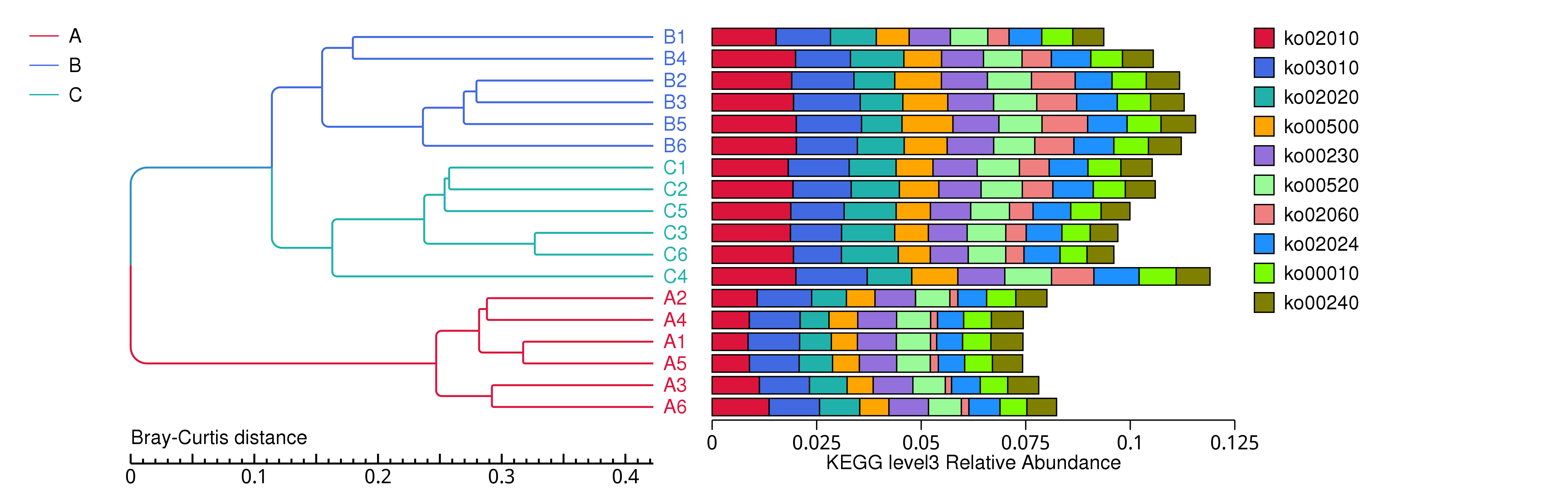
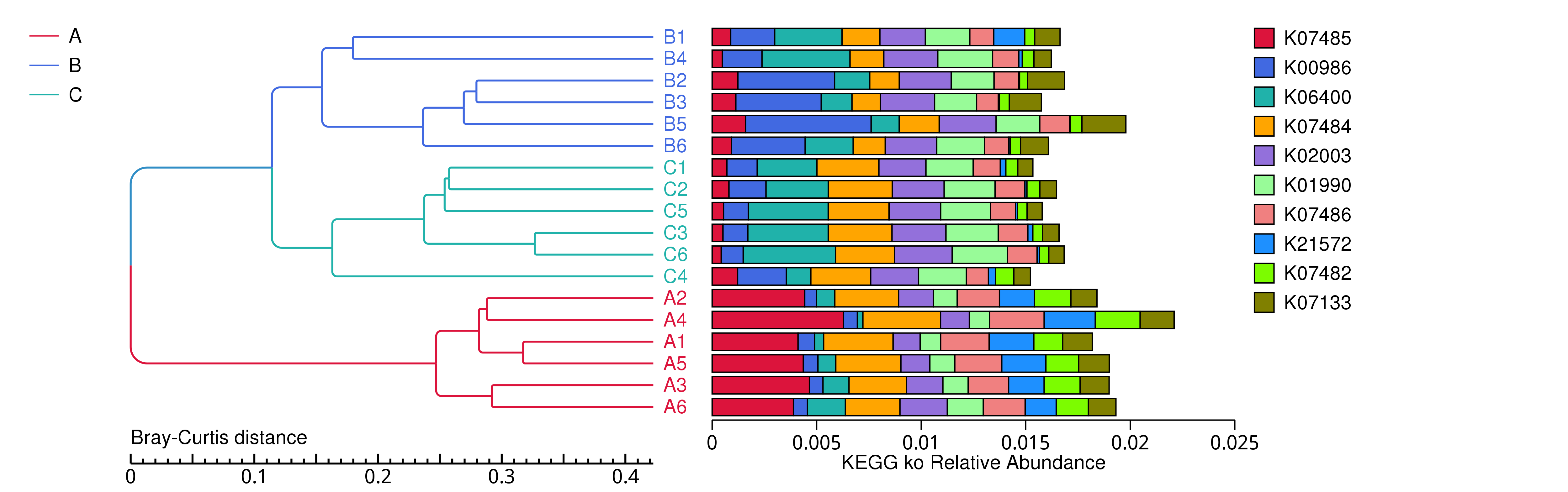
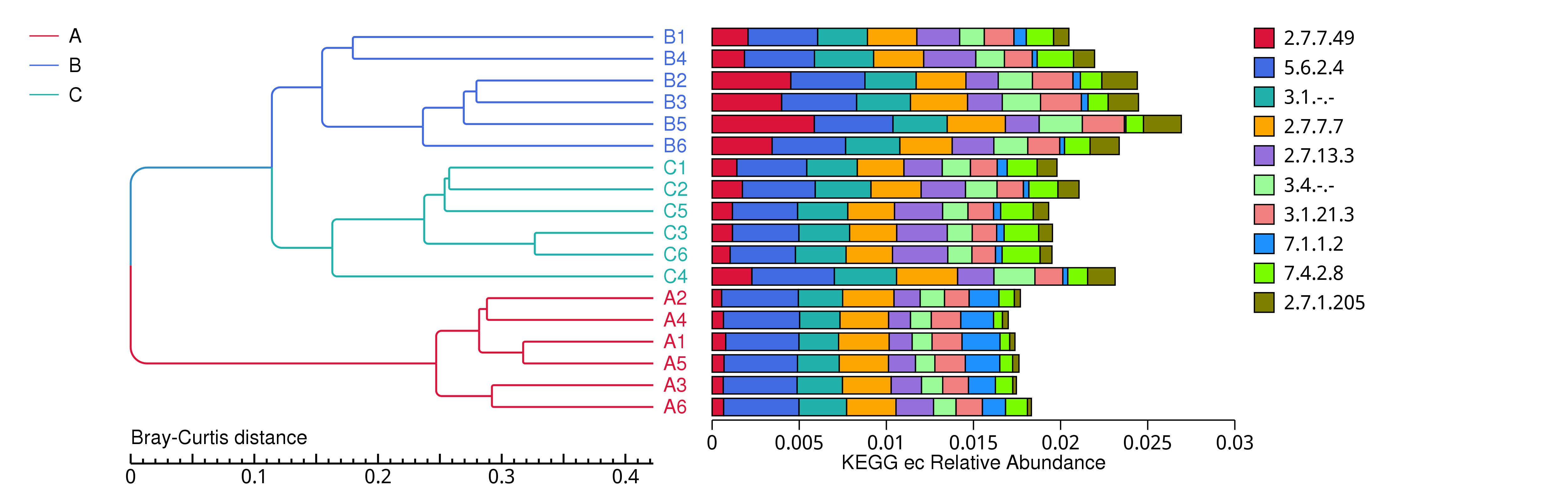
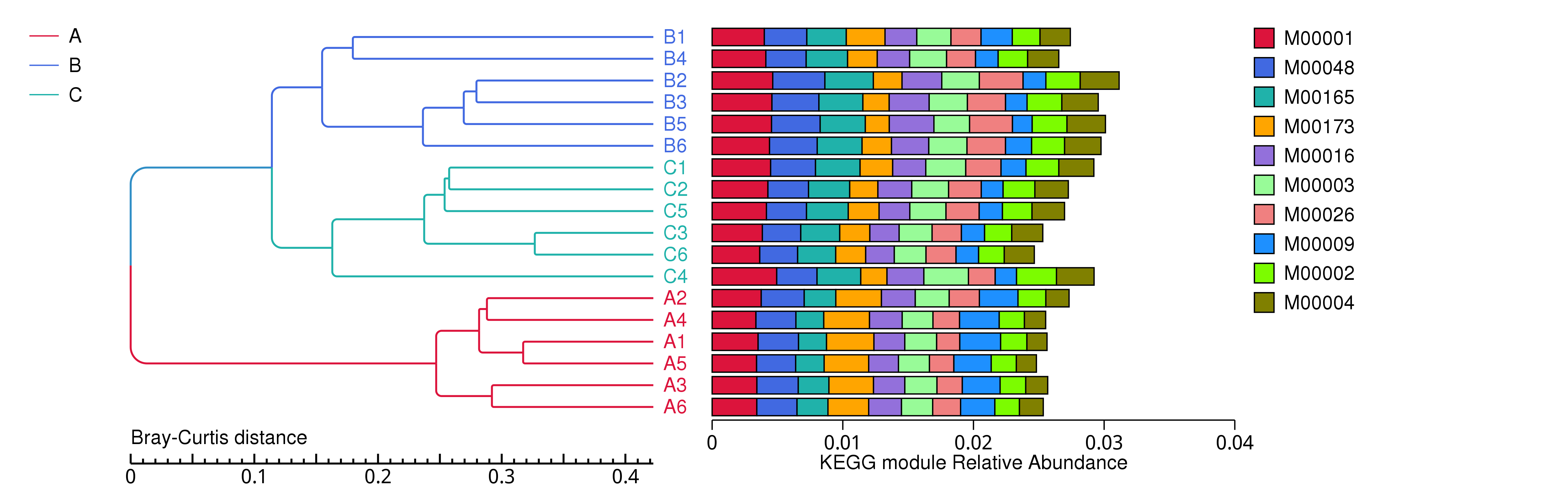
4.5.2.2 KEGG
图4.23 基于 Bray-Curtis 距离的KEGG聚类树
说明:图中,左侧是 Bray-Curtis 距离聚类树结构;右侧是各样品的功能相对丰度分布图
结果目录:
基于 Bray-Curtis 距离的聚类树见 : result/05.Diversity/KEGG/ClusterTree_group1
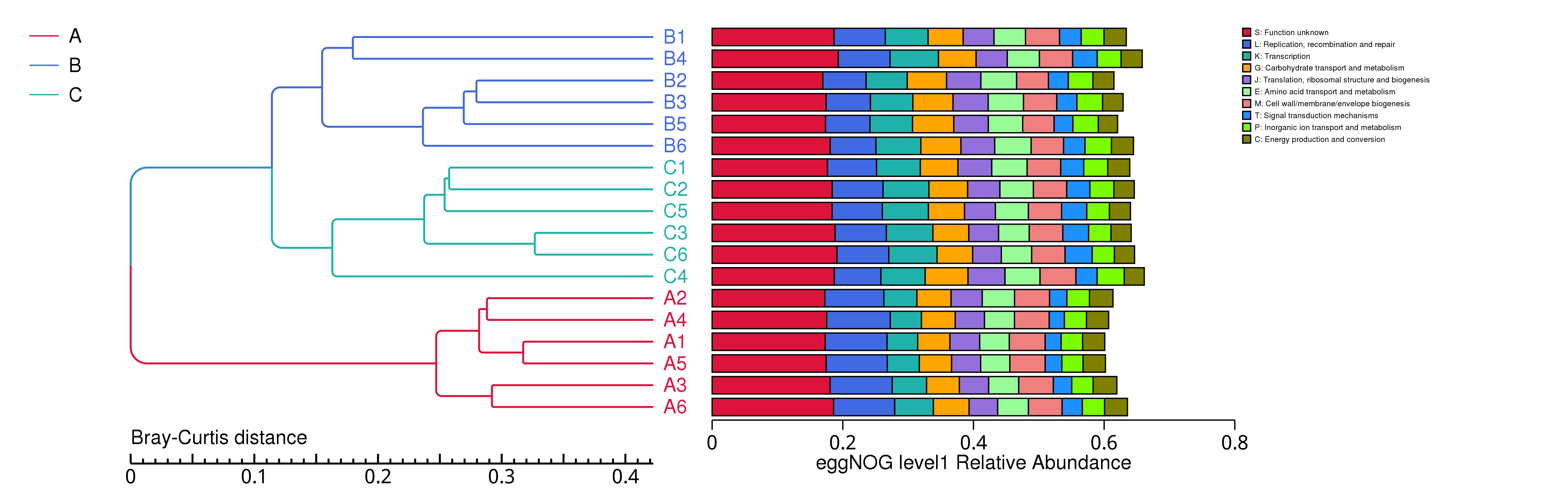
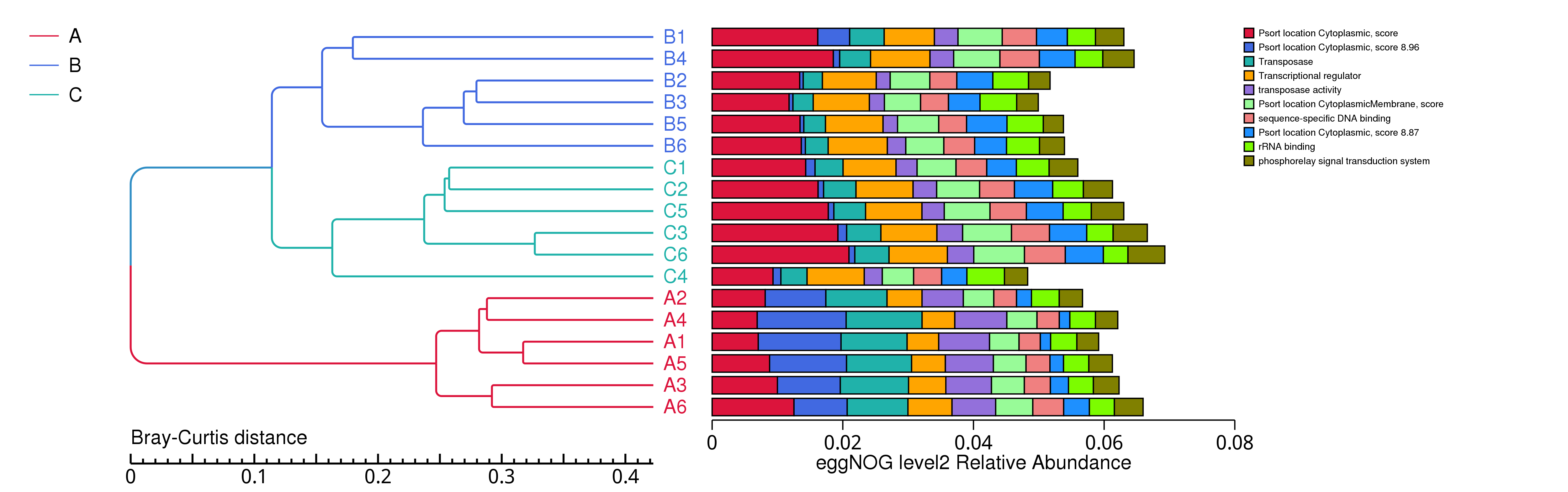
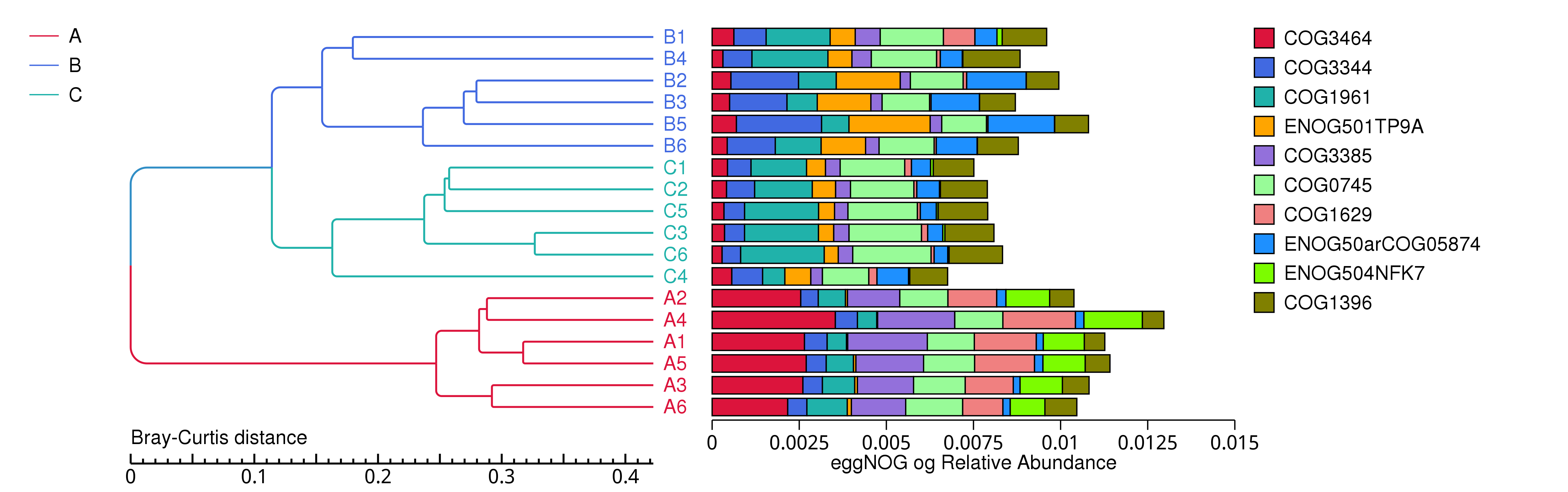
4.5.2.3 eggNOG
图4.24 基于 Bray-Curtis 距离的eggNOG聚类树
说明:图中,左侧是 Bray-Curtis 距离聚类树结构;右侧是各样品的功能相对丰度分布图
结果目录:
基于 Bray-Curtis 距离的聚类树见 : result/05.Diversity/eggNOG/ClusterTree_group1
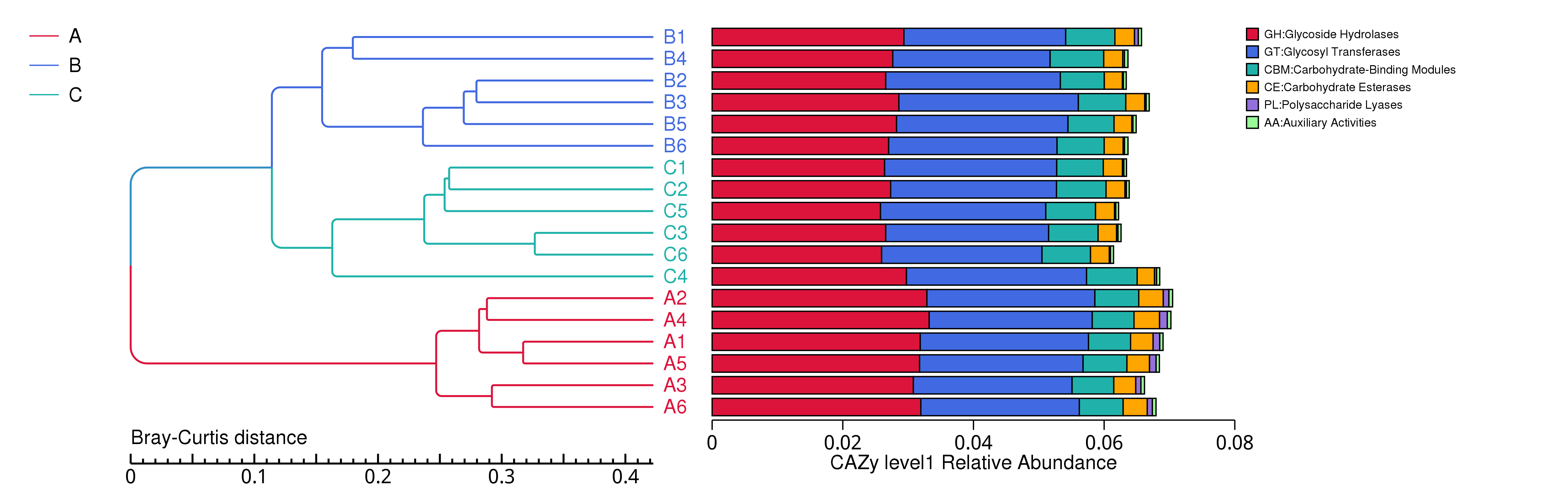
4.5.2.4 CAZy
图4.25 基于 Bray-Curtis 距离的CAZy聚类树
说明:图中,左侧是 Bray-Curtis 距离聚类树结构;右侧是各样品的功能相对丰度分布图
结果目录:
基于 Bray-Curtis 距离的聚类树见 : result/05.Diversity/CAZy/ClusterTree_group1
4.5.2.5 VFDB
图4.26 基于 Bray-Curtis 距离的VFDB聚类树
说明:图中,左侧是 Bray-Curtis 距离聚类树结构;右侧是各样品的功能相对丰度分布图
结果目录:
基于 Bray-Curtis 距离的聚类树见 : result/05.Diversity/VFDB/ClusterTree_group1
4.5.2.6 PHI
图4.27 基于 Bray-Curtis 距离的PHI聚类树
说明:图中,左侧是 Bray-Curtis 距离聚类树结构;右侧是各样品的功能相对丰度分布图
结果目录:
基于 Bray-Curtis 距离的聚类树见 : result/05.Diversity/PHI/ClusterTree_group1
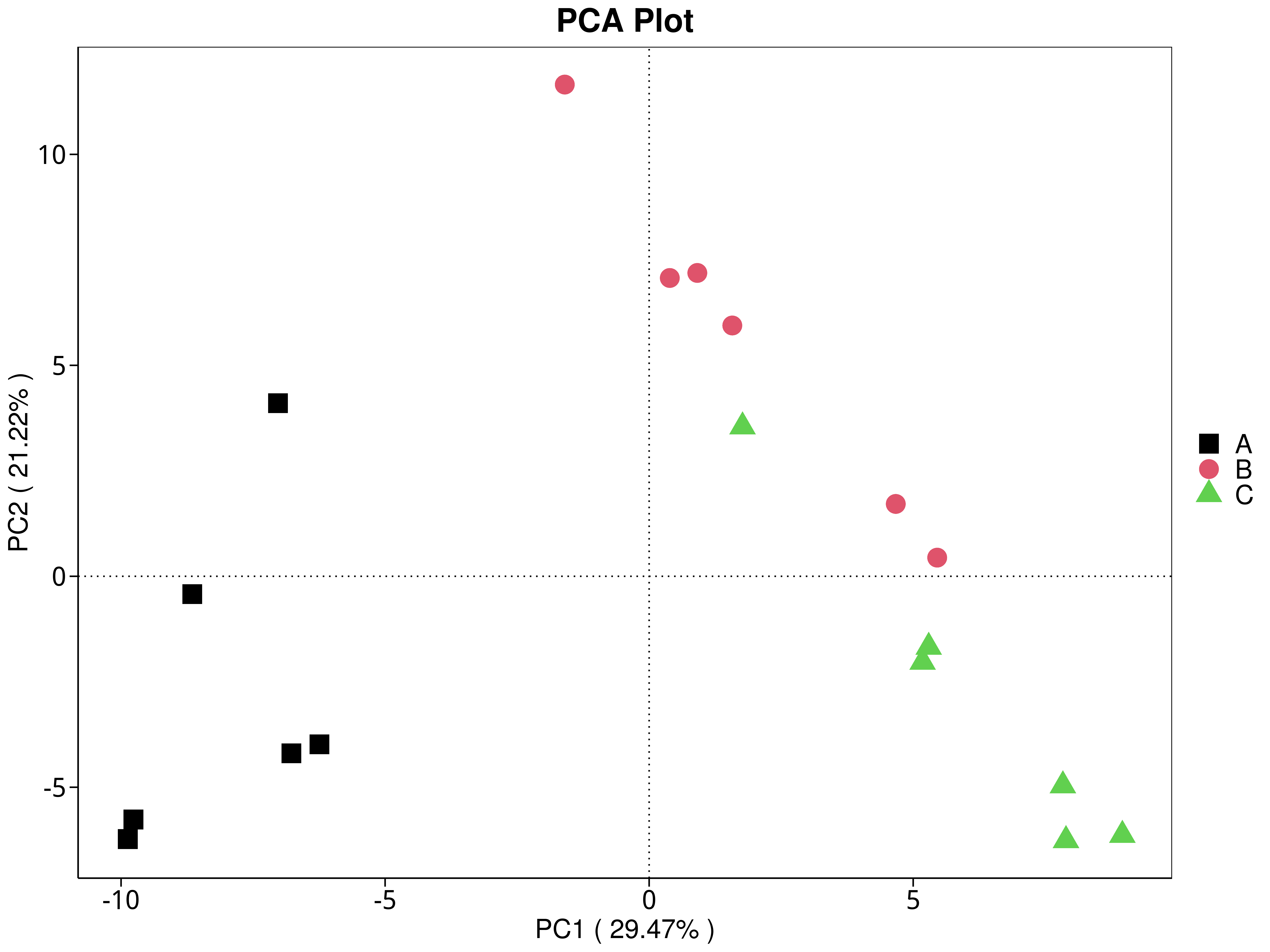
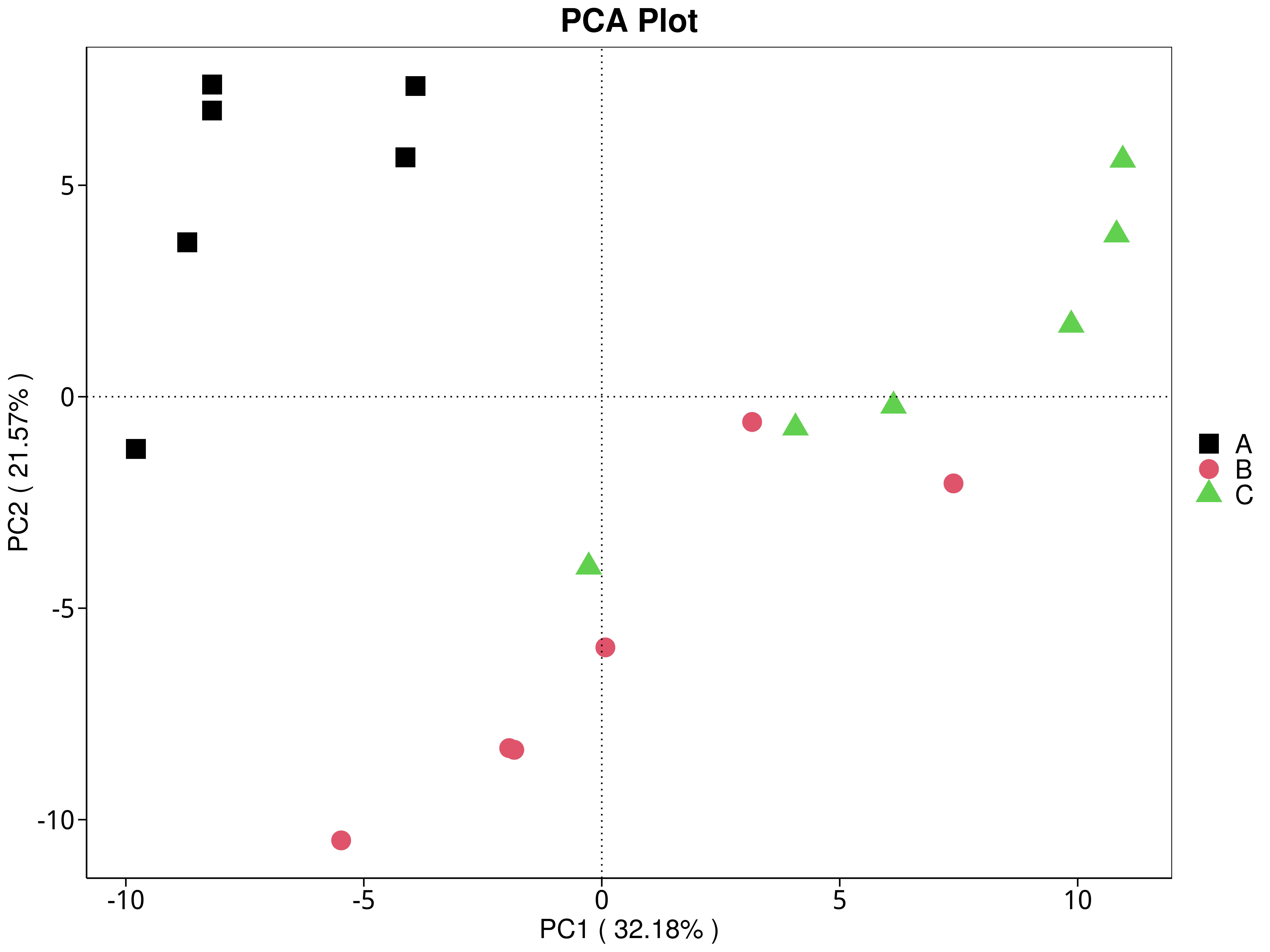
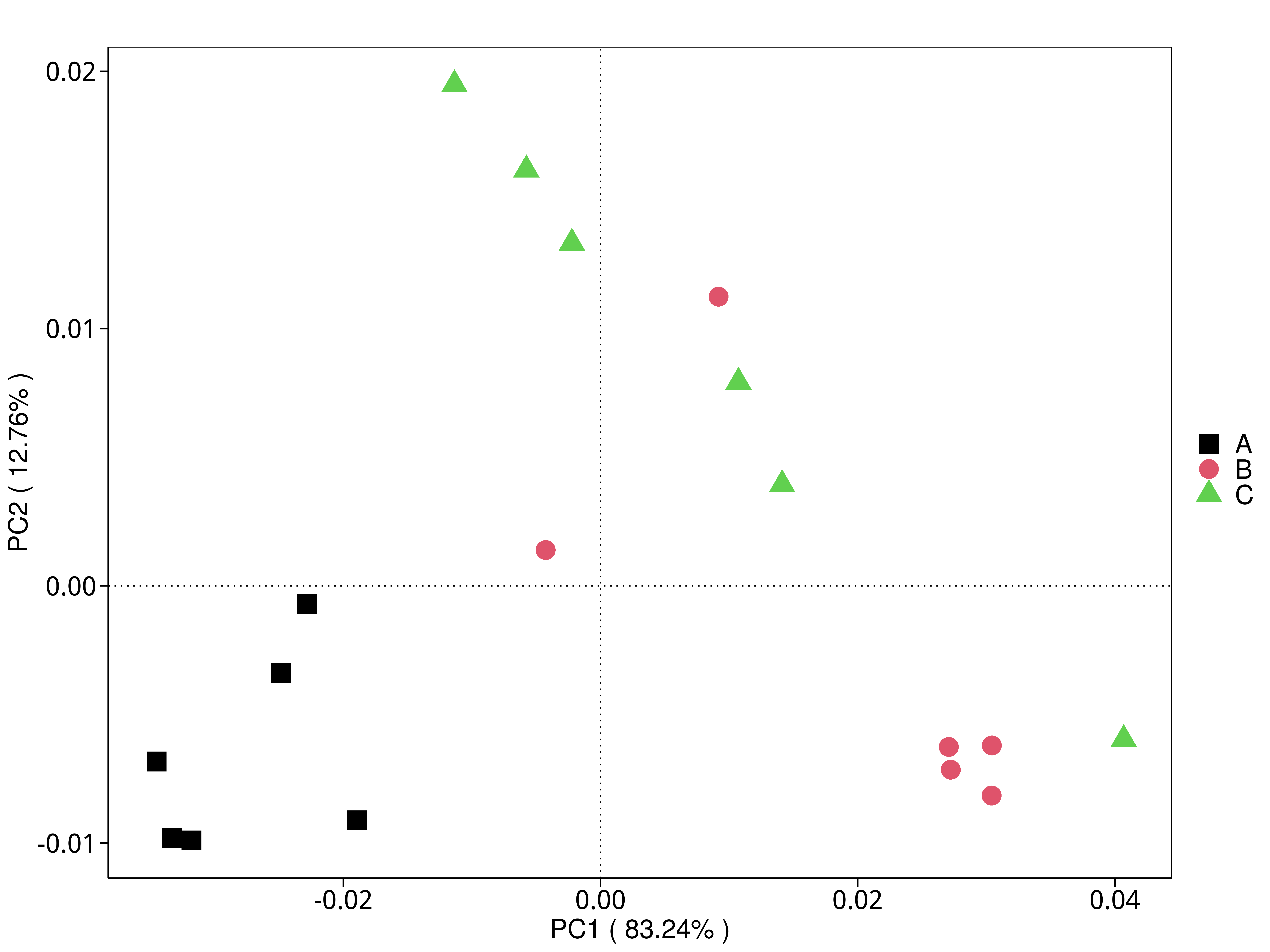
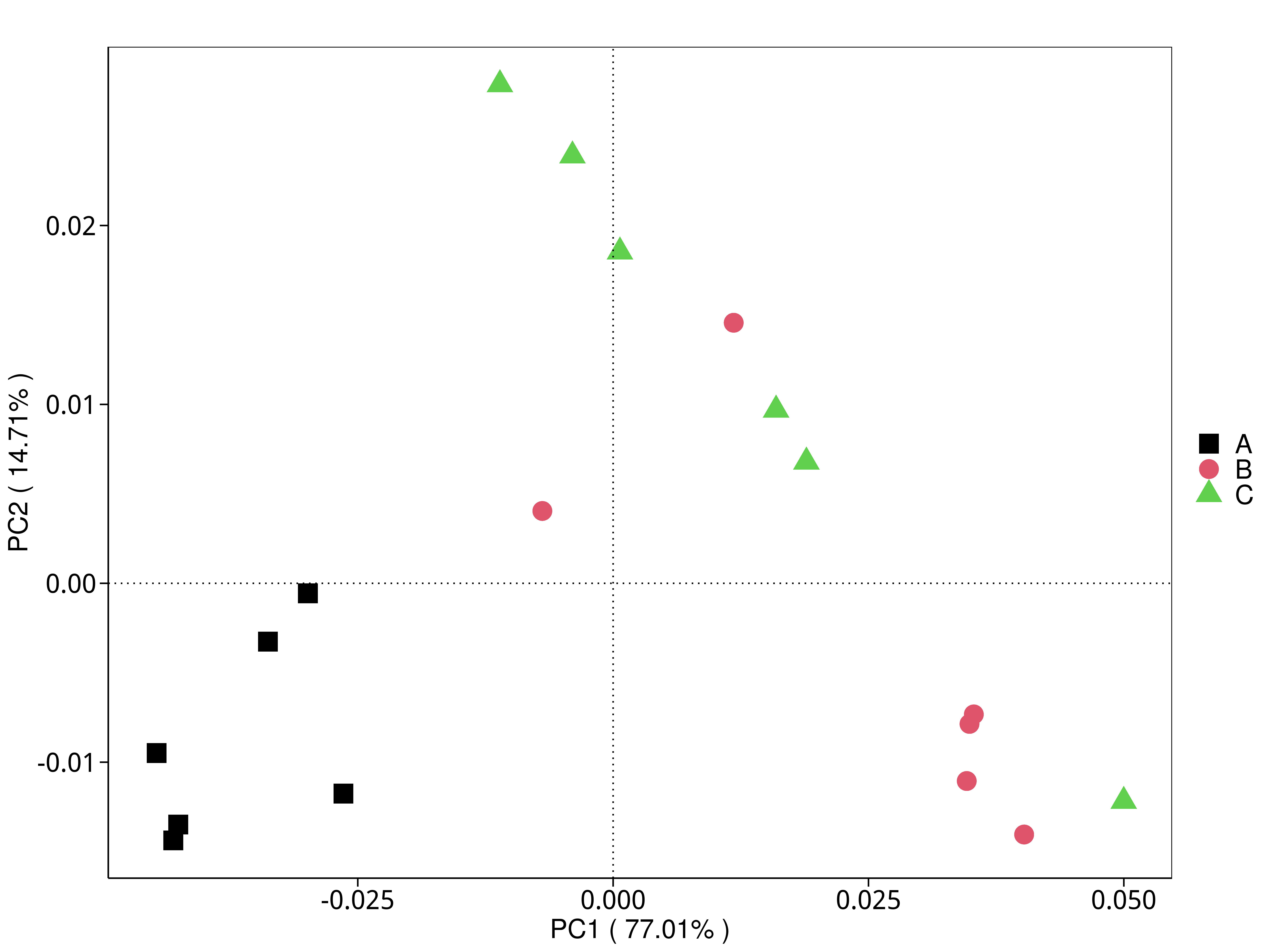
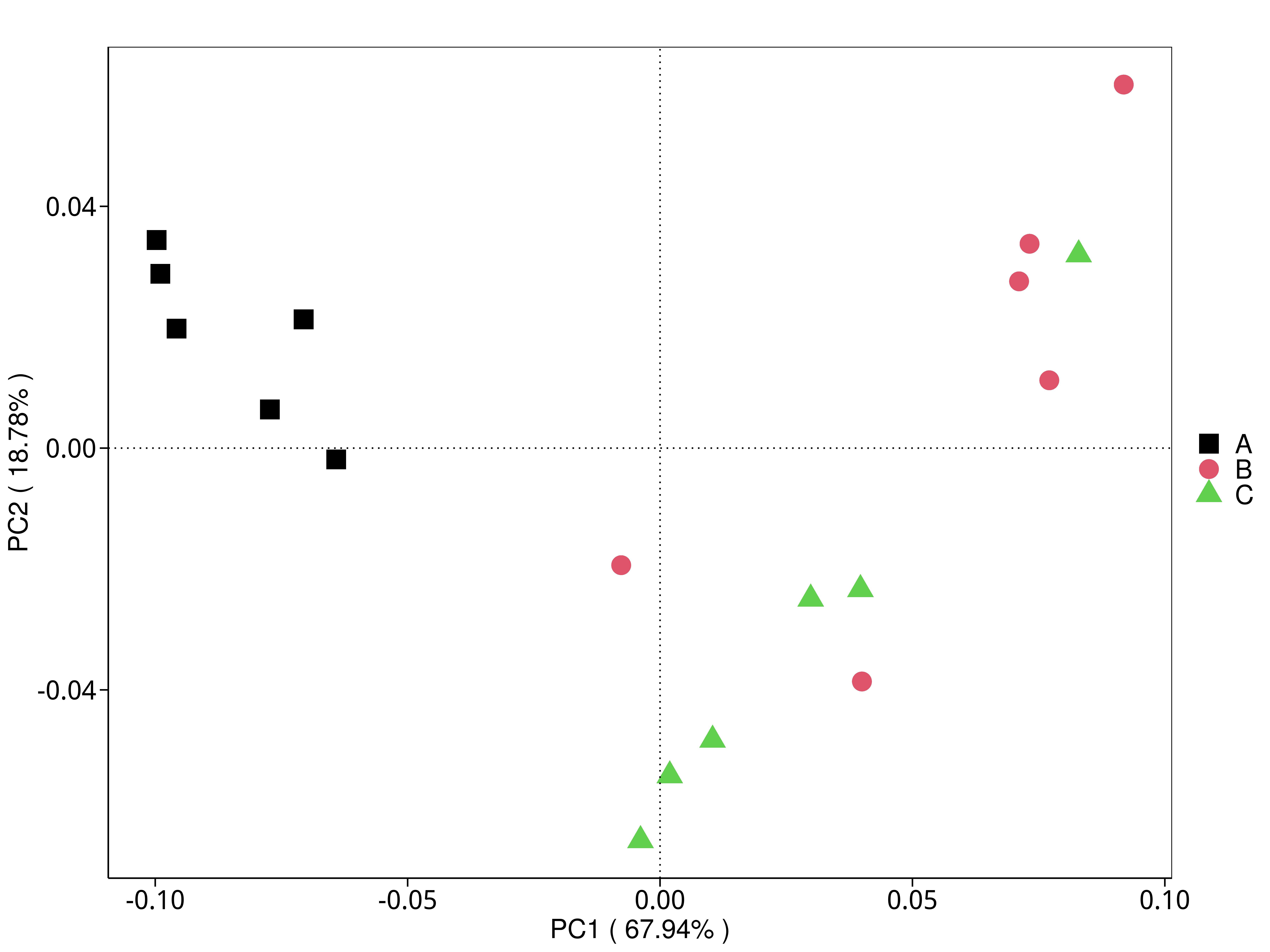
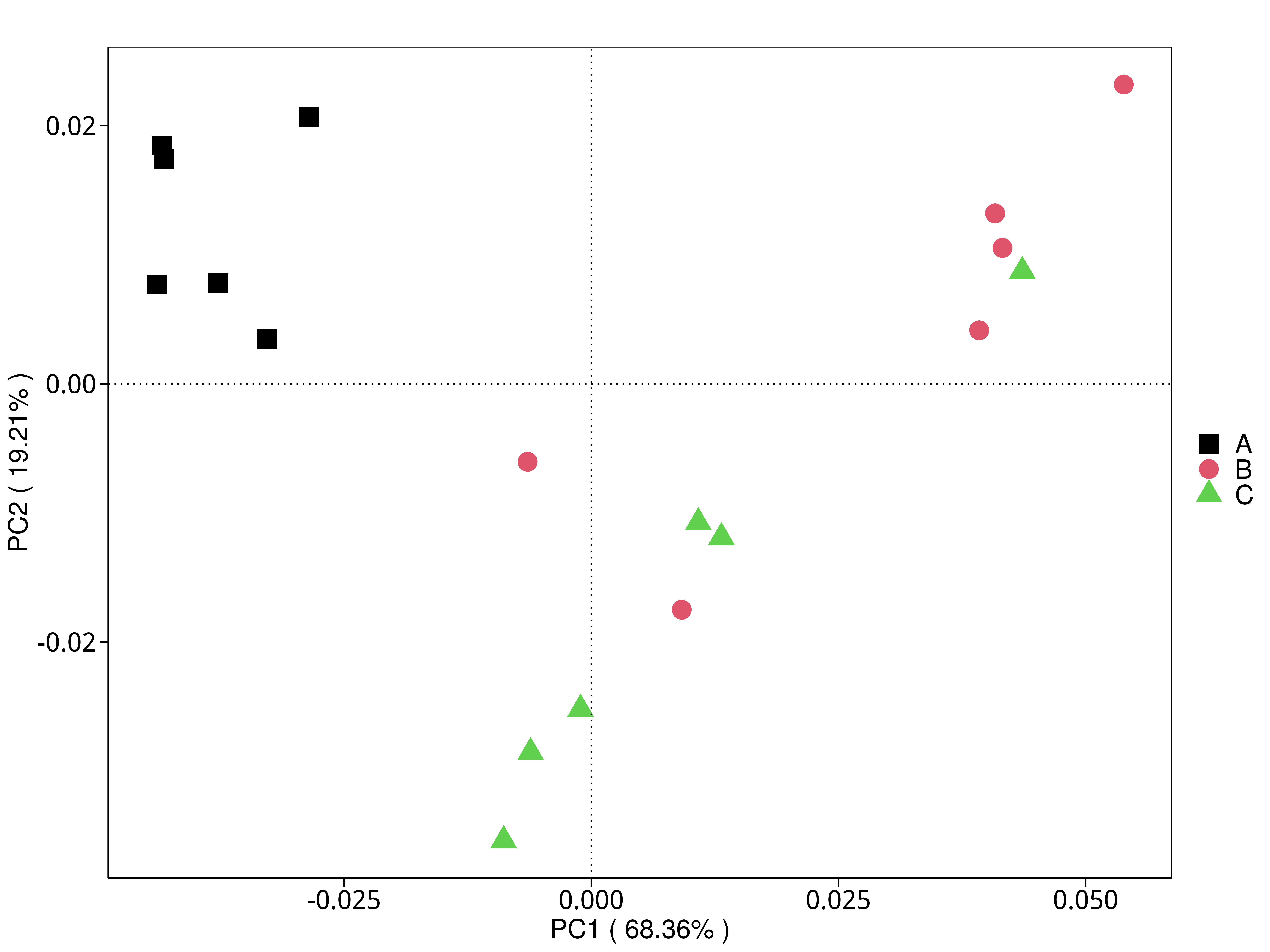
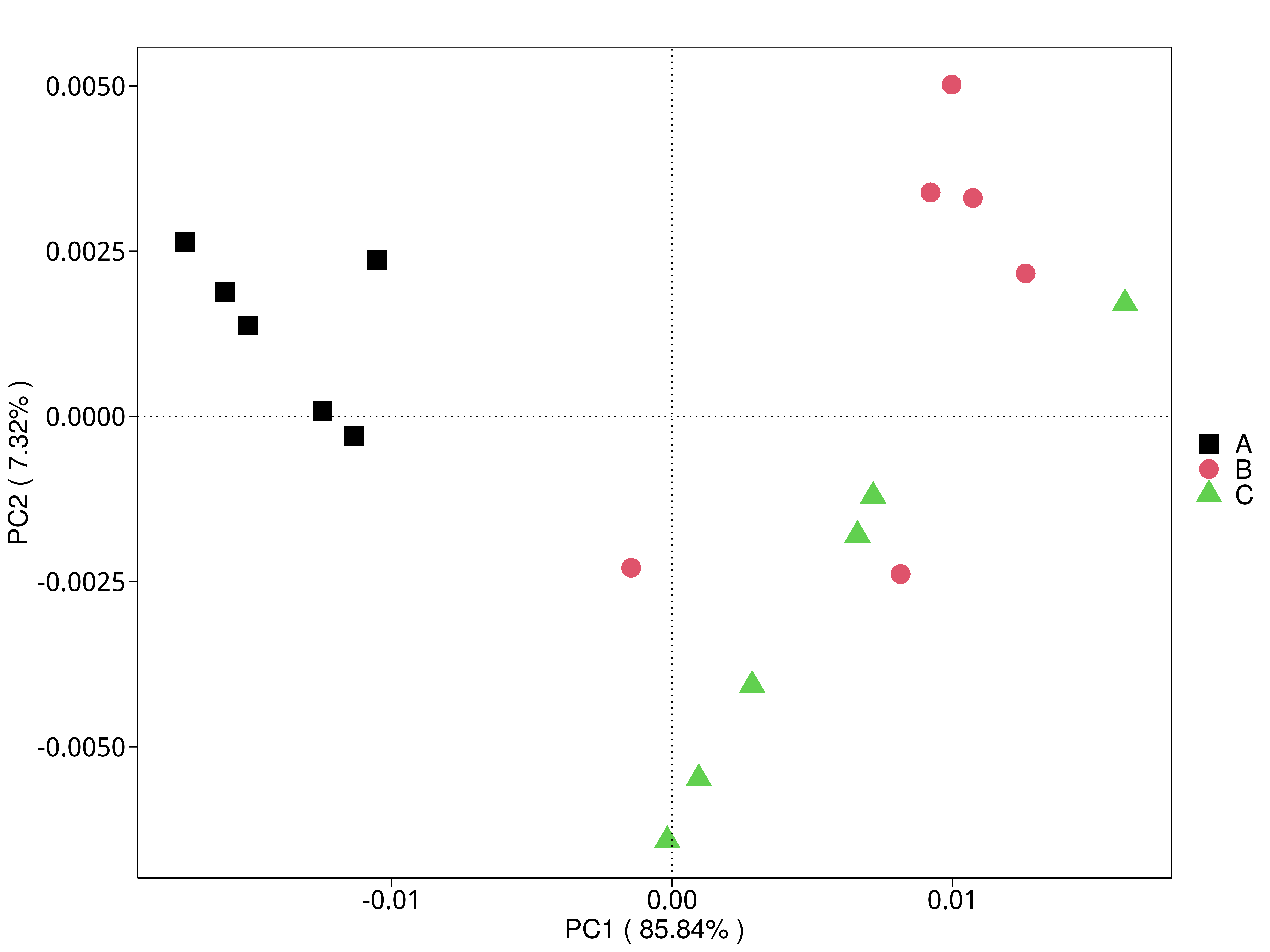
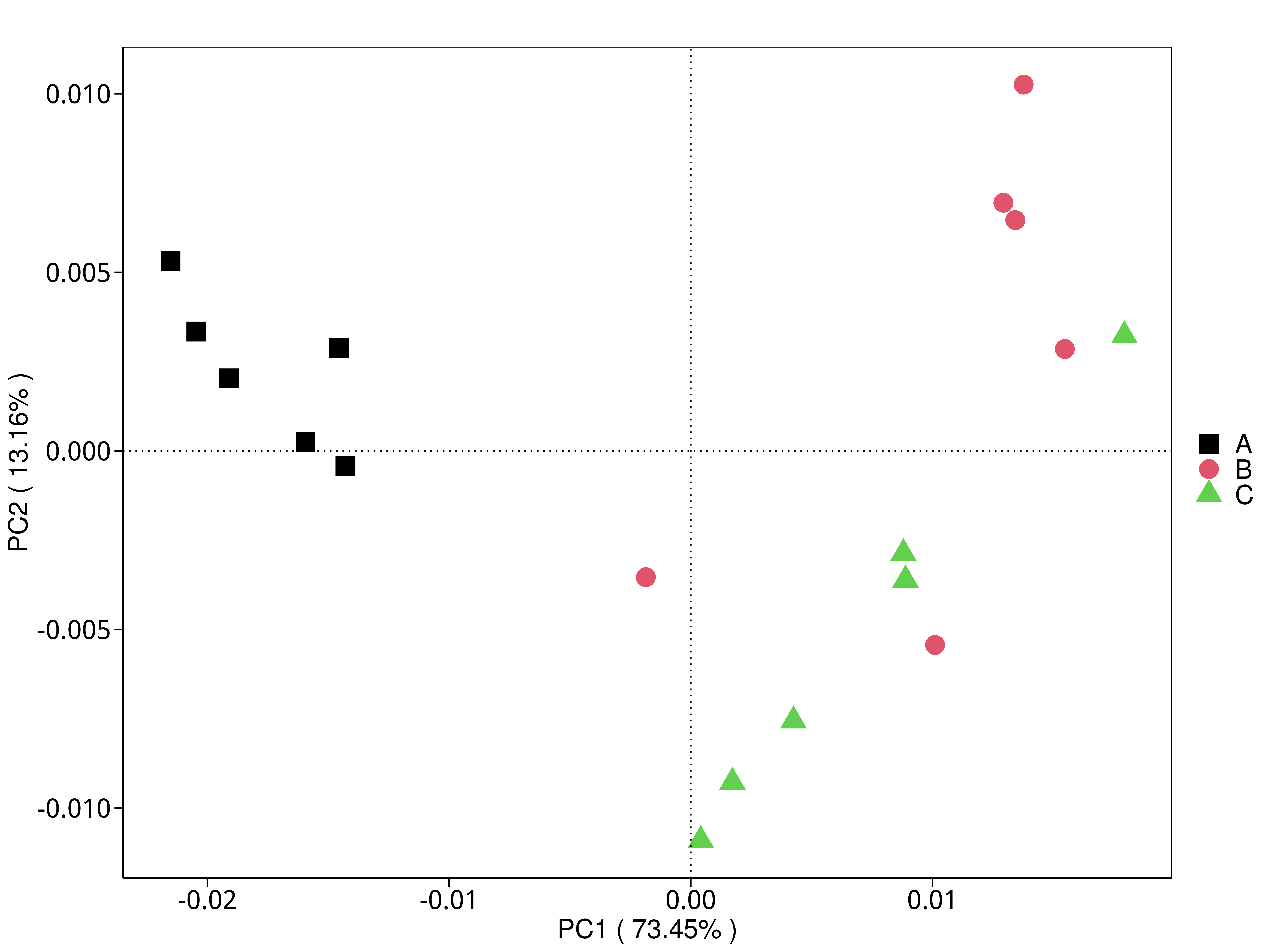
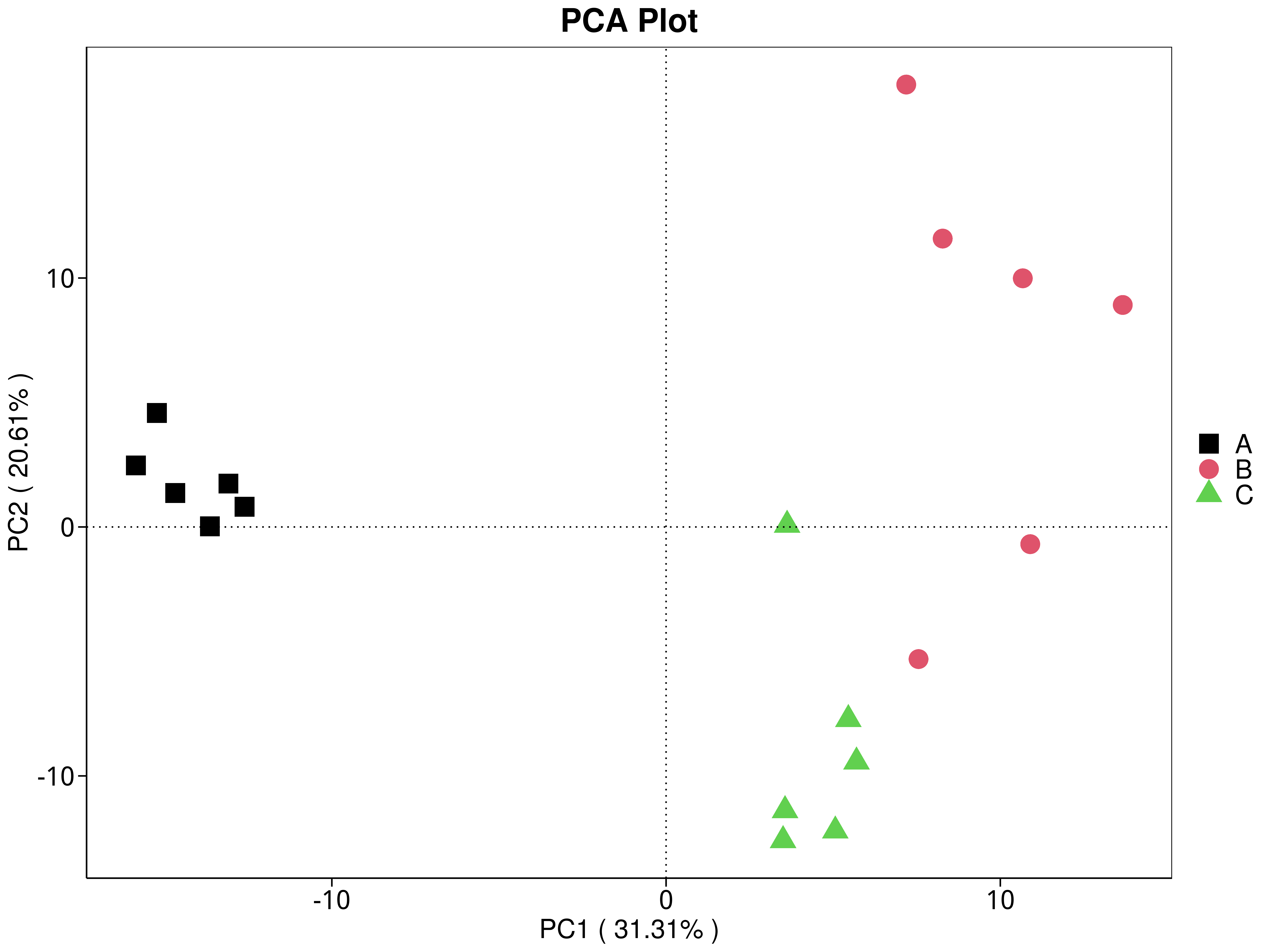
4.5.3 主成分(PCA)分析
主成分分析(PCA,Principal Component Analysis)是基于线型模型的一种降维分析,它应用方差分解的方法对多维数据进行降维,从而提取出数据中最主要的元素和结构(Rao et al., 2002);PCA 能够提取出最大程度反映样品间差异的两个坐标轴,从而将多维数据的差异反映在二维坐标图上,进而揭示复杂数据背景下的简单规律。
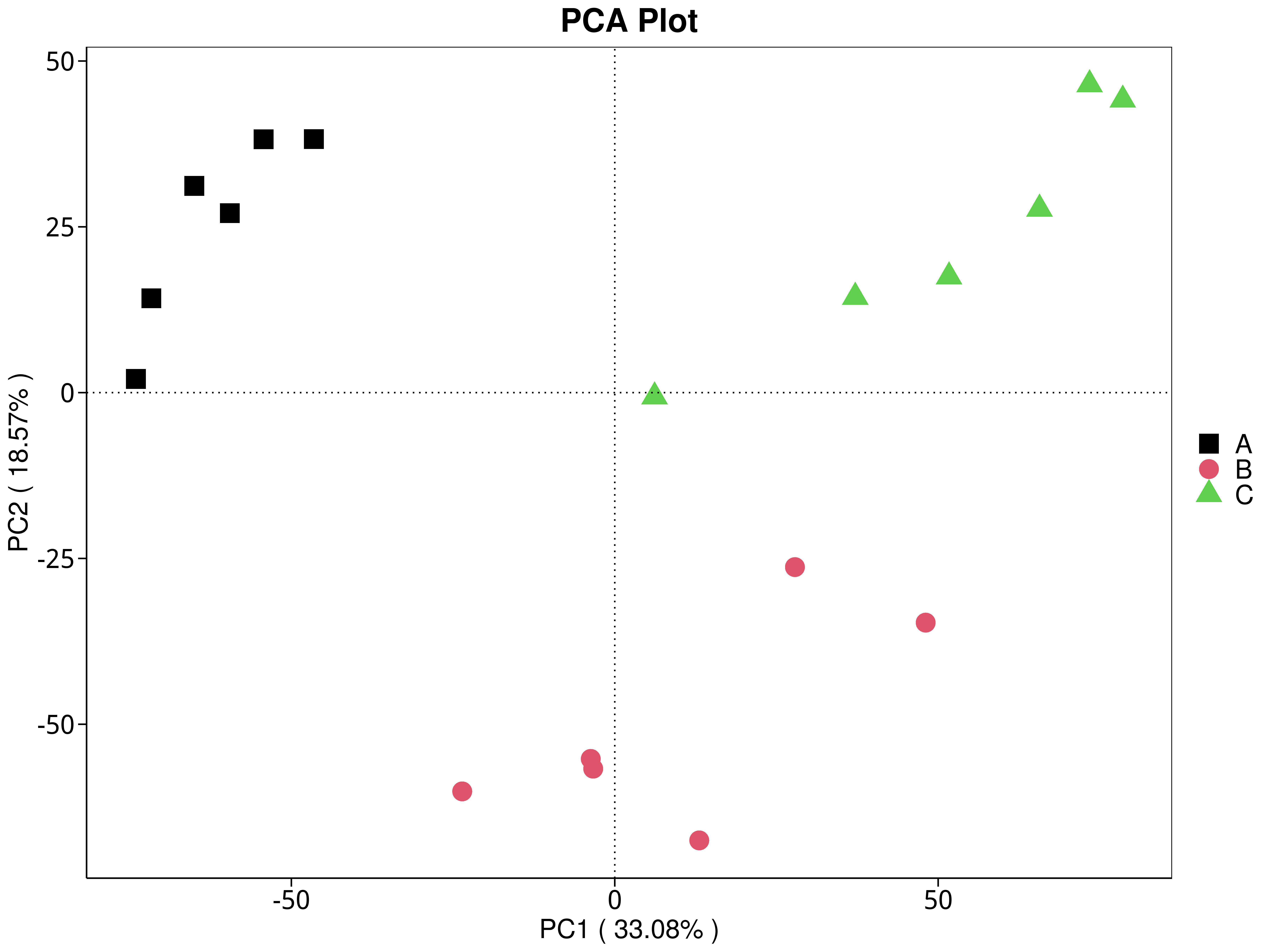
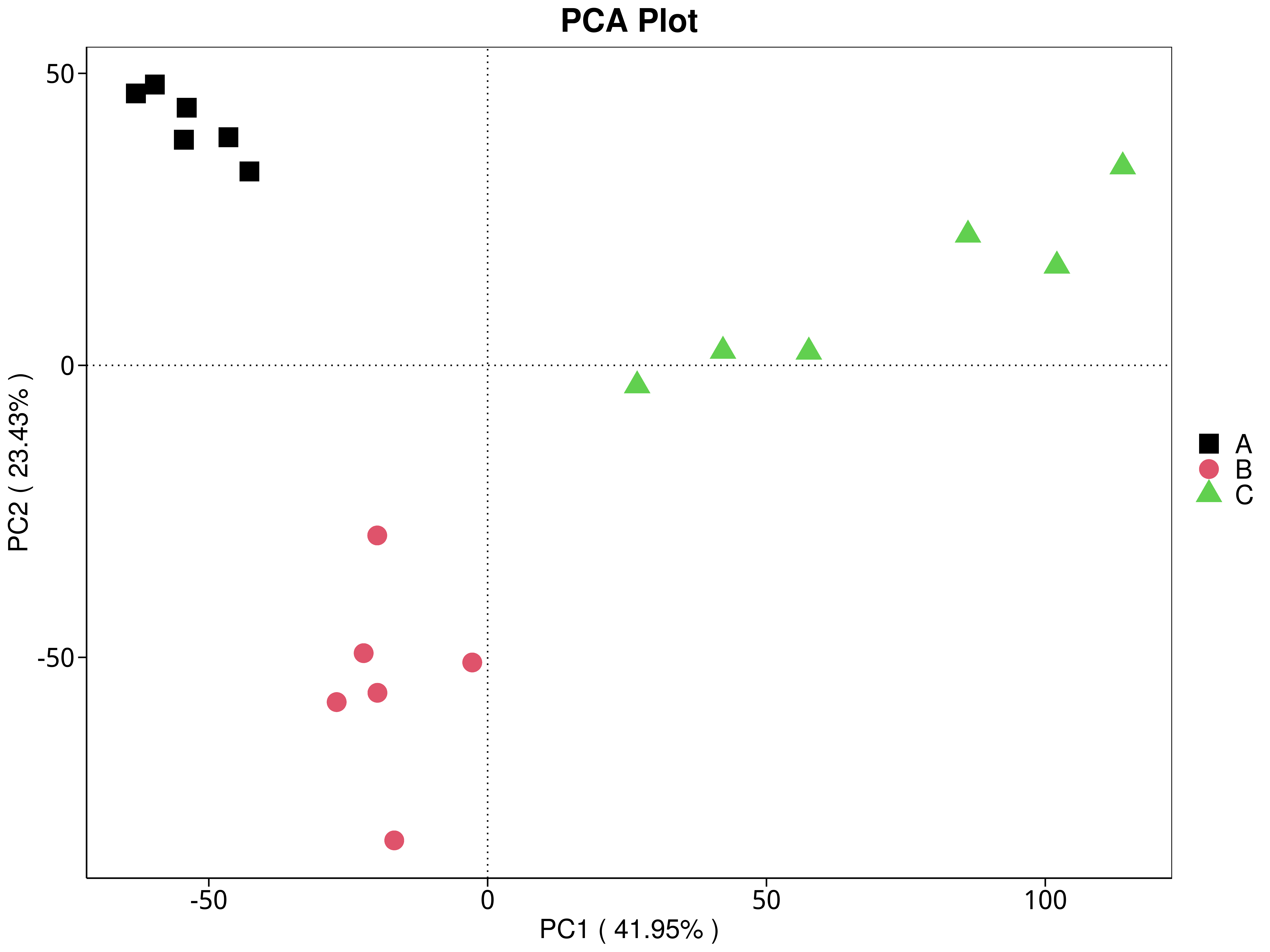
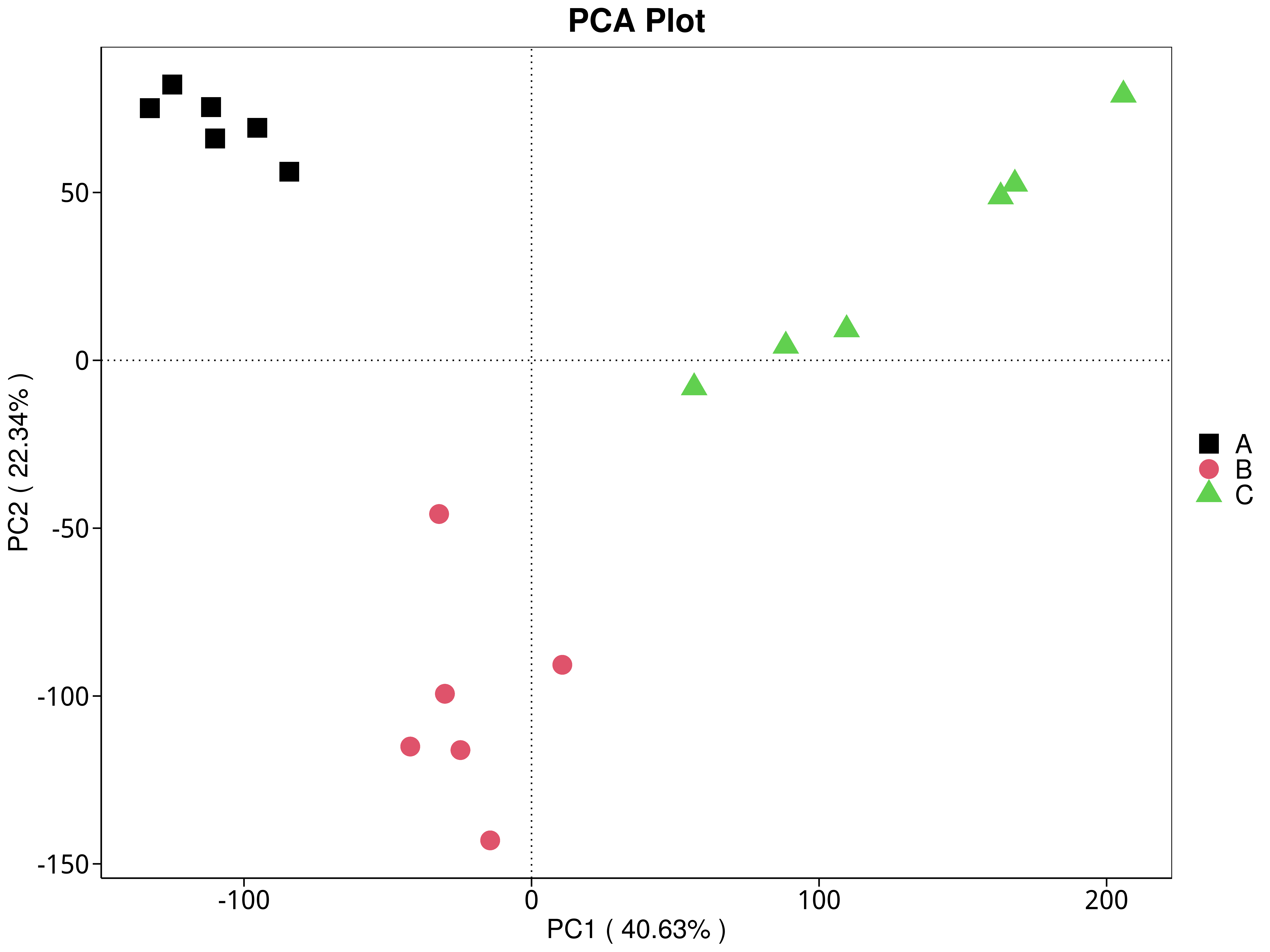
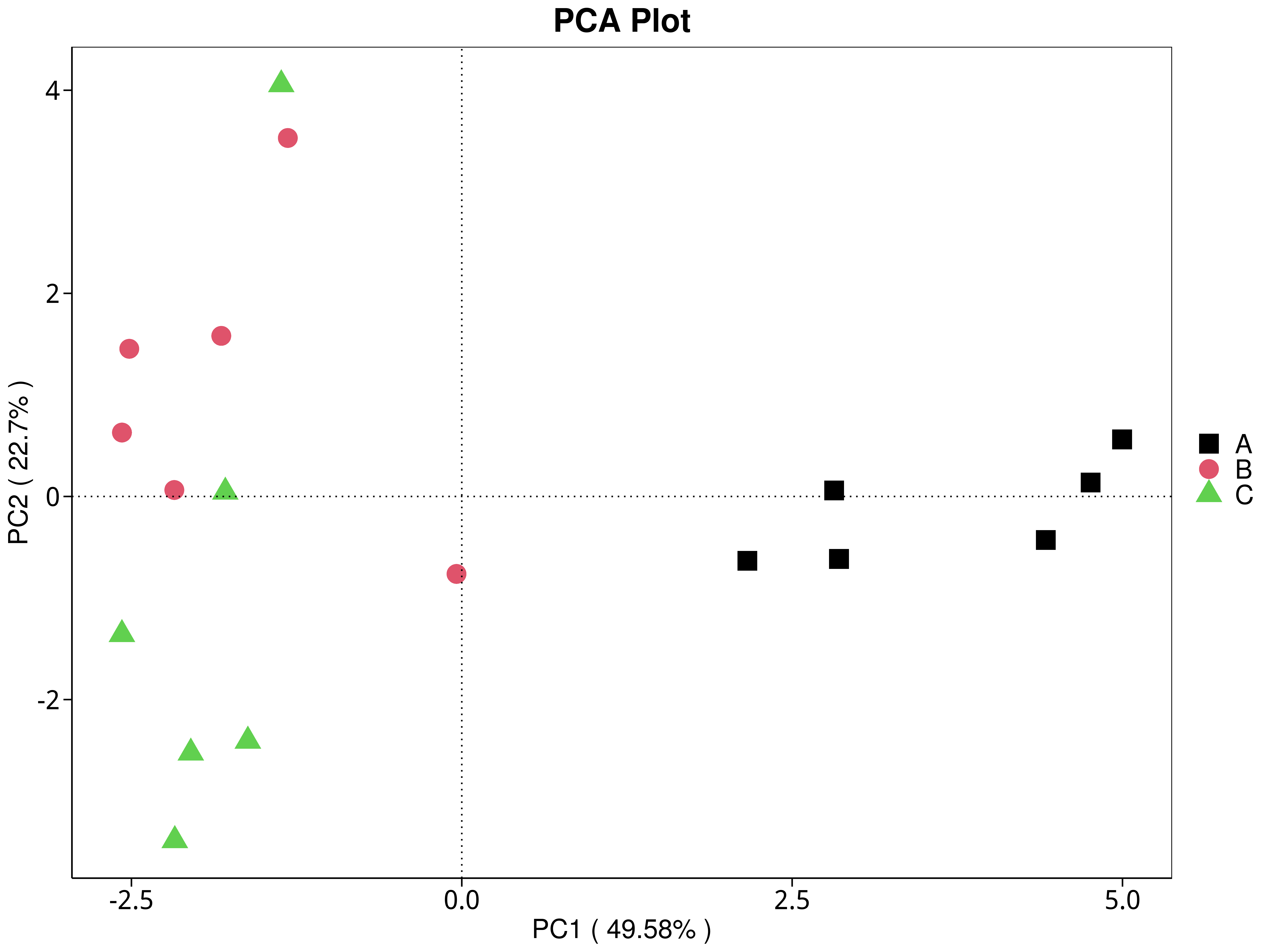
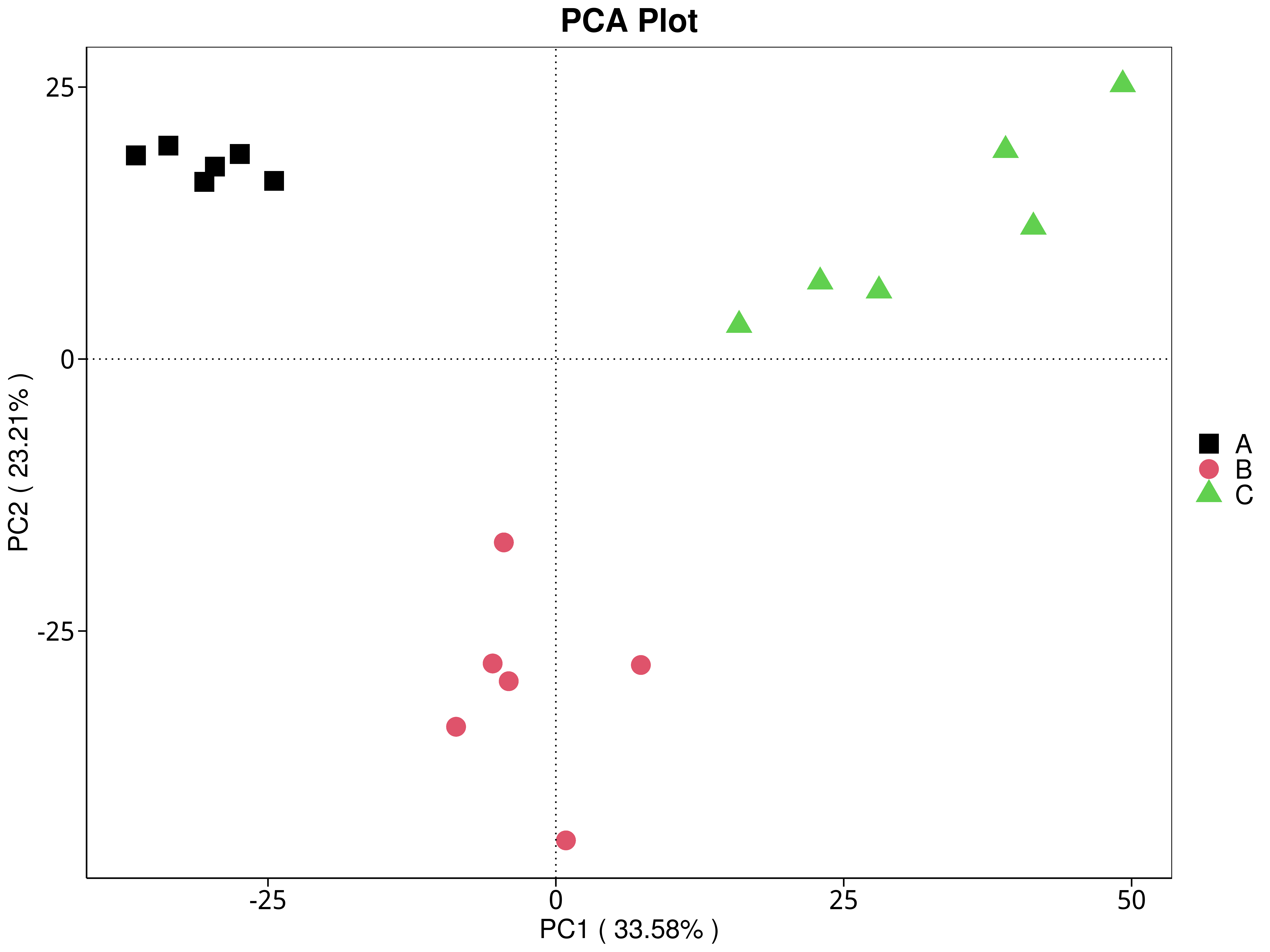
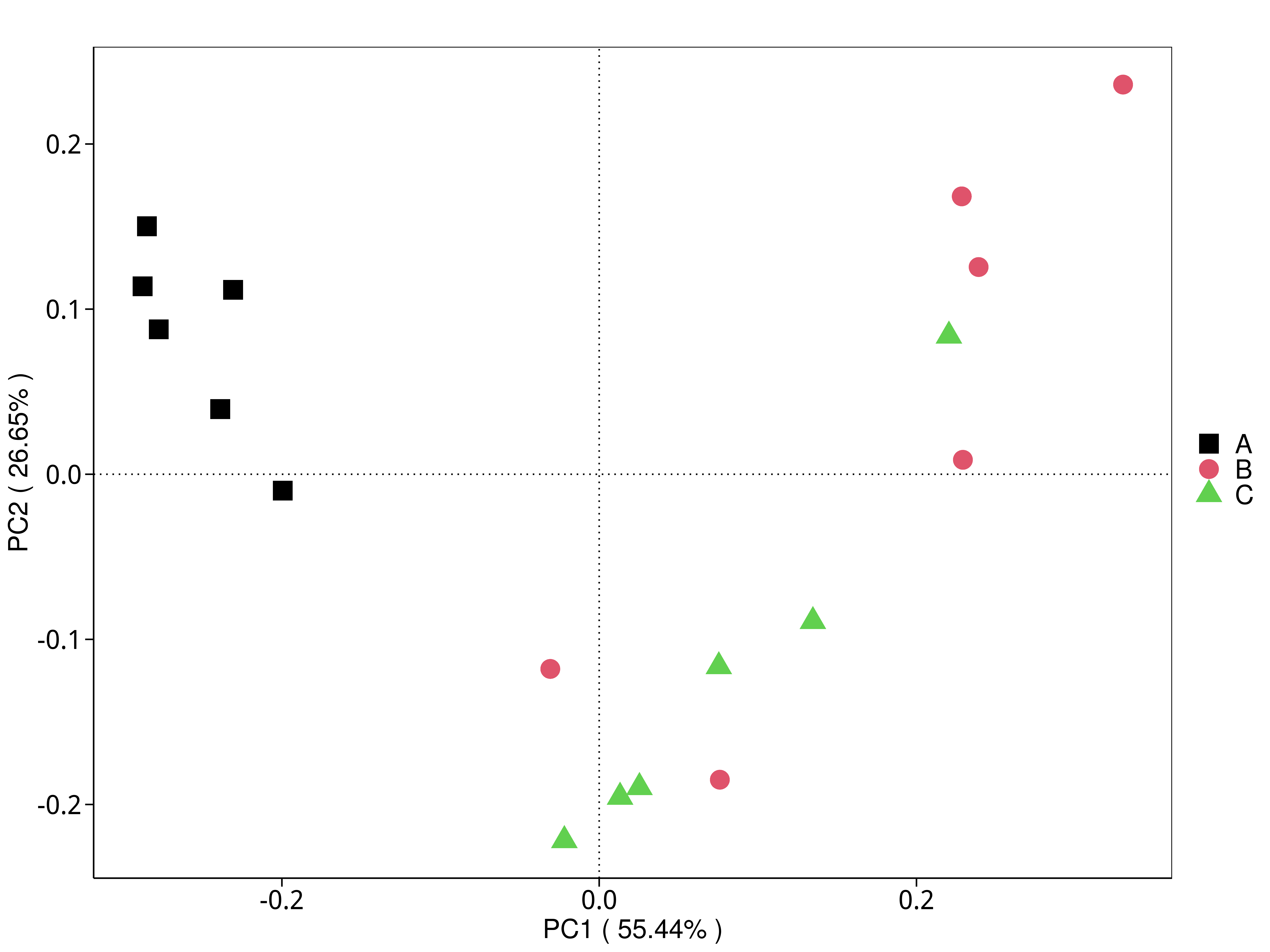
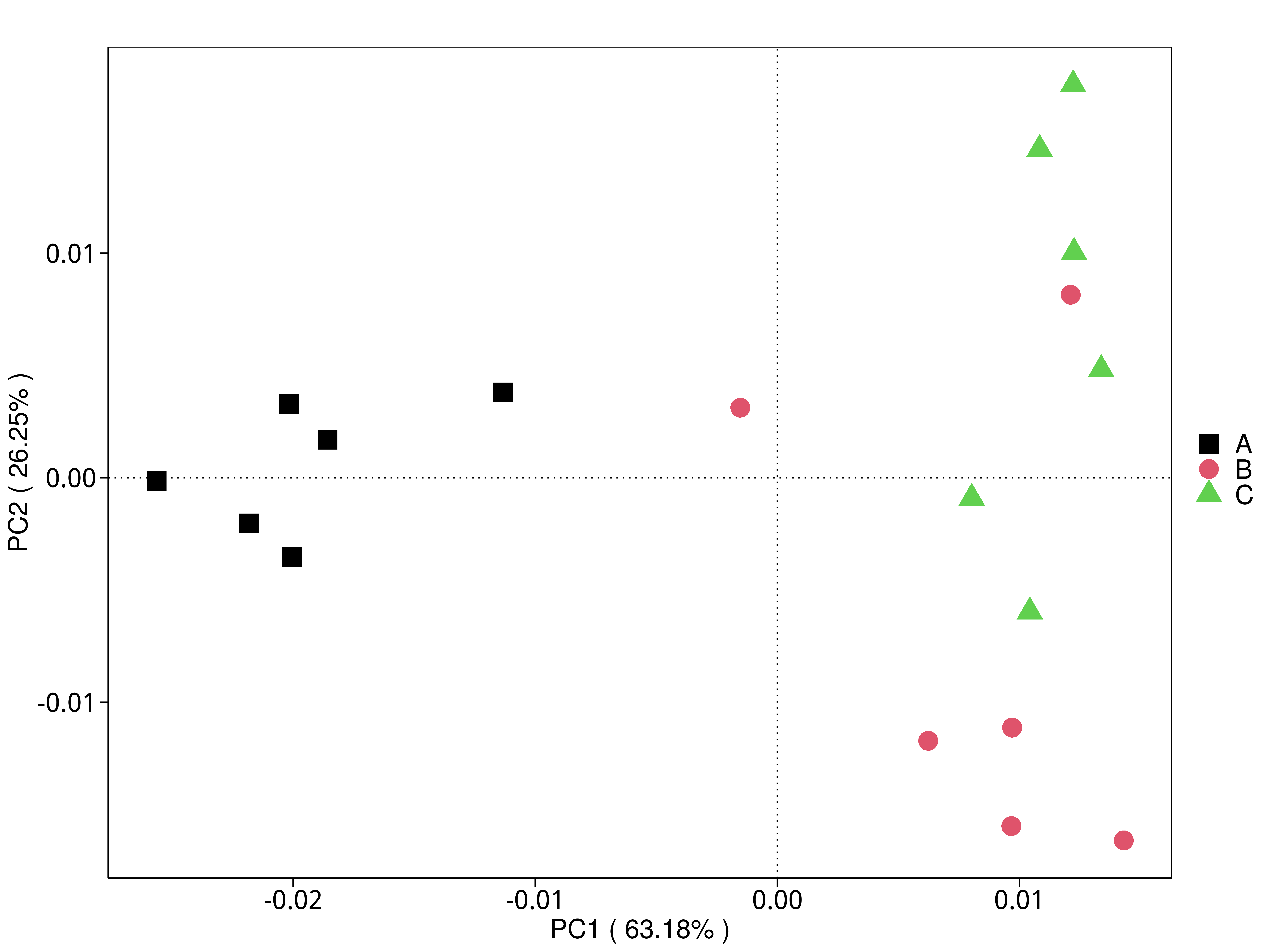
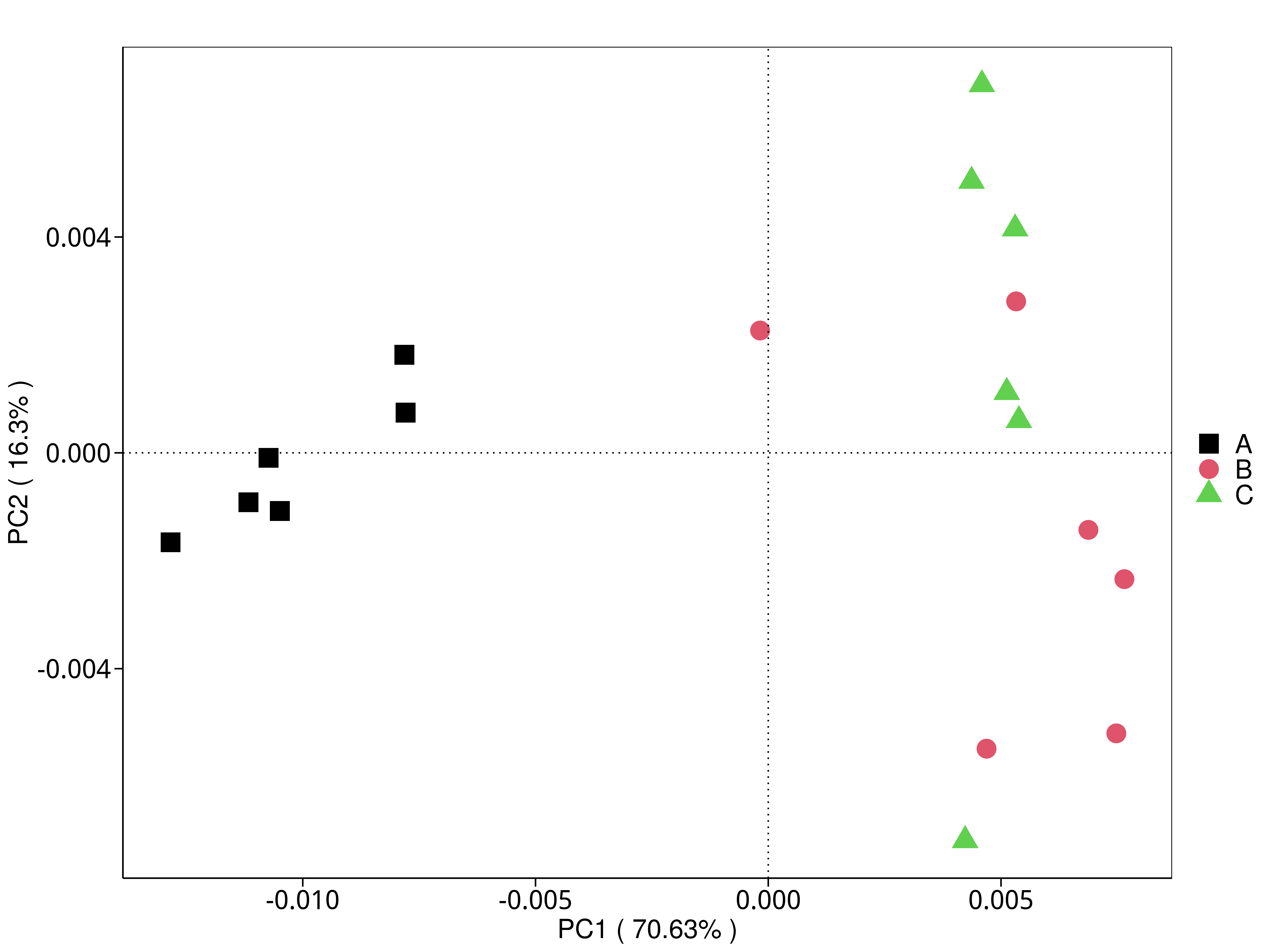
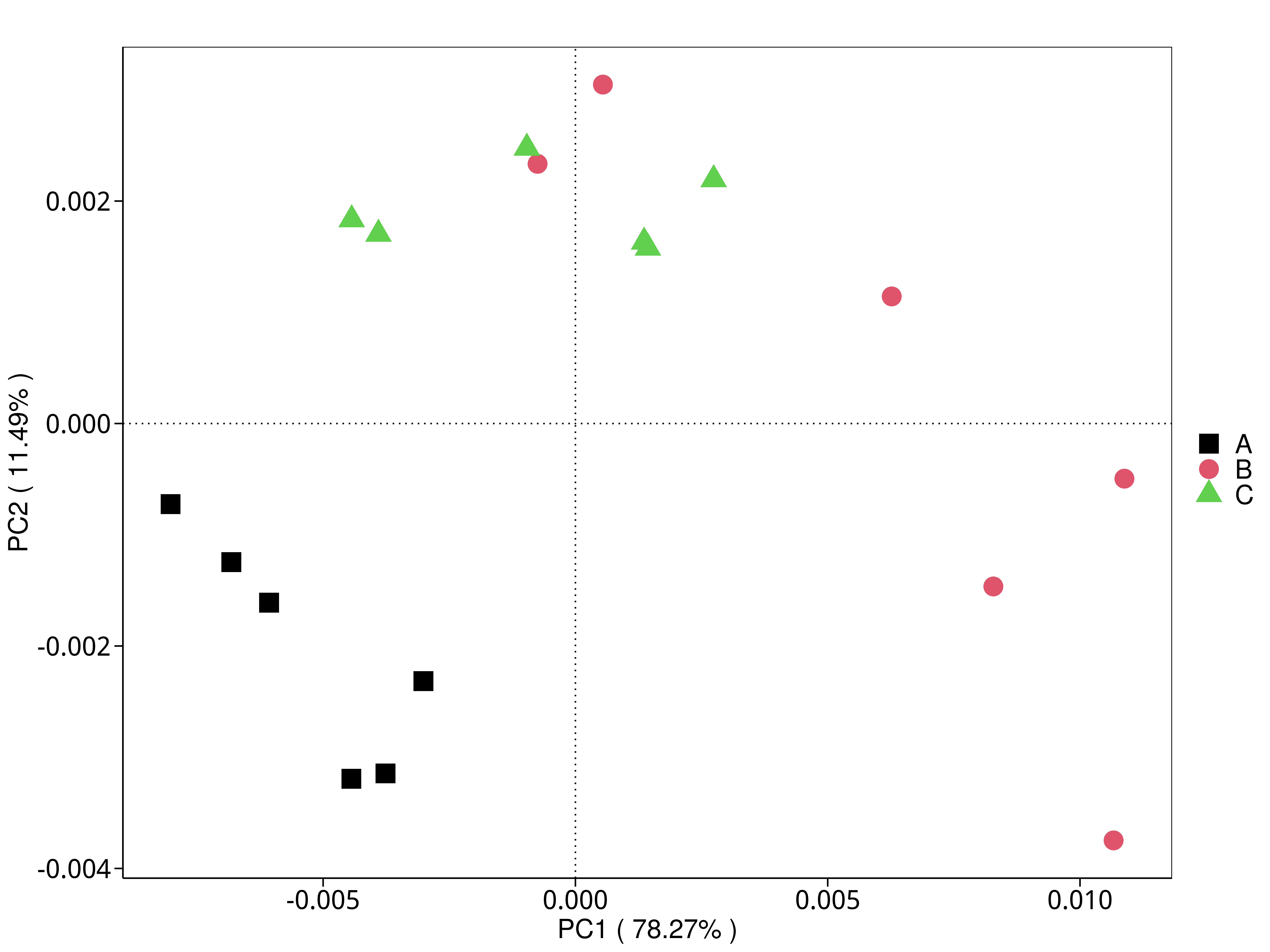
4.5.3.1 Micro_NR
图4.28 基于 Micro_NR 水平的 PCA 结果展示
说明:PCA分析,横坐标表示第一主成分,百分比则表示第一主成分对样品差异的贡献值;纵坐标表示第二主成分,百分比表示第二主成分对样品差异的贡献值;图中的每个点表示一个样品,同一个组的样品使用同一种颜色表示,没有分组默认按照样本出图
结果目录:
标注样品名的PCA图见 : result/05.Diversity/MicroNR/PCA_group1/*/PCA_lable.{png,pdf}
未标注样品名的PCA图见 : result/05.Diversity/MicroNR/PCA_group1/*/PCA.{png,pdf}
未标示样品名称的带聚类圈的PCA图见 : result/05.Diversity/MicroNR/PCA_group1/*/PCA_circle.{png,pdf}
标示样品名称的带聚类圈的PCA图见 : result/05.Diversity/MicroNR/PCA_group1/*/PCA_circleLable.{png,pdf}
各个主成分分析结果见 : result/05.Diversity/MicroNR/PCA_group1/*/PCA.xls
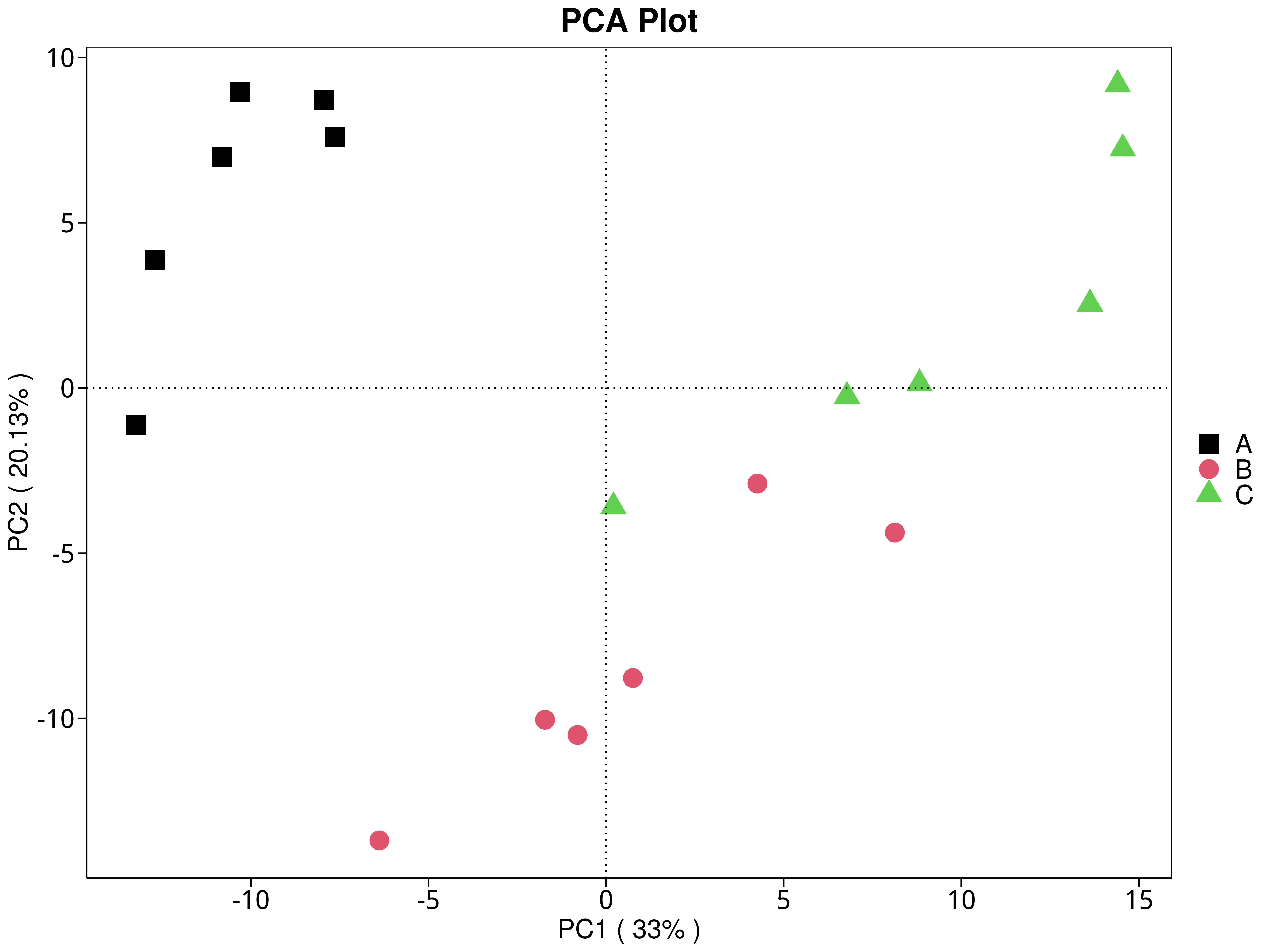
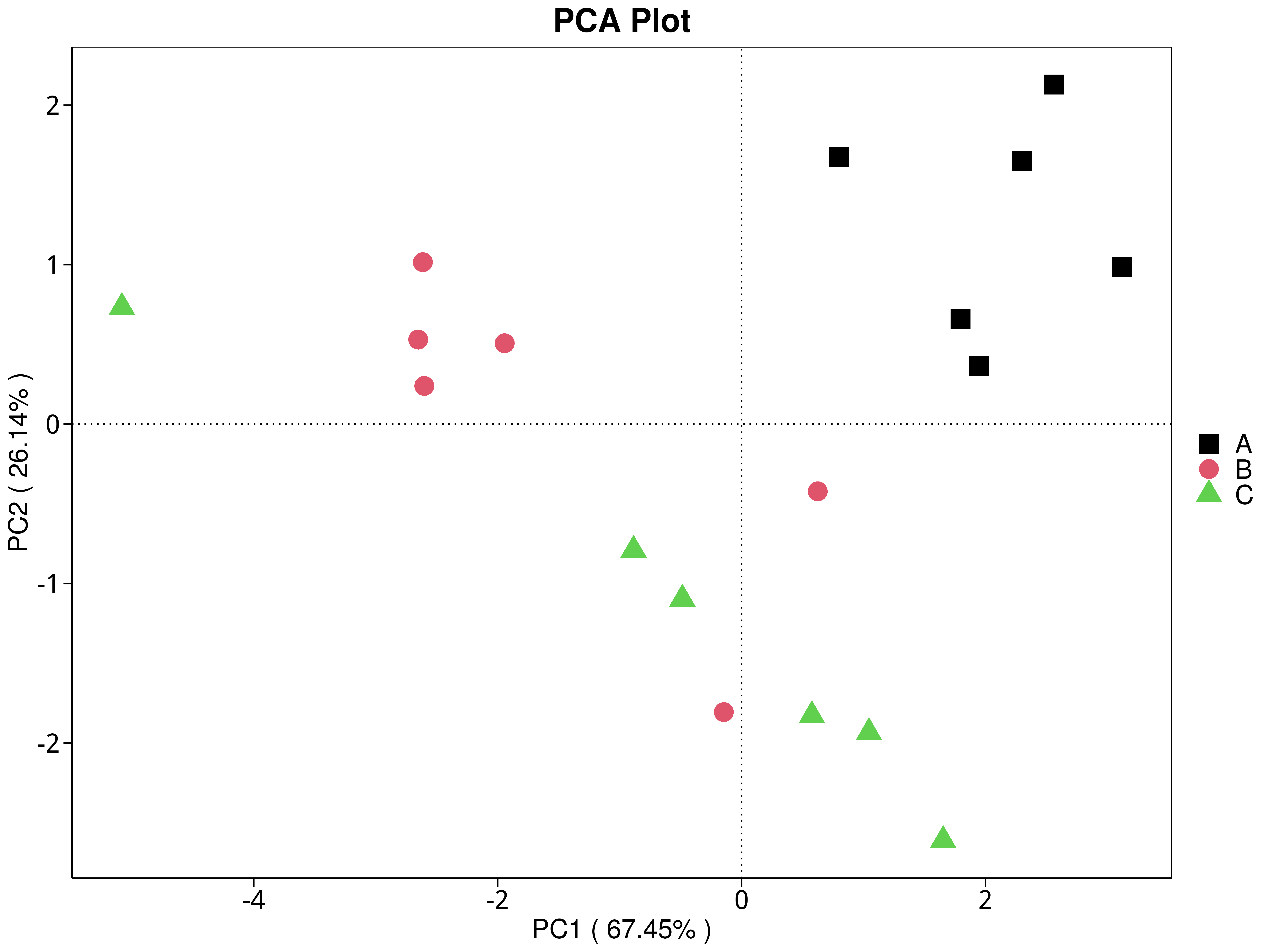
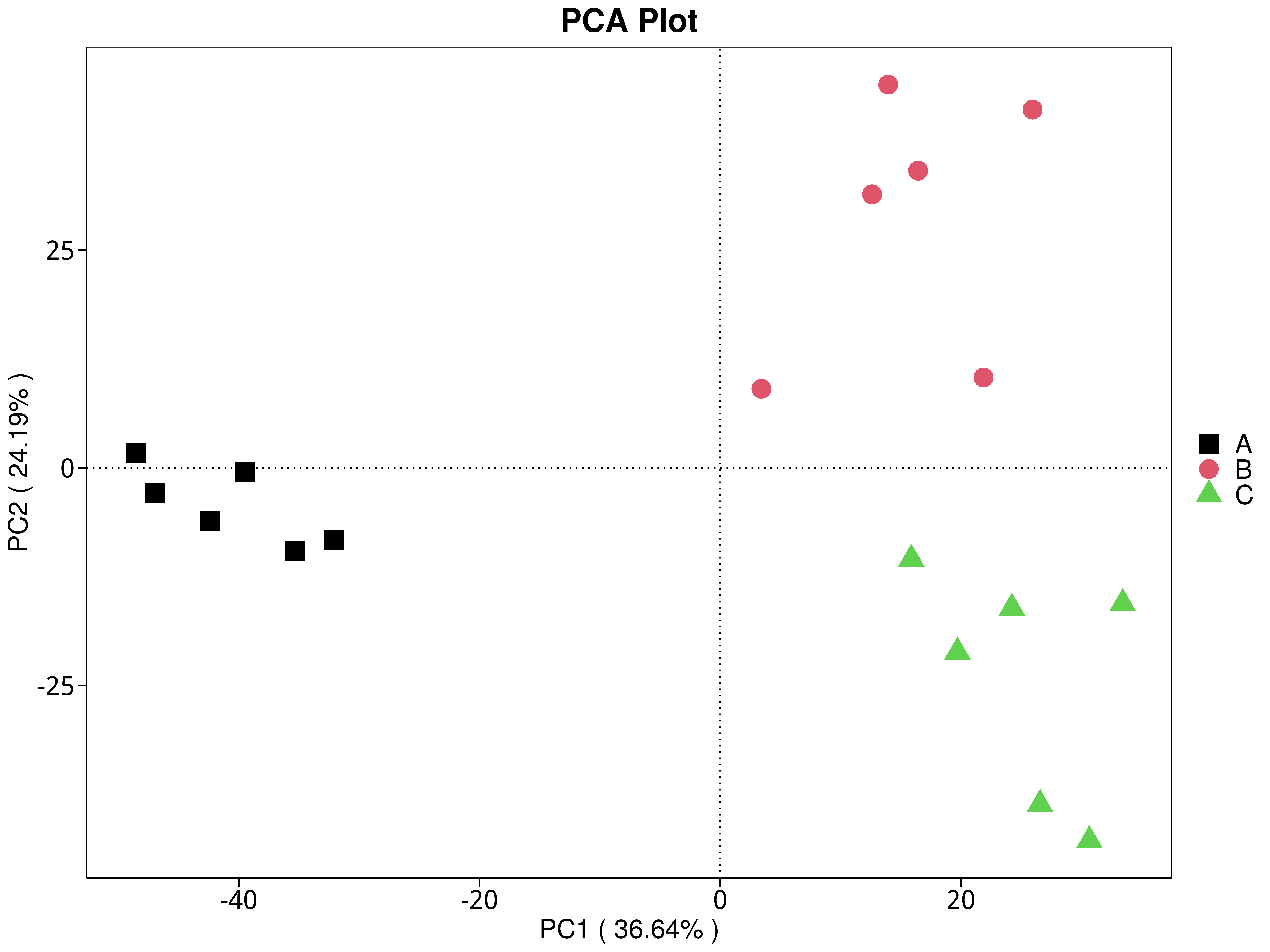
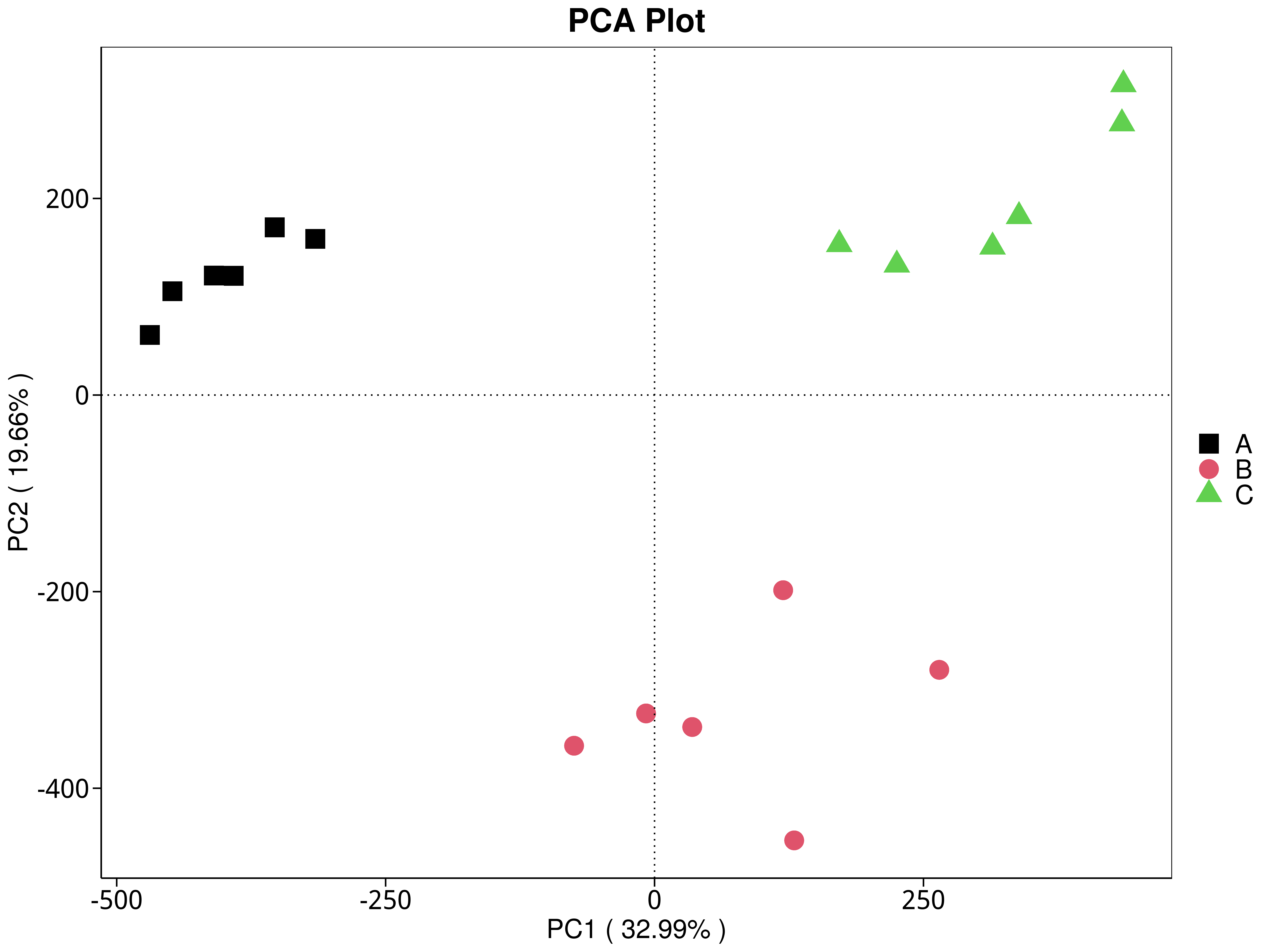
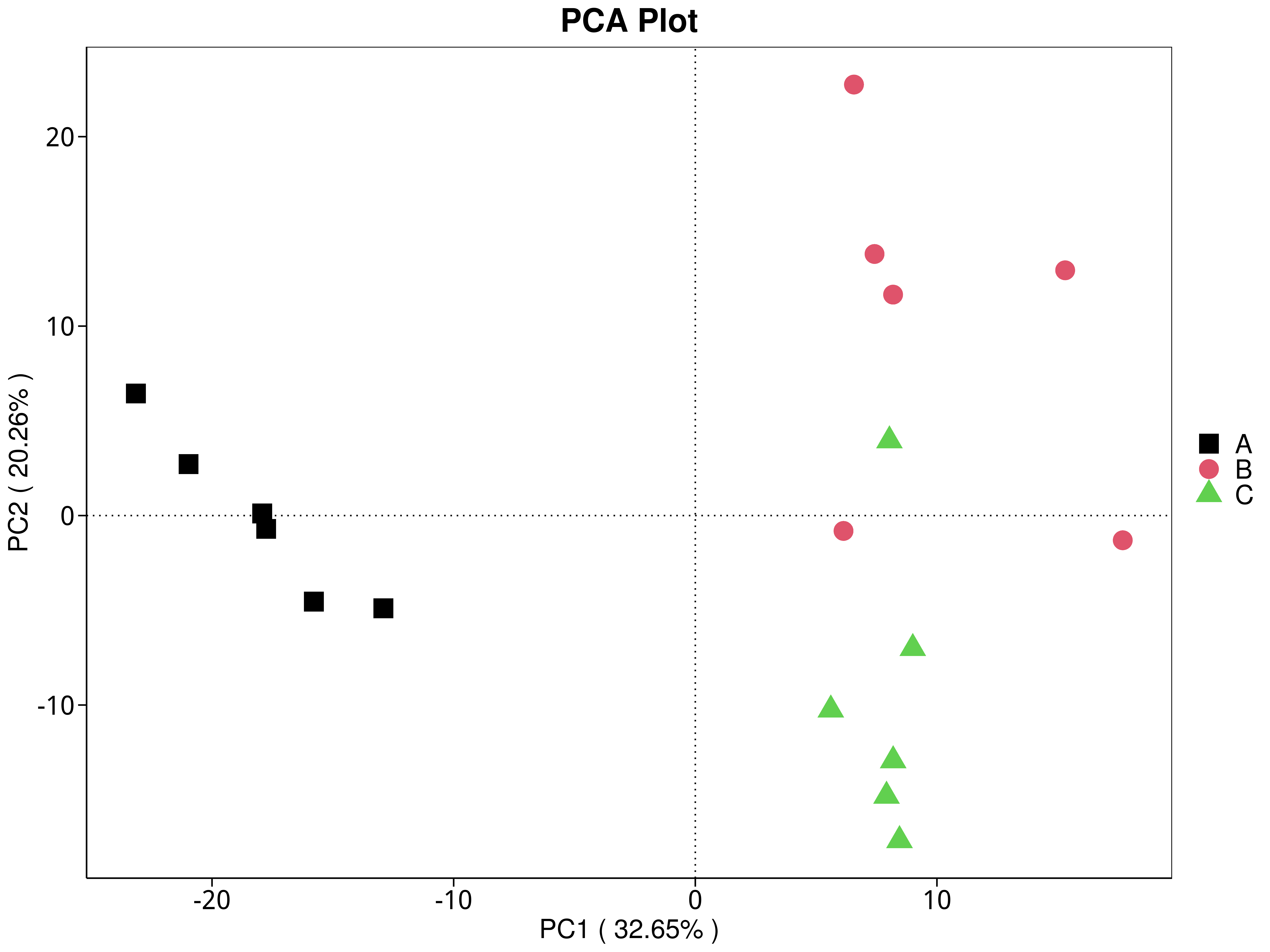
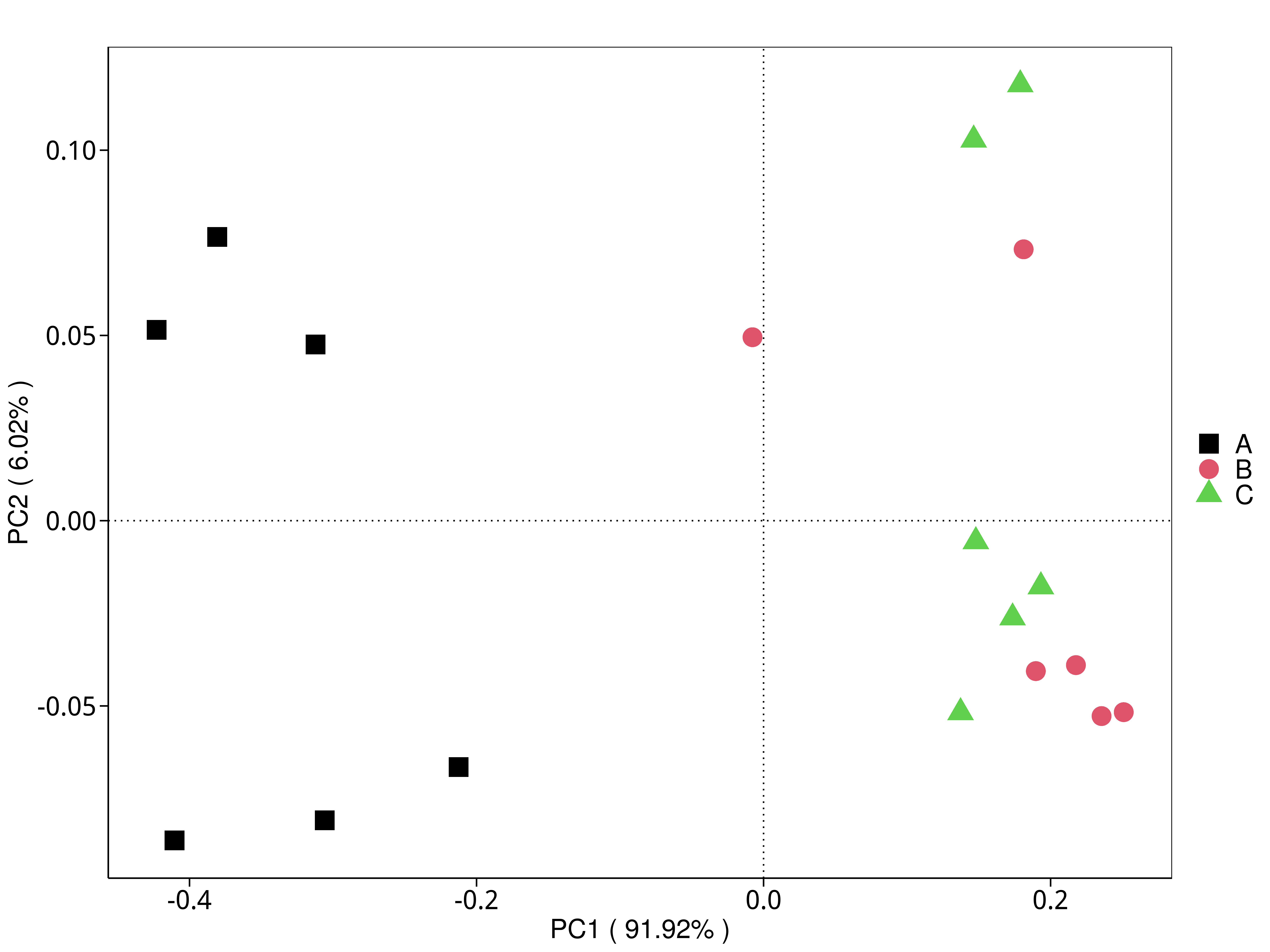
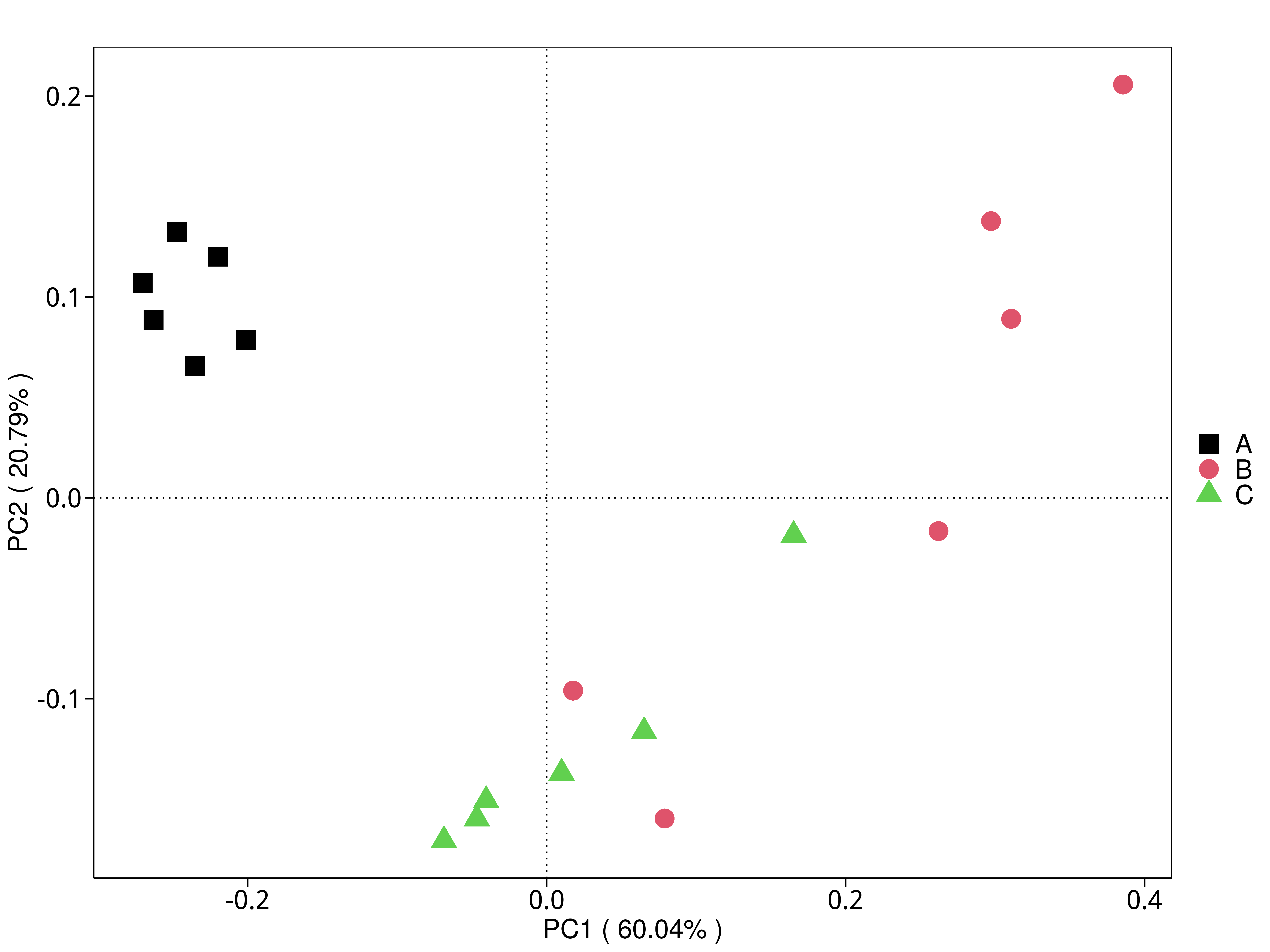
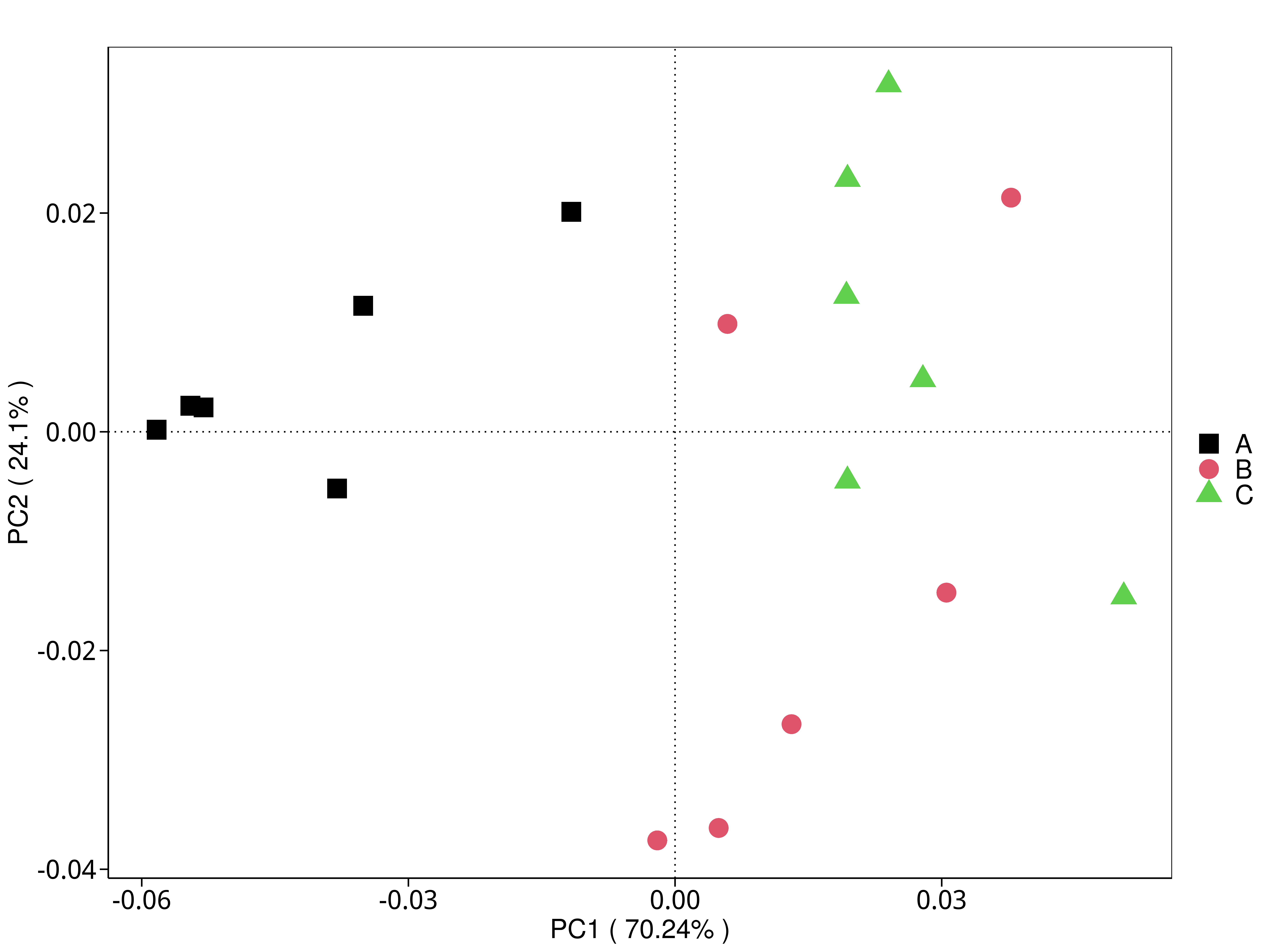
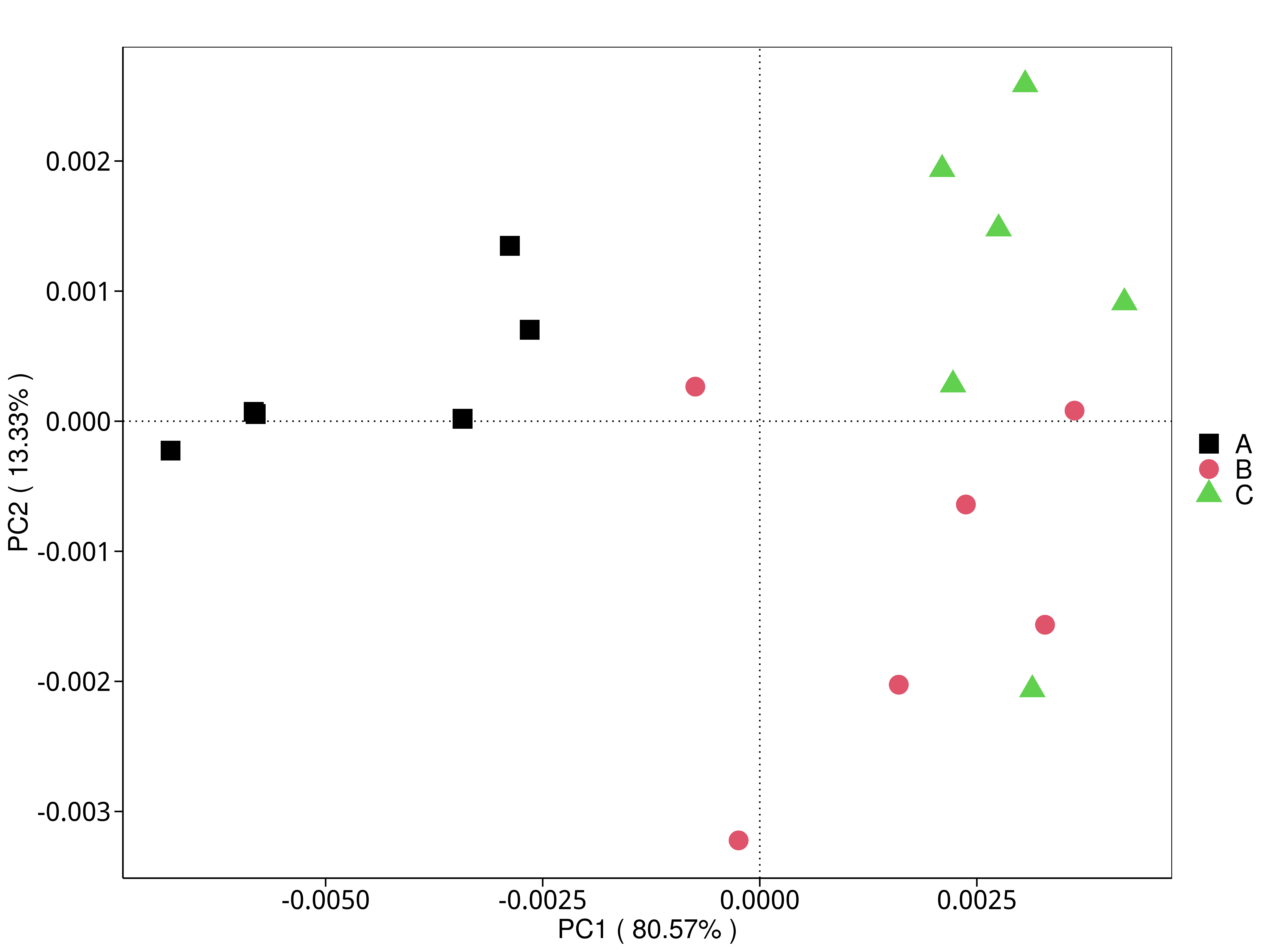
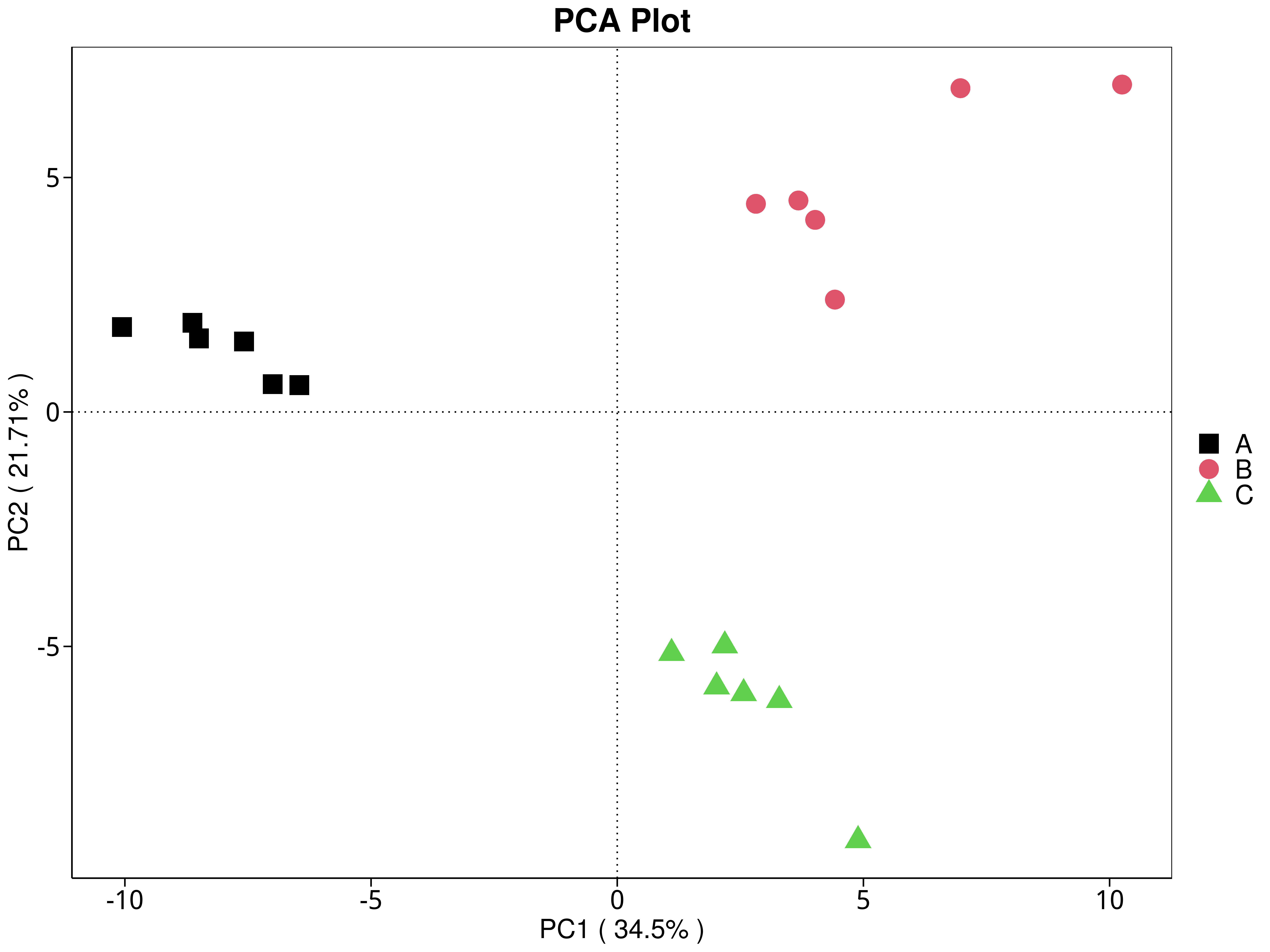
4.5.3.2 KEGG
图4.29 基于 KEGG 水平的 PCA 结果展示
说明:功能水平PCA分析,横坐标表示第一主成分,百分比则表示第一主成分对样品差异的贡献值;纵坐标表示第二主成分,百分比表示第二主成分对样品差异的贡献值;图中的每个点表示一个样品,同一个组的样品使用同一种颜色表示,没有分组默认按照样本出图
结果目录:
标注样品名的PCA图见 : result/05.Diversity/KEGG/PCA_group1/*/PCA_lable.{png,pdf}
未标注样品名的PCA图见 : result/05.Diversity/KEGG/PCA_group1/*/PCA.{png,pdf};
未标示样品名称的带聚类圈的PCA图见 : result/05.Diversity/KEGG/PCA_group1/*/PCA_circle.{png,pdf}
标示样品名称的带聚类圈的PCA图见 : result/05.Diversity/KEGG/PCA_group1/*/PCA_circleLable.{png,pdf}
各个主成分分析结果见 : result/05.Diversity/KEGG/PCA_group1/*/PCA.xls
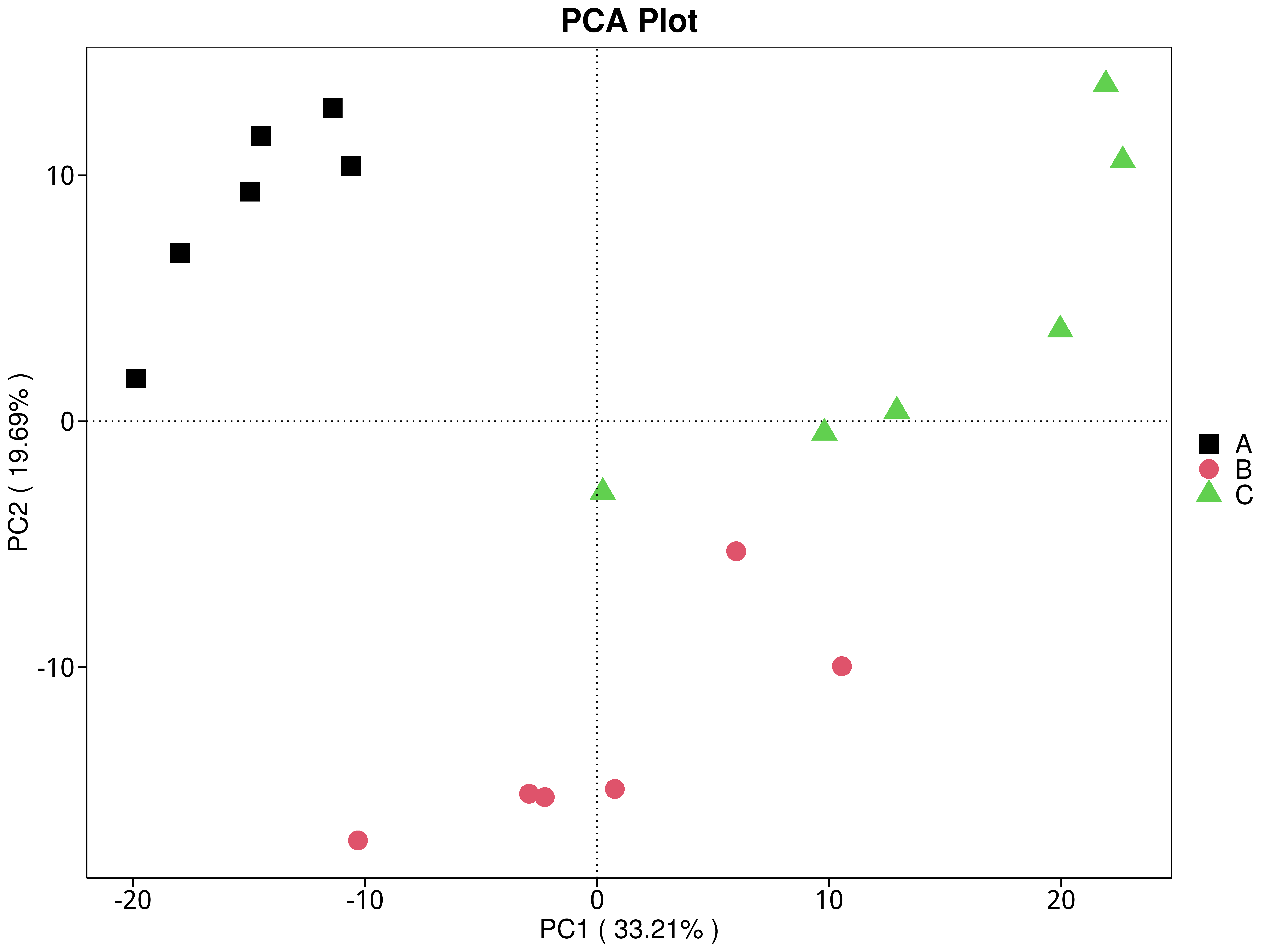
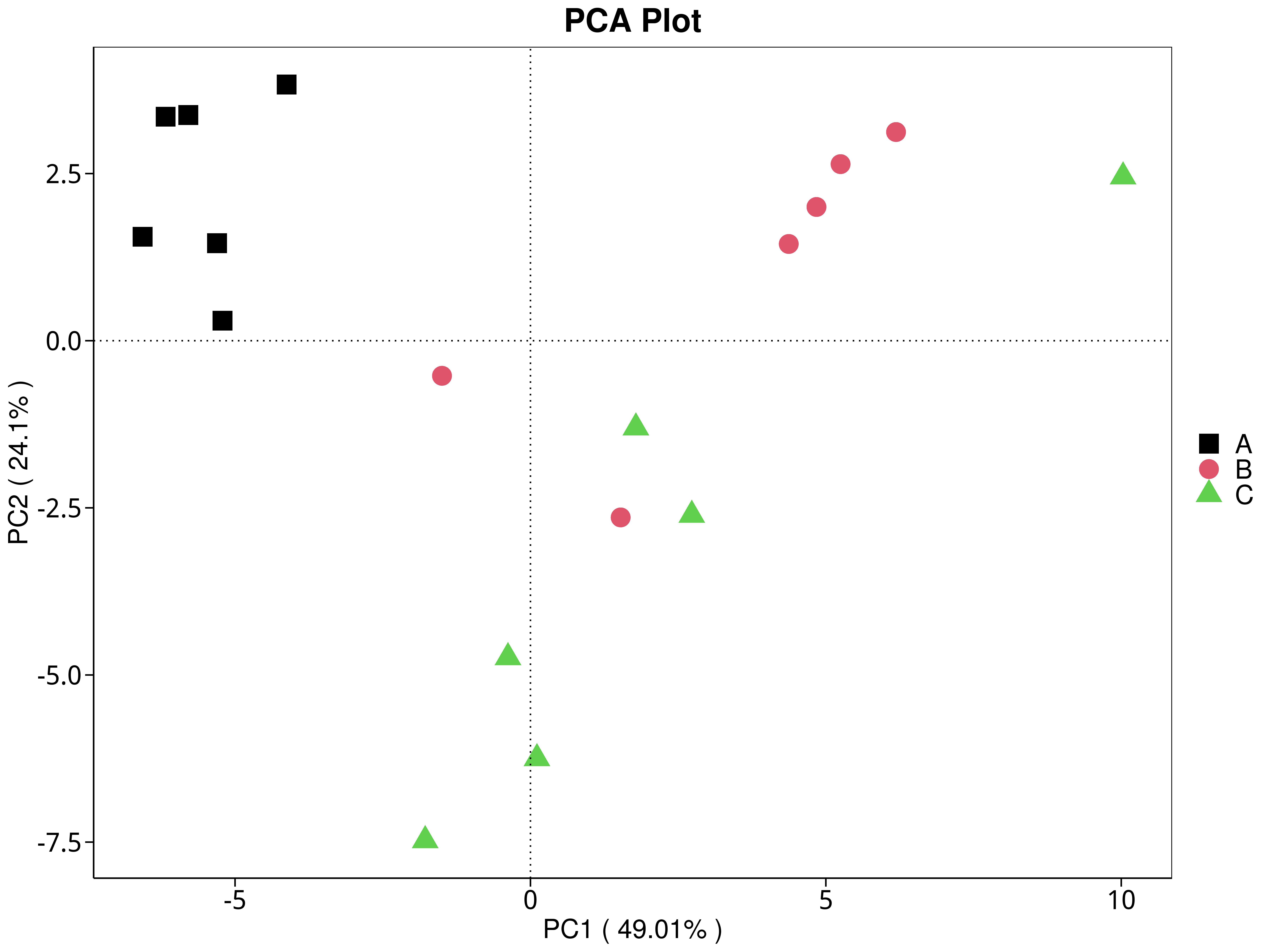
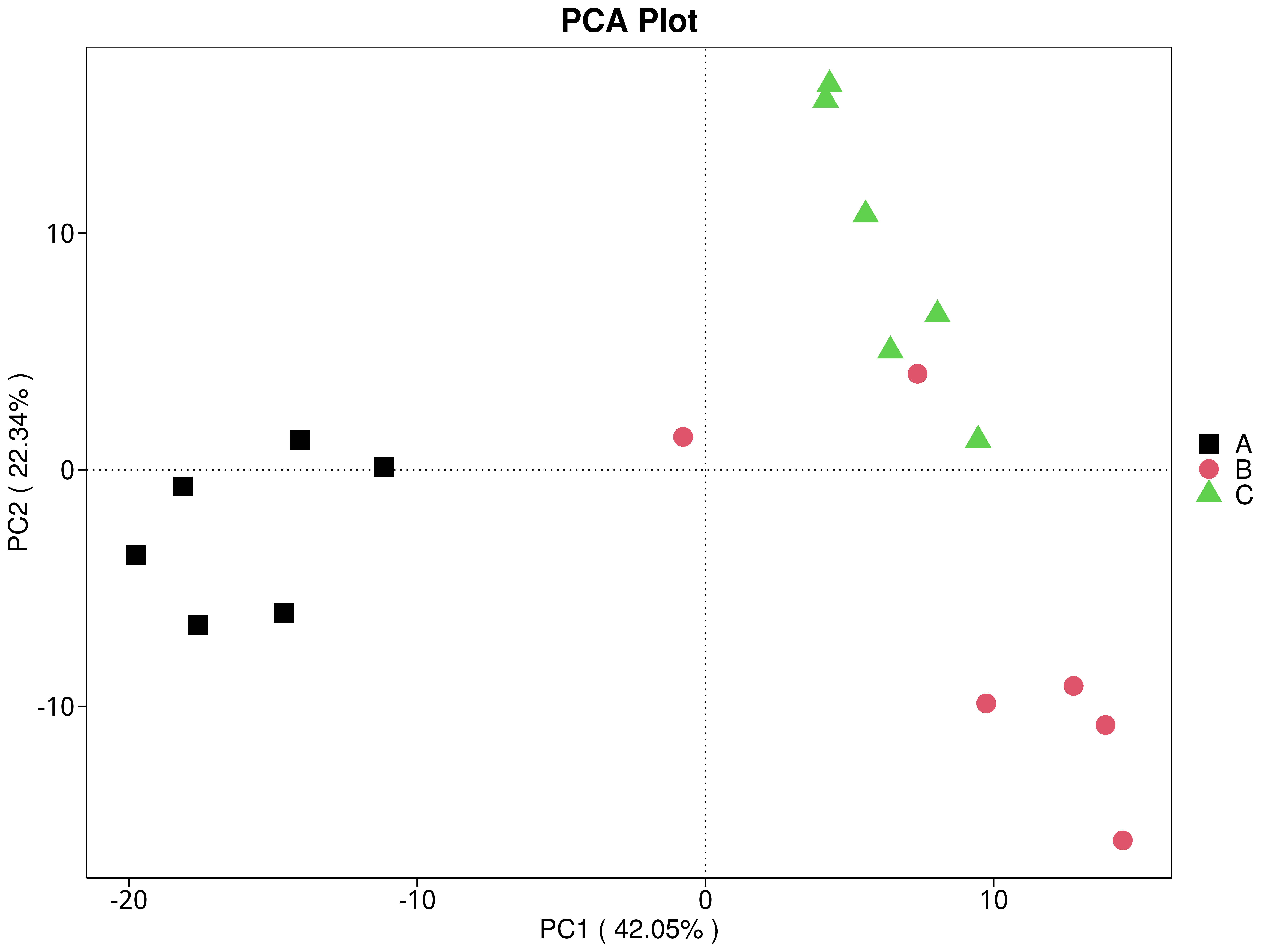
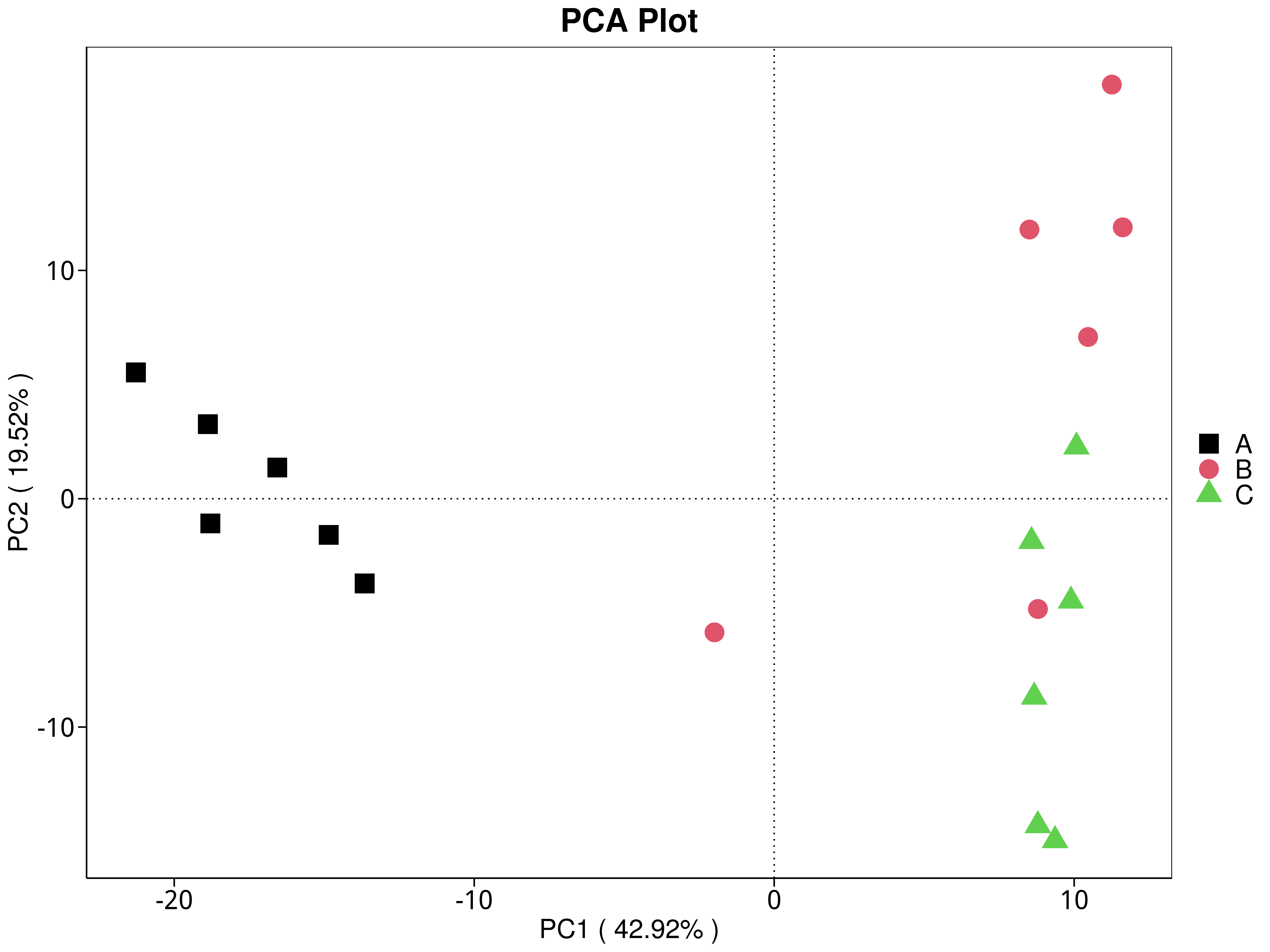
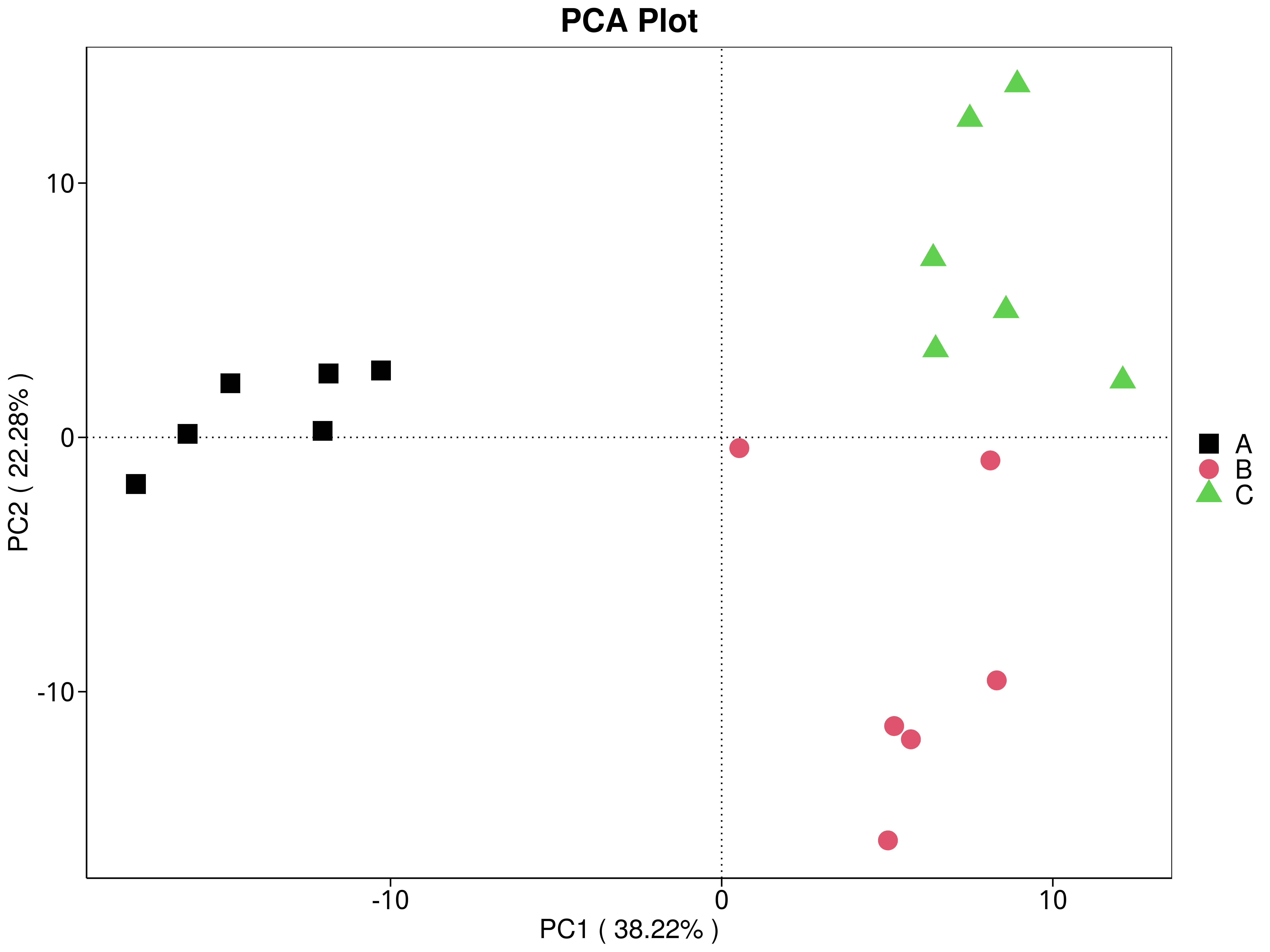
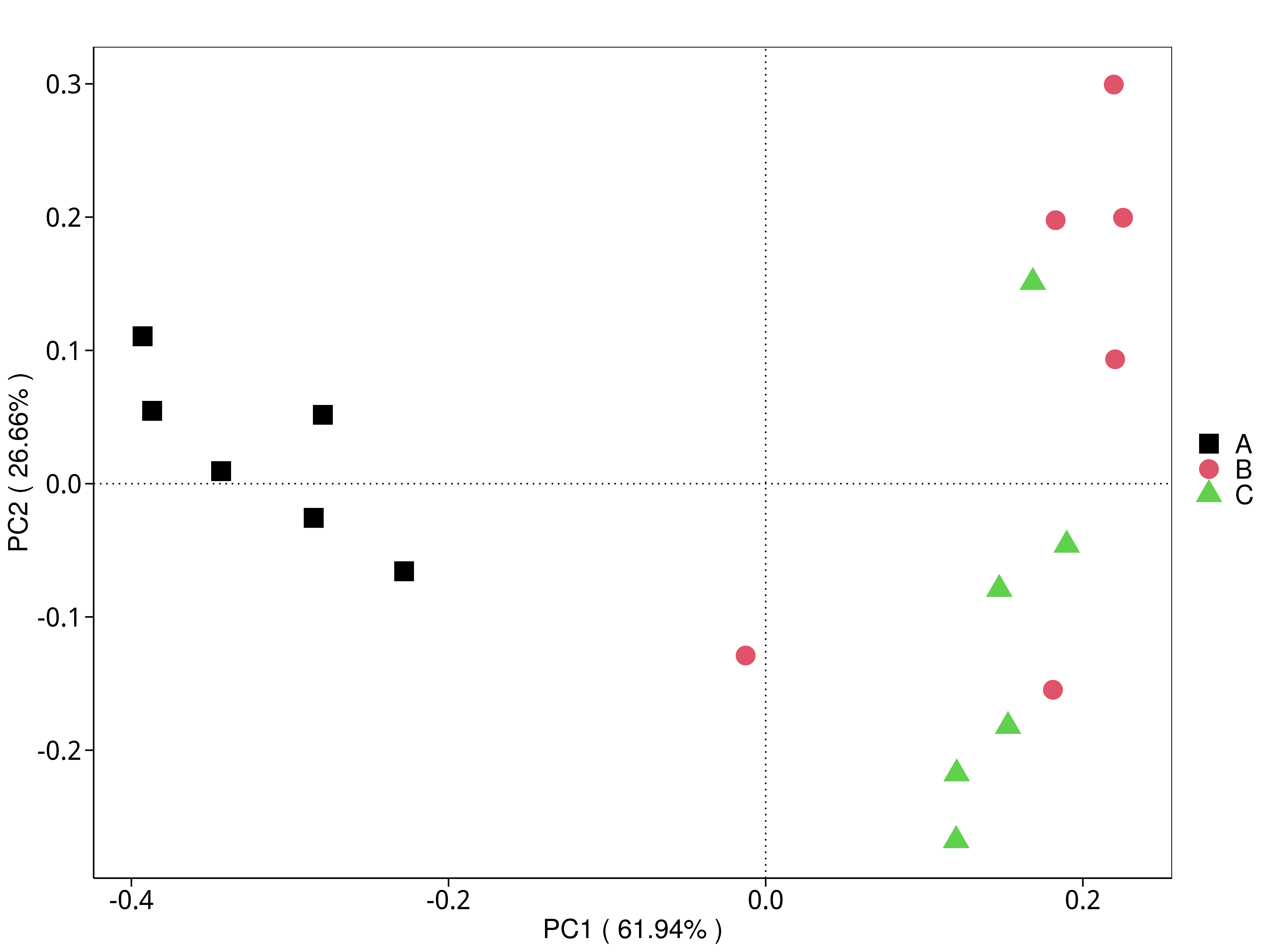
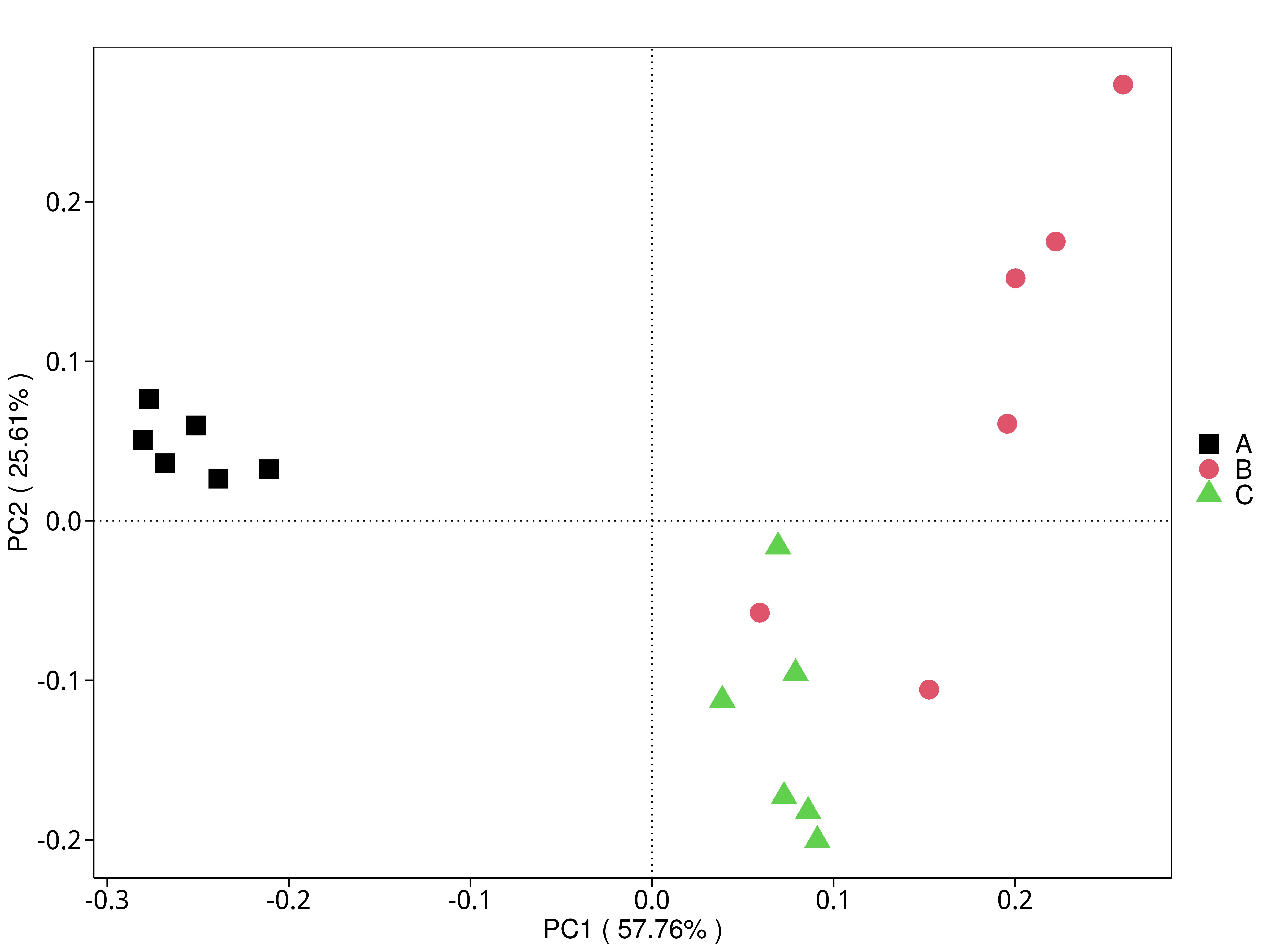
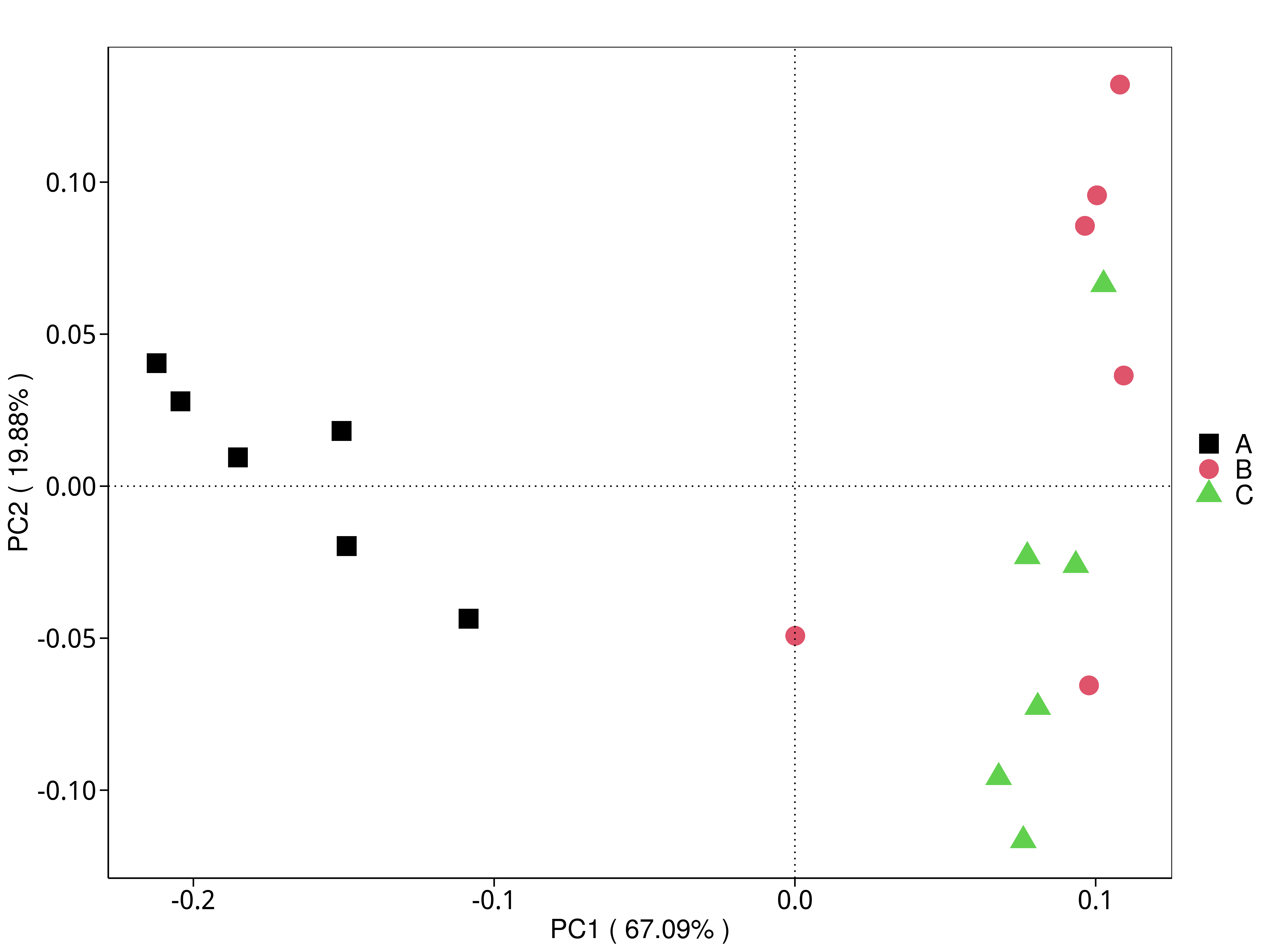
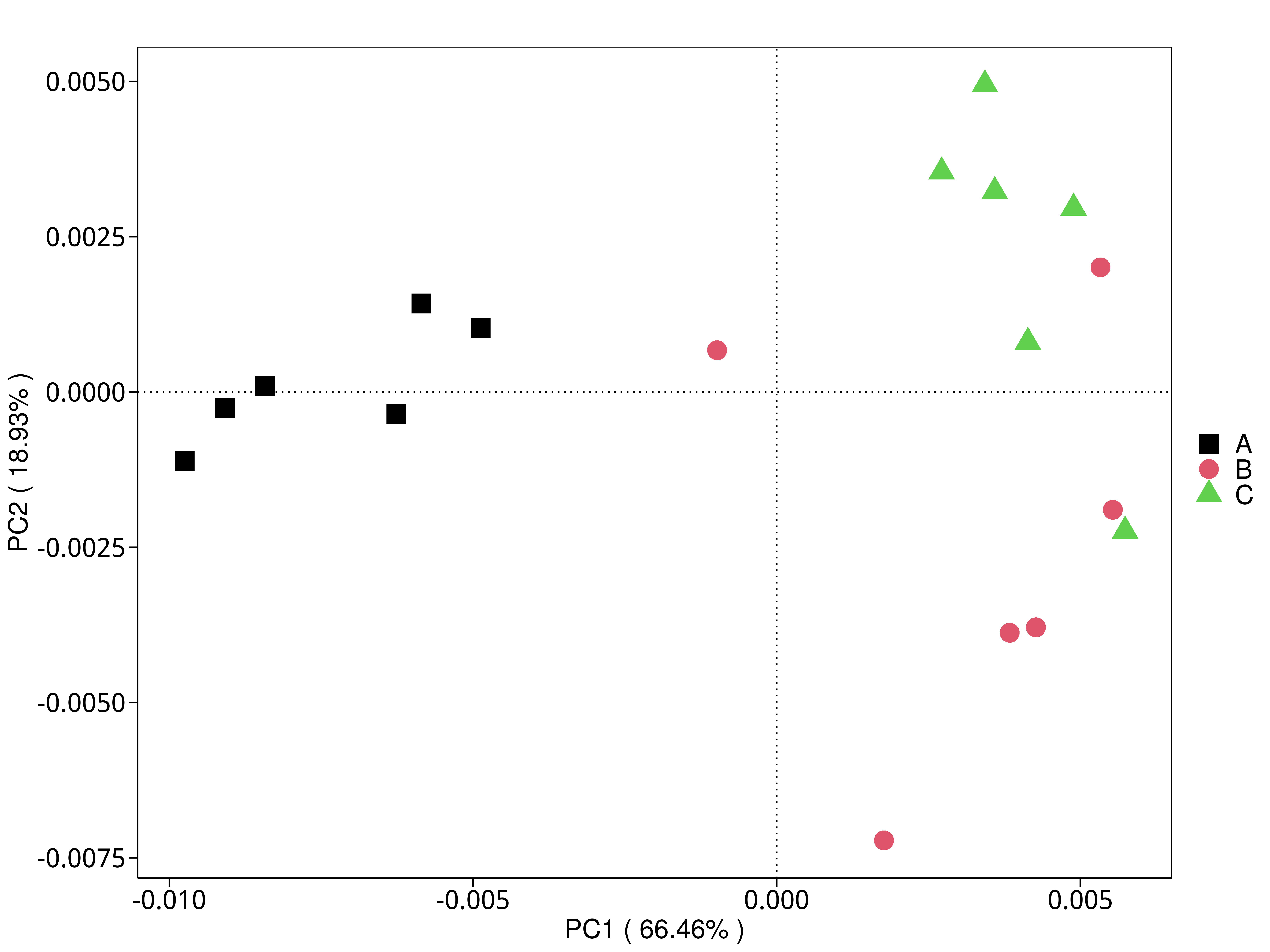
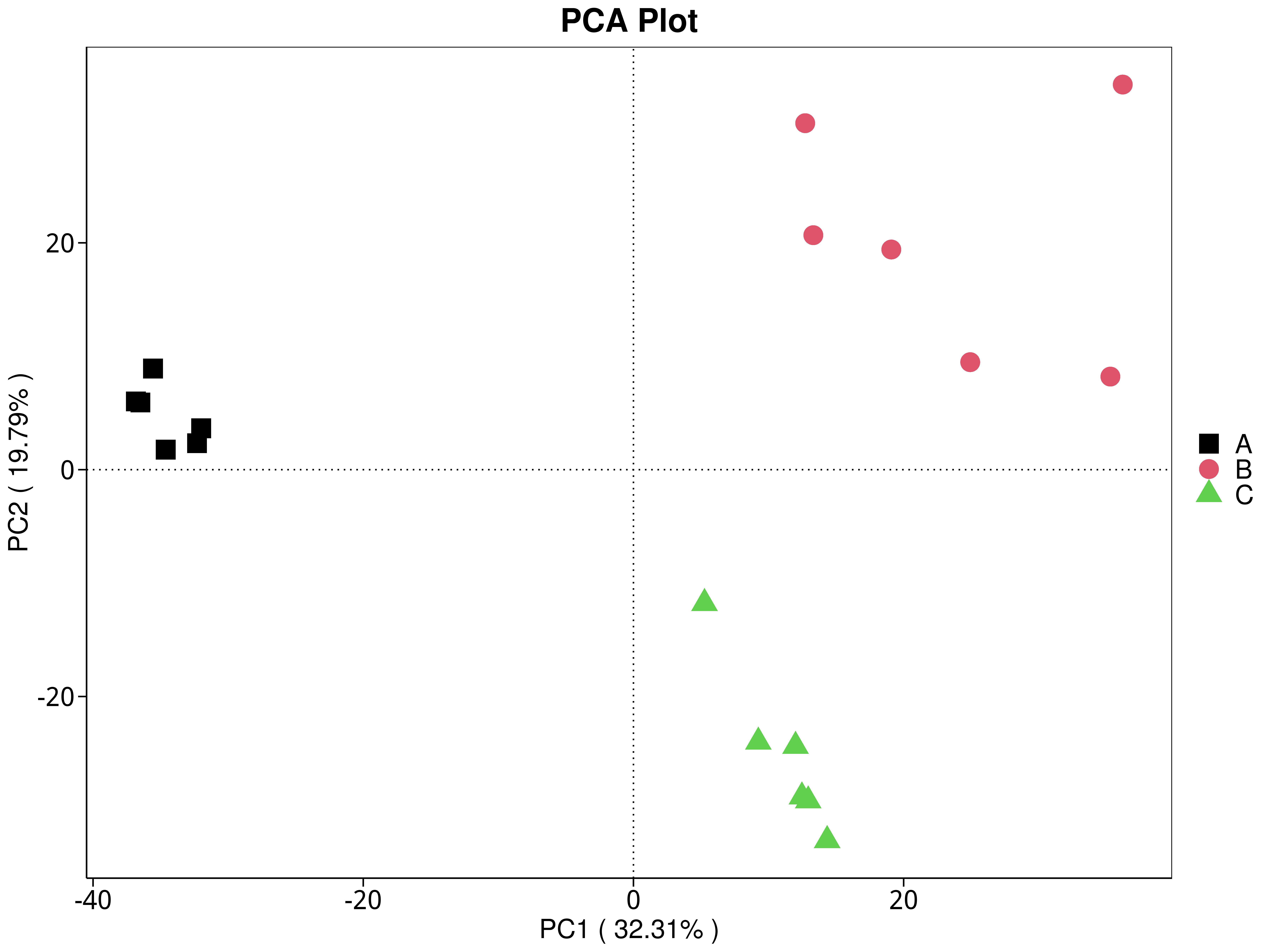
4.5.3.3 eggNOG
图4.30 基于 eggNOG 水平的 PCA 结果展示
说明:功能水平PCA分析,横坐标表示第一主成分,百分比则表示第一主成分对样品差异的贡献值;纵坐标表示第二主成分,百分比表示第二主成分对样品差异的贡献值;图中的每个点表示一个样品,同一个组的样品使用同一种颜色表示,没有分组默认按照样本出图
结果目录:
标注样品名的PCA图见 : result/05.Diversity/eggNOG/PCA_group1/*/PCA_lable.{png,pdf}
未标注样品名的PCA图见 : result/05.Diversity/eggNOG/PCA_group1/*/PCA.{png,pdf};
未标示样品名称的带聚类圈的PCA图见 : result/05.Diversity/eggNOG/PCA_group1/*/PCA_circle.{png,pdf}
标示样品名称的带聚类圈的PCA图见 : result/05.Diversity/eggNOG/PCA_group1/*/PCA_circleLable.{png,pdf}
各个主成分分析结果见 : result/05.Diversity/eggNOG/PCA_group1/*/PCA.xls
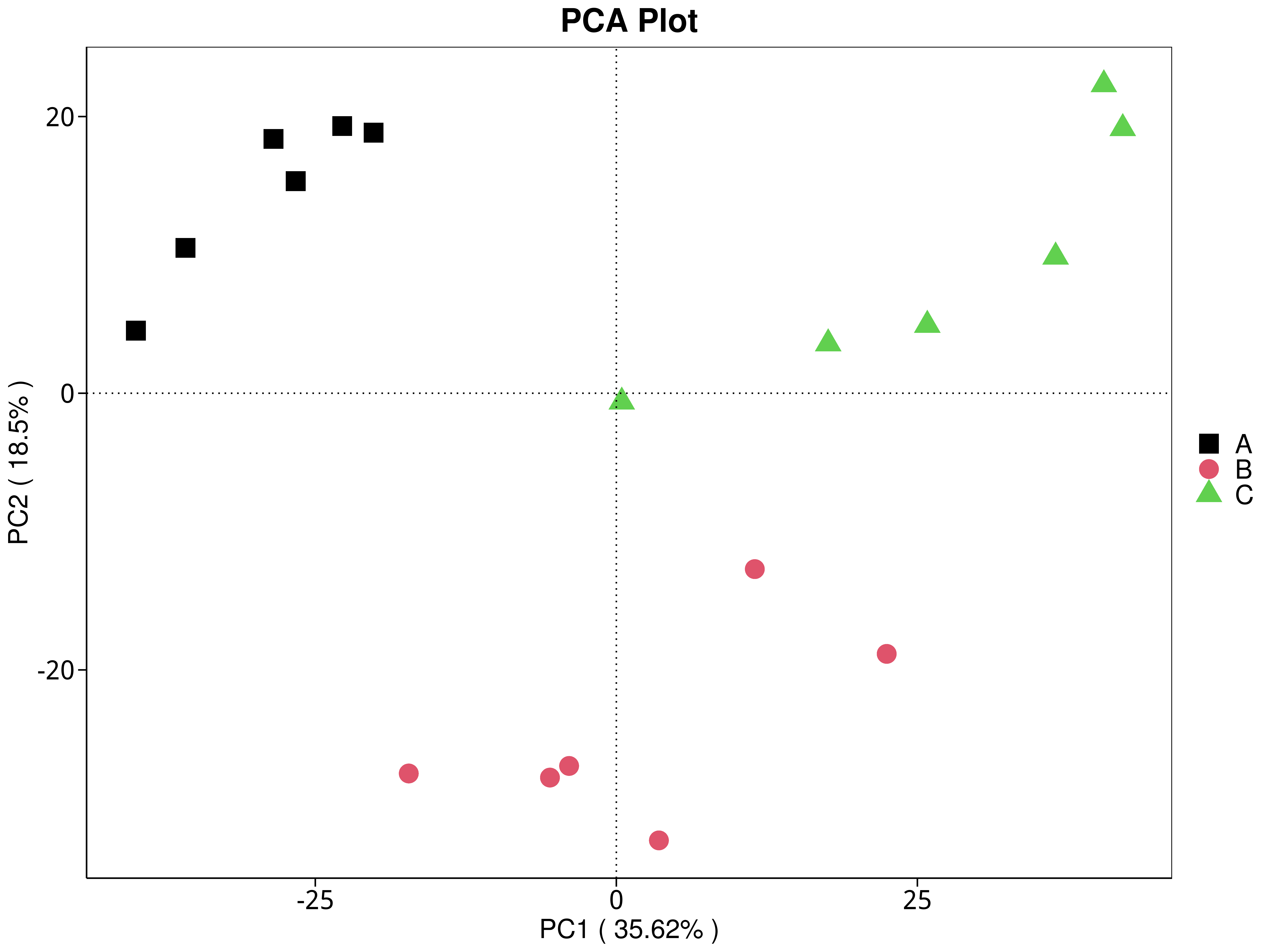
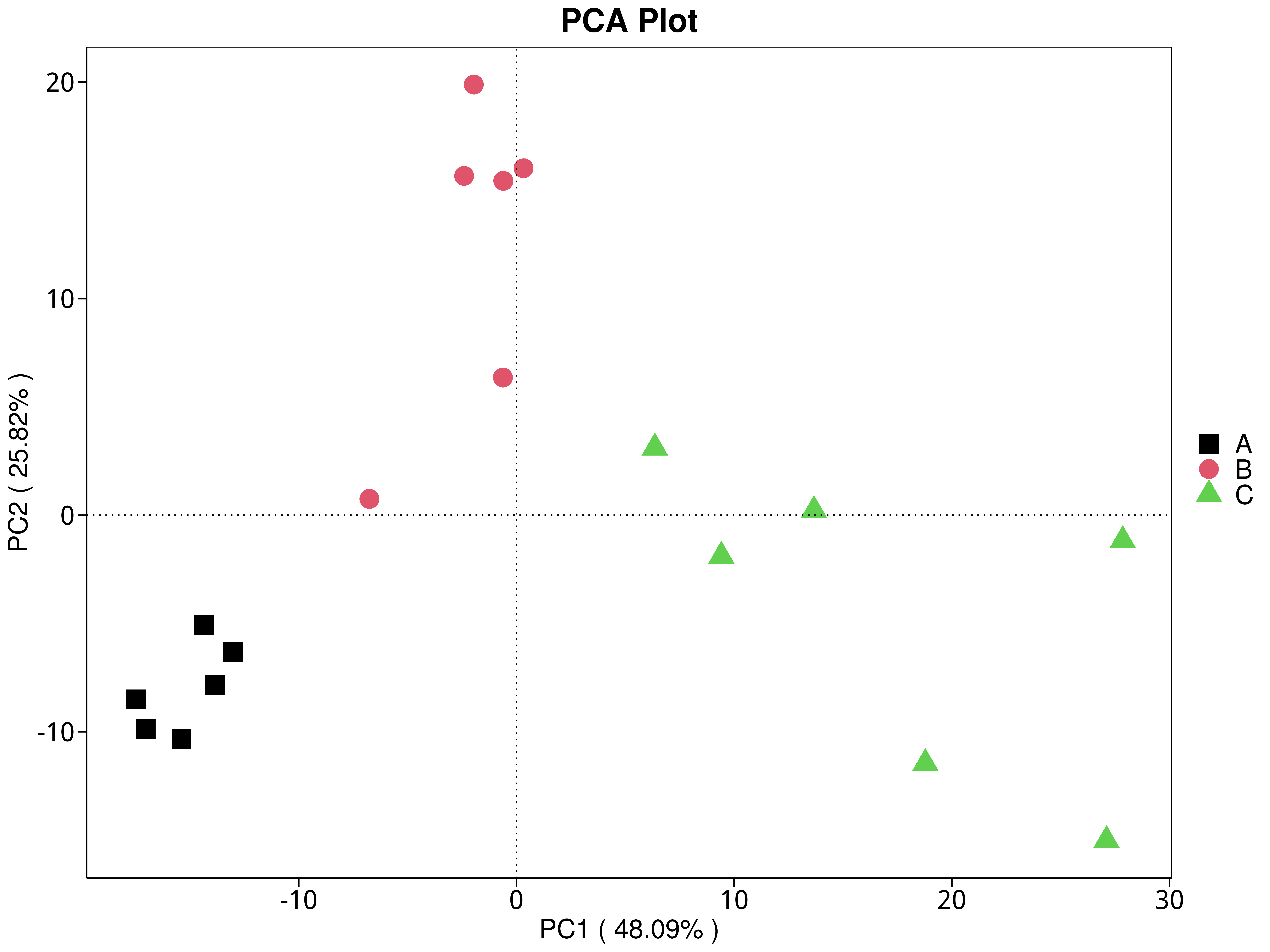
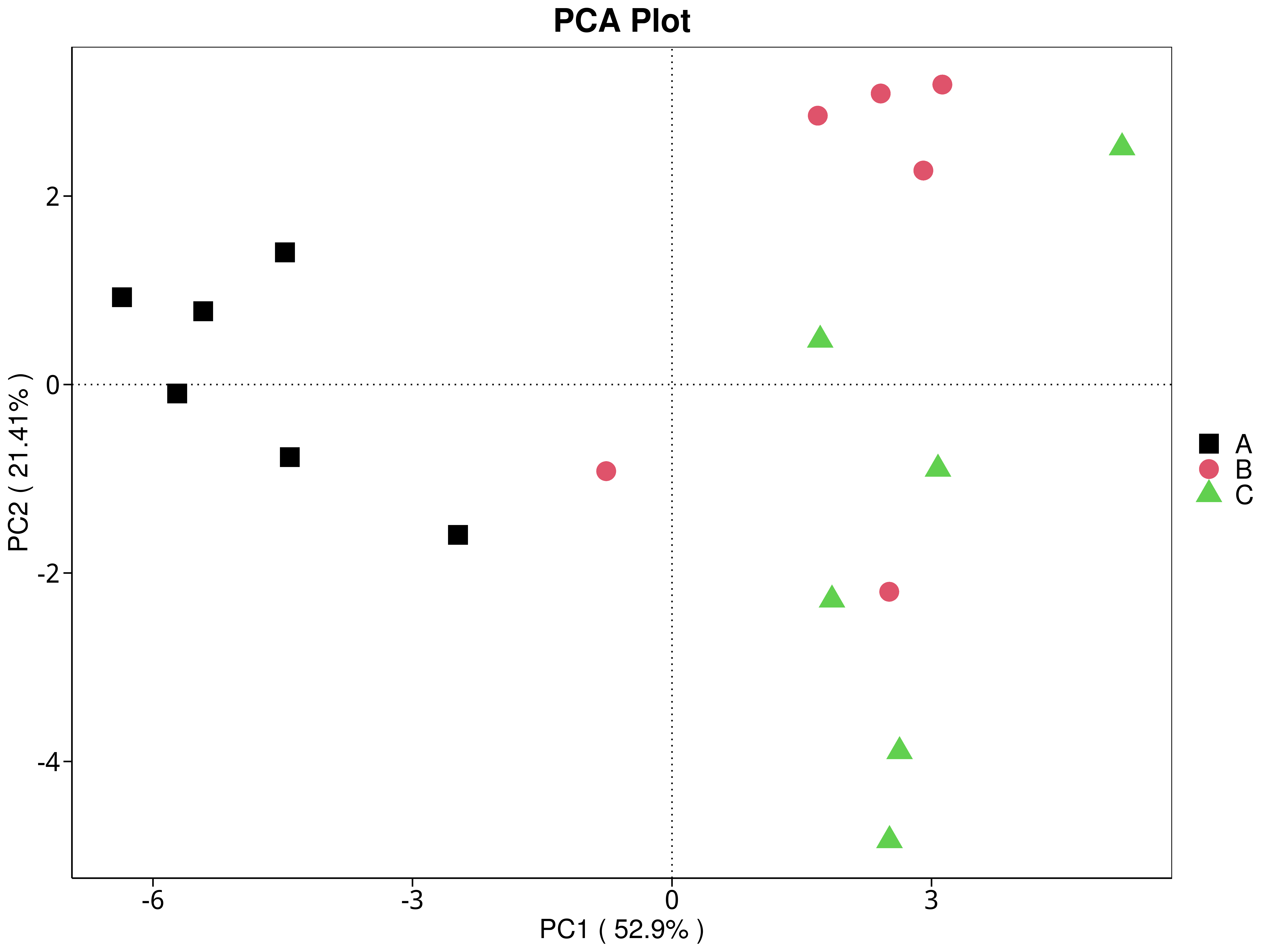
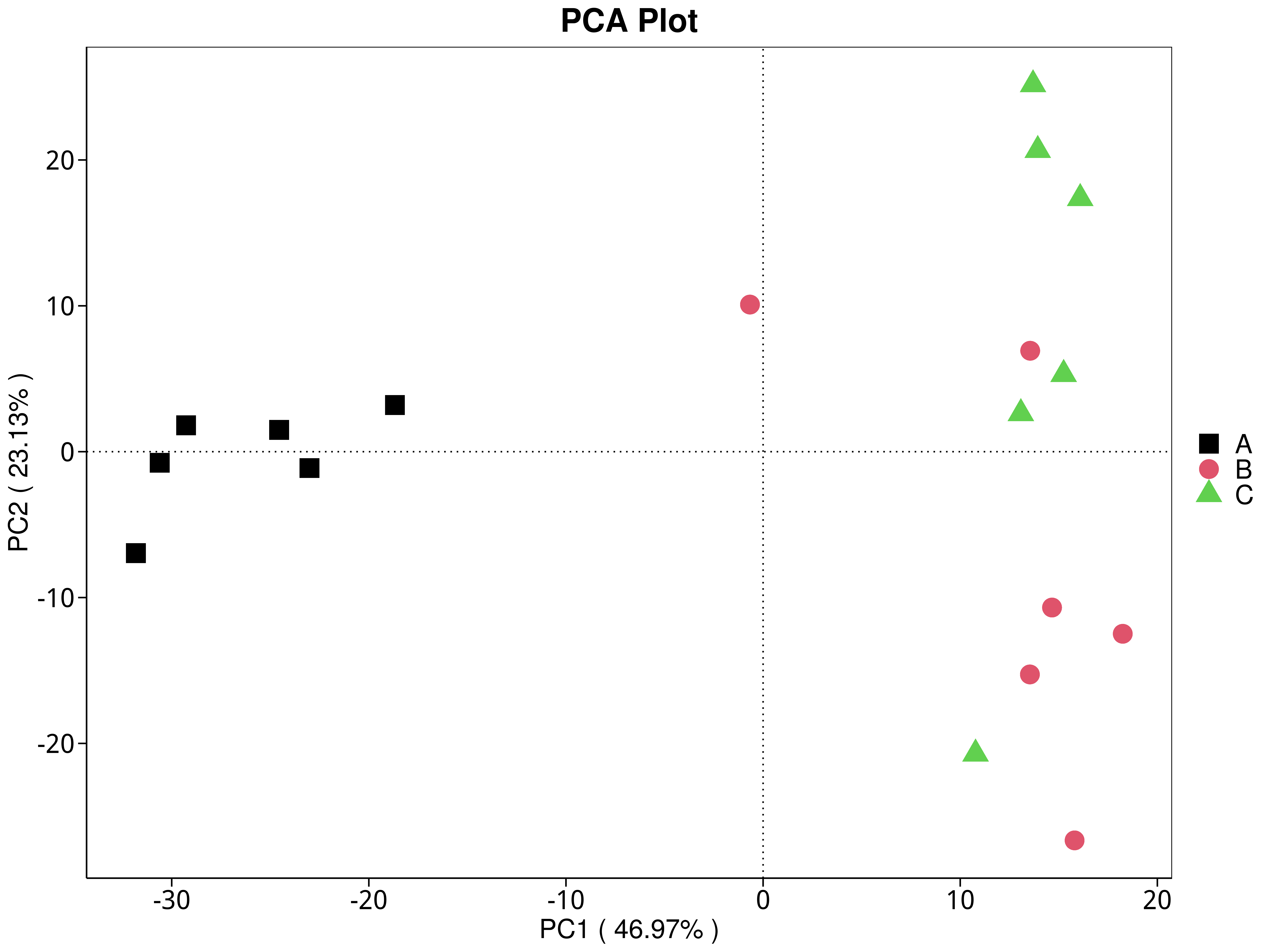
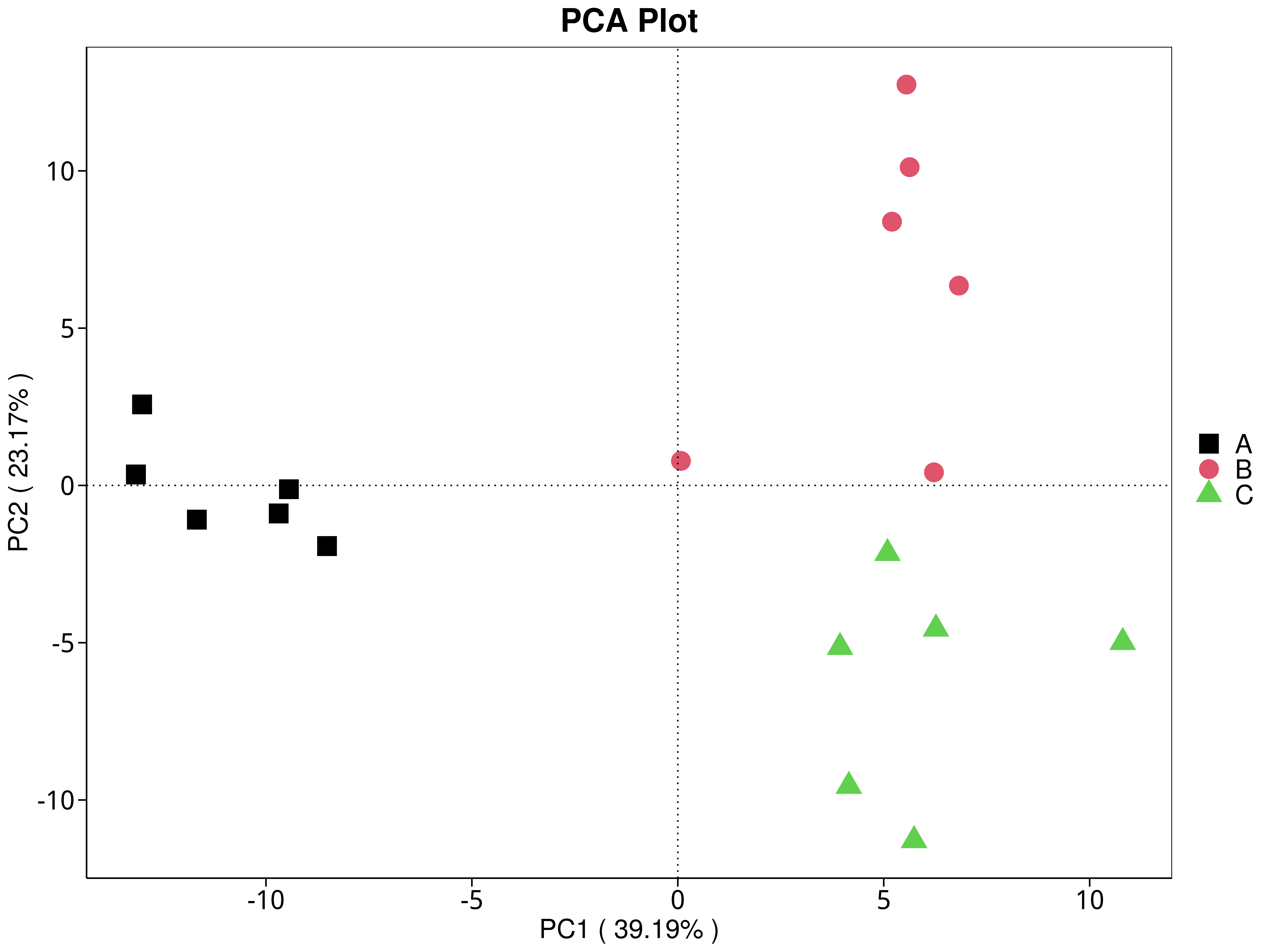
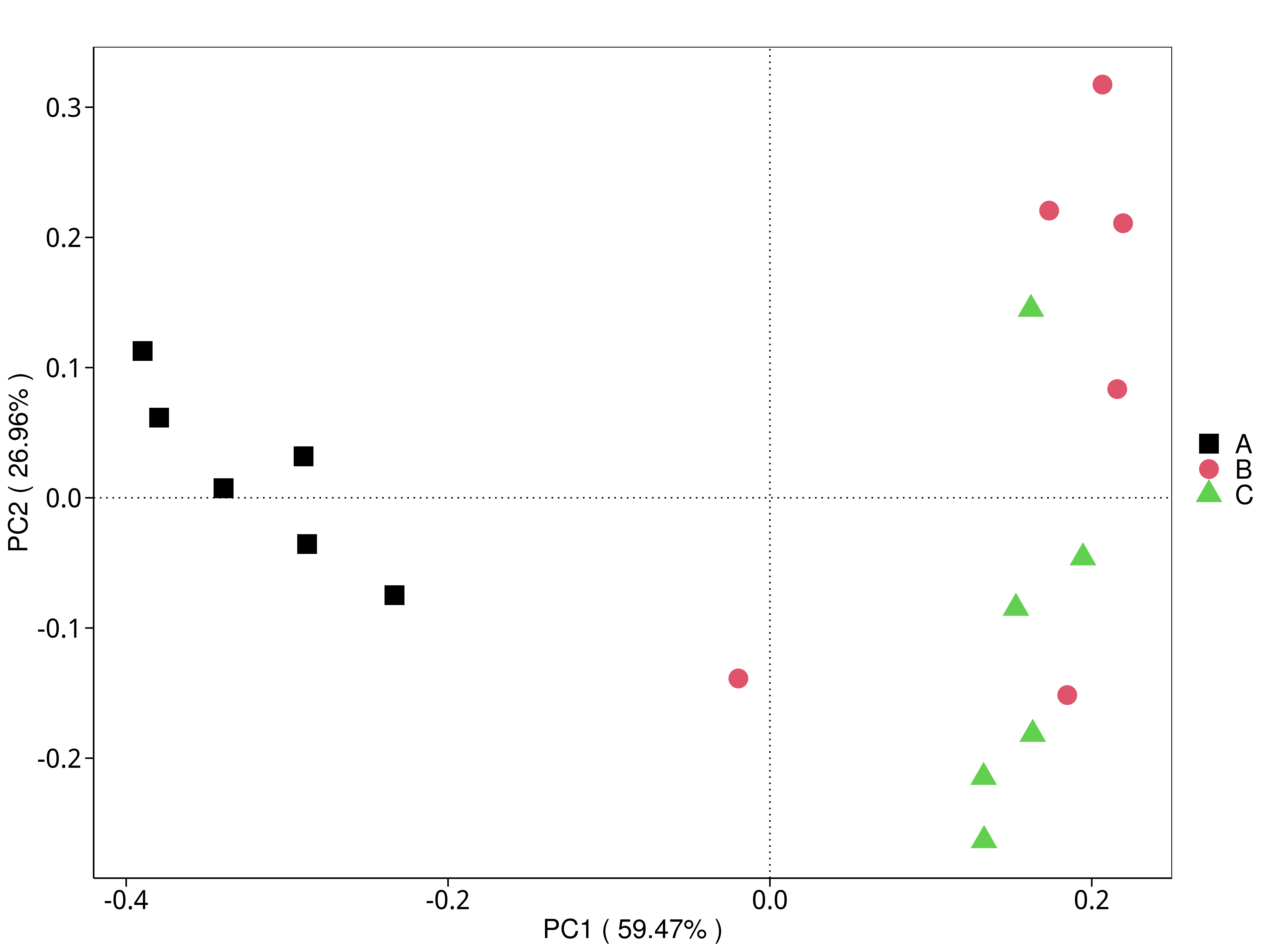
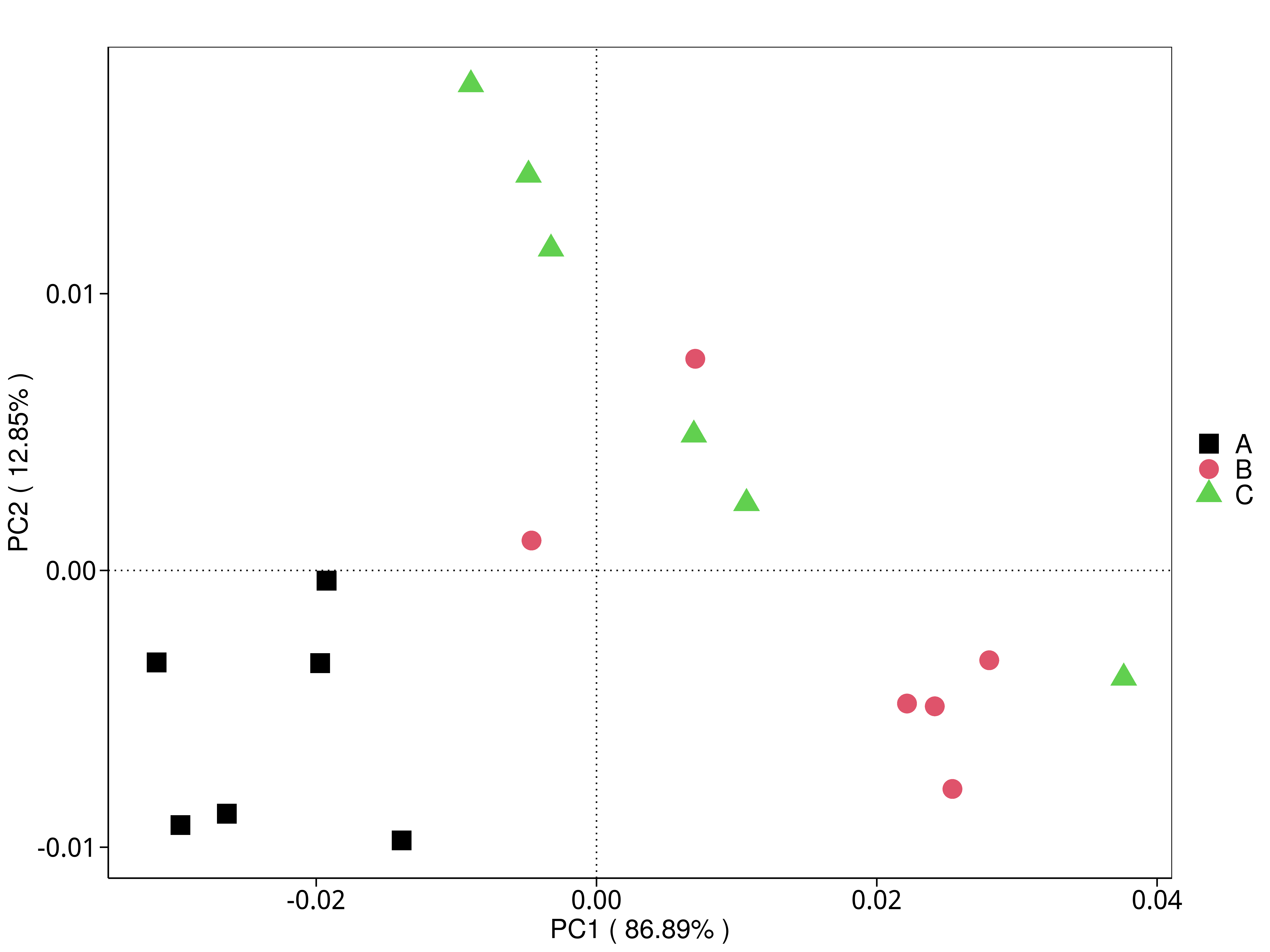
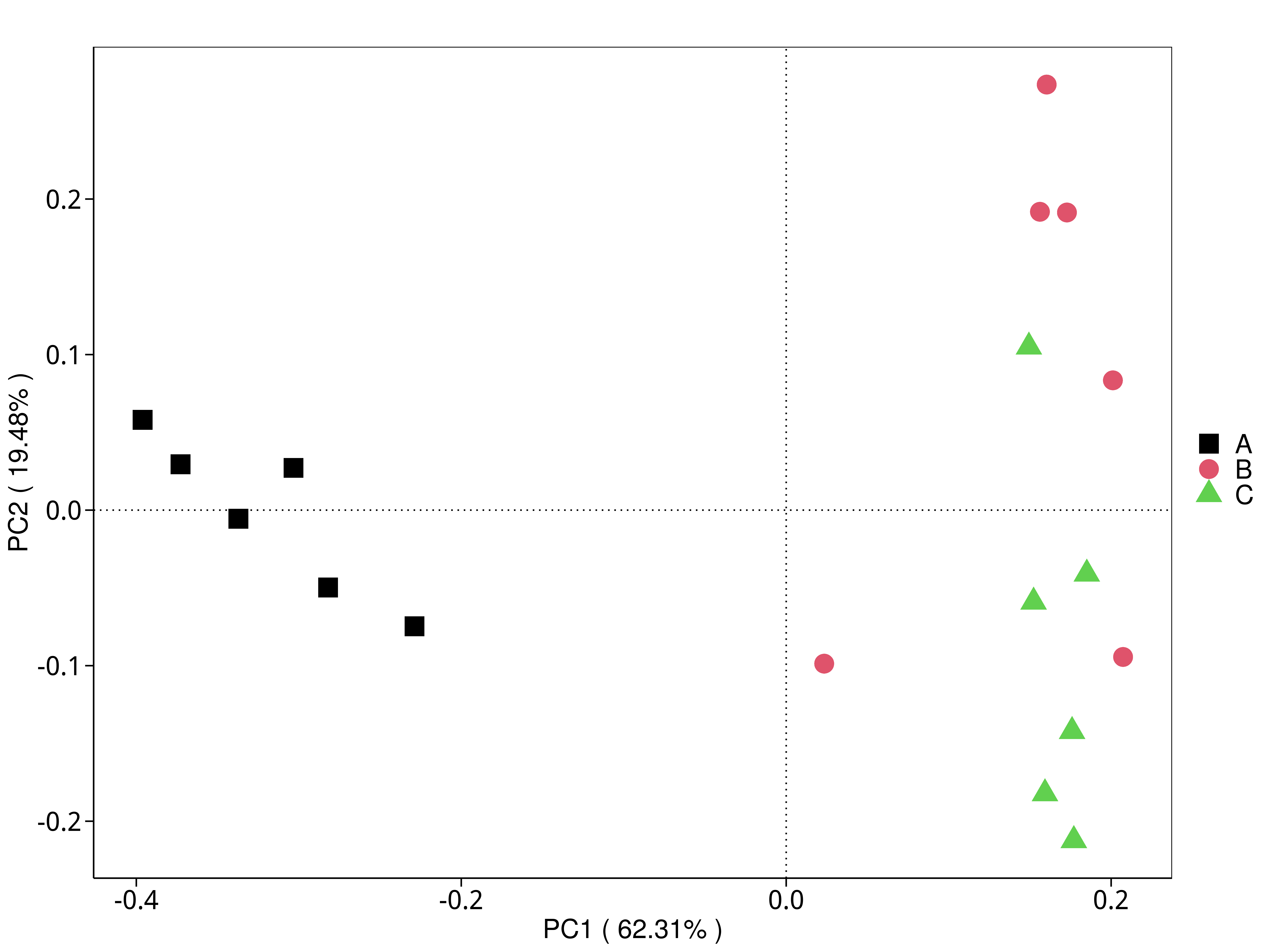
4.5.3.4 CAZy
图4.31 基于 CAZy 水平的 PCA 结果展示
说明:功能水平PCA分析,横坐标表示第一主成分,百分比则表示第一主成分对样品差异的贡献值;纵坐标表示第二主成分,百分比表示第二主成分对样品差异的贡献值;图中的每个点表示一个样品,同一个组的样品使用同一种颜色表示,没有分组默认按照样本出图
结果目录:
标注样品名的PCA图见 : result/05.Diversity/CAZy/PCA_group1/*/PCA_lable.{png,pdf}
未标注样品名的PCA图见 : result/05.Diversity/CAZy/PCA_group1/*/PCA.{png,pdf}
未标示样品名称的带聚类圈的PCA图见 : result/05.Diversity/CAZy/PCA_group1/*/PCA_circle.{png,pdf}
标示样品名称的带聚类圈的PCA图见 : result/05.Diversity/CAZy/PCA_group1/*/PCA_circleLable.{png,pdf}
各个主成分分析结果见 : result/05.Diversity/CAZy/PCA_group1/*/PCA.xls
4.5.3.5 VFDB
图4.32 基于 VFDB 水平的 PCA 结果展示
说明:功能水平PCA分析,横坐标表示第一主成分,百分比则表示第一主成分对样品差异的贡献值;纵坐标表示第二主成分,百分比表示第二主成分对样品差异的贡献值;图中的每个点表示一个样品,同一个组的样品使用同一种颜色表示,没有分组默认按照样本出图
结果目录:
标注样品名的PCA图见 : result/05.Diversity/VFDB/PCA_group1/*/PCA_lable.{png,pdf}
未标注样品名的PCA图见 : result/05.Diversity/VFDB/PCA_group1/*/PCA.{png,pdf}
未标示样品名称的带聚类圈的PCA图见 : result/05.Diversity/VFDB/PCA_group1/*/PCA_circle.{png,pdf}
标示样品名称的带聚类圈的PCA图见 : result/05.Diversity/VFDB/PCA_group1/*/PCA_circleLable.{png,pdf}
各个主成分分析结果见 : result/05.Diversity/VFDB/PCA_group1/*/PCA.xls
4.5.3.6 PHI
图4.33 基于 PHI 水平的 PCA 结果展示
说明:功能水平PCA分析,横坐标表示第一主成分,百分比则表示第一主成分对样品差异的贡献值;纵坐标表示第二主成分,百分比表示第二主成分对样品差异的贡献值;图中的每个点表示一个样品,同一个组的样品使用同一种颜色表示,没有分组默认按照样本出图
结果目录:
标注样品名的PCA图见 : result/05.Diversity/PHI/PCA_group1/*/PCA_lable.{png,pdf}
未标注样品名的PCA图见 : result/05.Diversity/PHI/PCA_group1/*/PCA.{png,pdf}
未标示样品名称的带聚类圈的PCA图见 : result/05.Diversity/PHI/PCA_group1/*/PCA_circle.{png,pdf}
标示样品名称的带聚类圈的PCA图见 : result/05.Diversity/PHI/PCA_group1/*/PCA_circleLable.{png,pdf}
各个主成分分析结果见 : result/05.Diversity/PHI/PCA_group1/*/PCA.xls
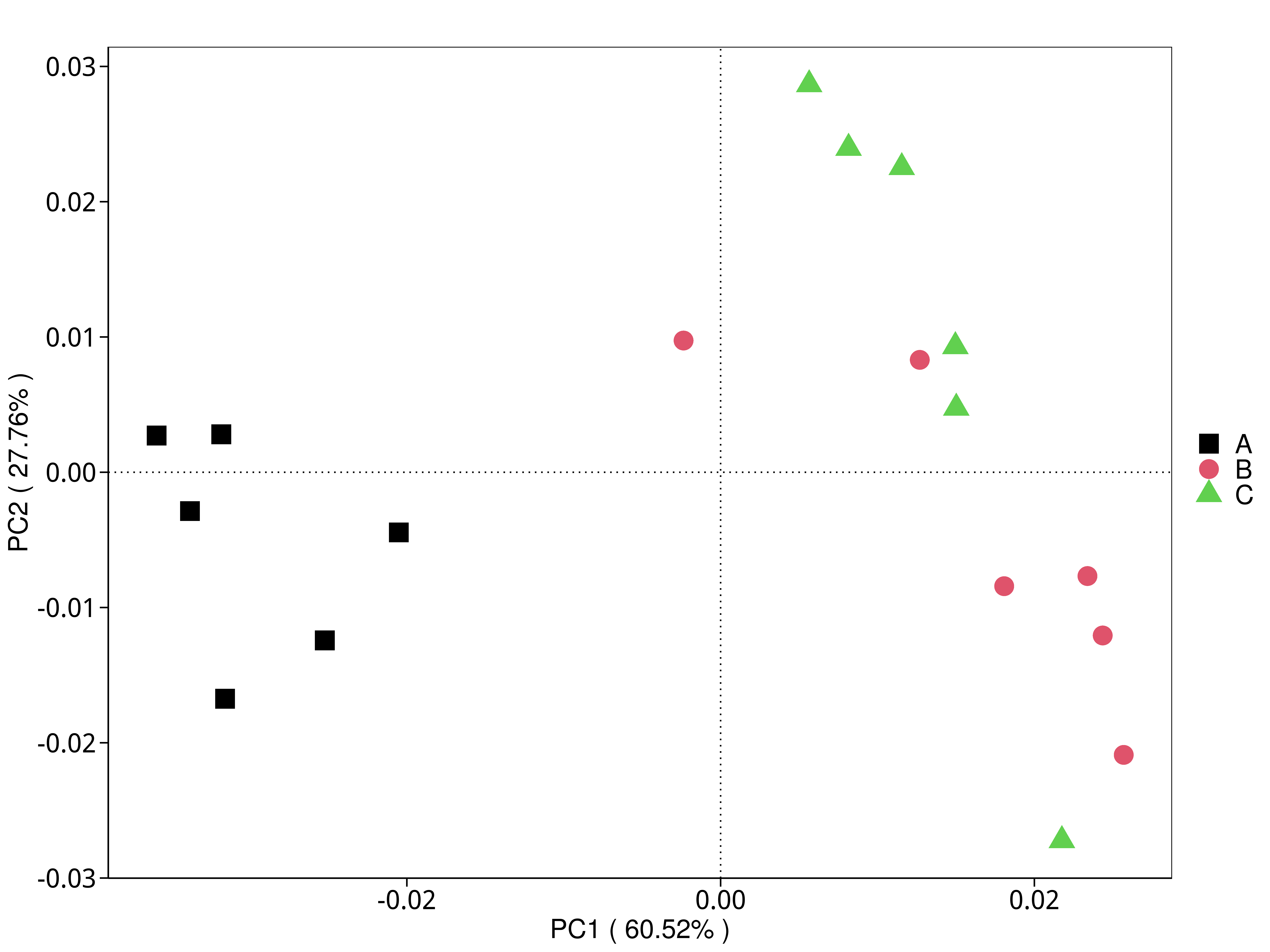
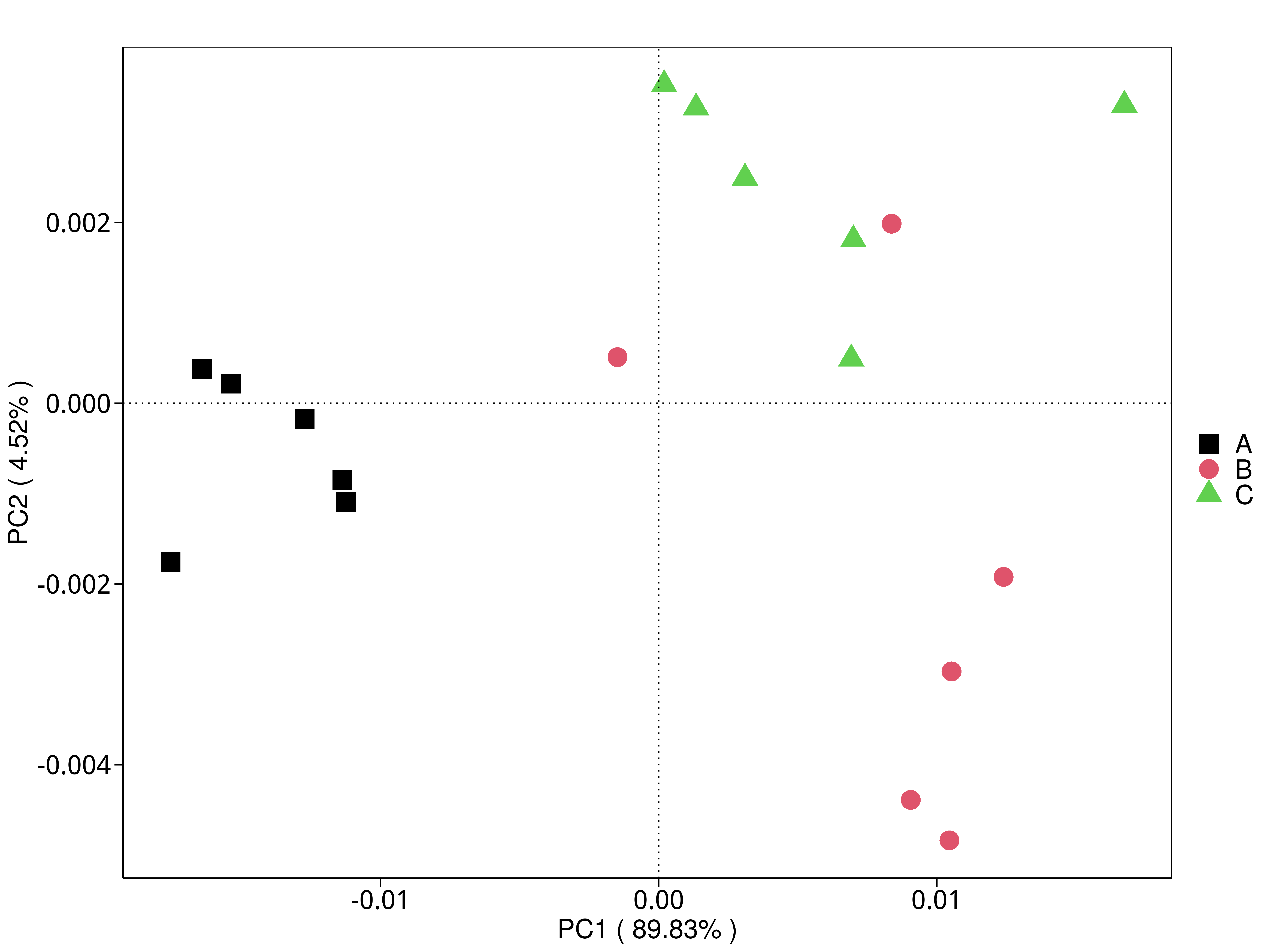
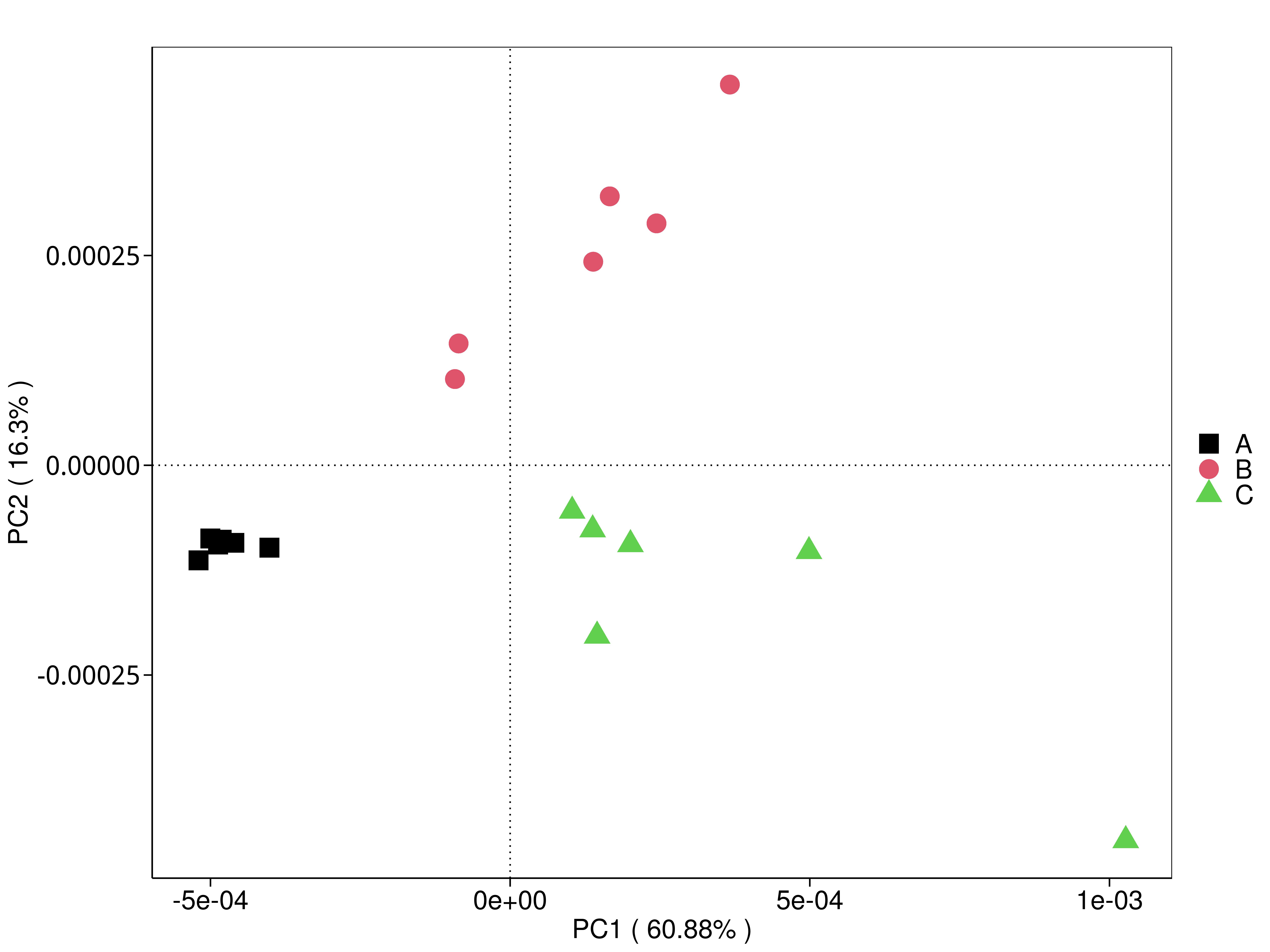
4.5.4 主坐标(PCoA)分析
主坐标分析(PCoA,Principal Coordinates Analysis),是通过一系列的特征值和特征向量排序从多维数据中提取出最主要的元素和结构。我们基于Bray-Curtis 距离来进行PCoA分析,并选取贡献率最大的主坐标组合进行作图展示。如果样品距离越接近,表示物种或功能组成结构越相似,因此群落结构相似度高的样品倾向于聚集在一起,群落差异很大的样品则会远远分开。 基于不同分类层级的物种或功能丰度表得到Bray-Curtis 距离矩阵,我们进行了 PCoA分析。
4.5.4.1 Micro_NR
图4.34 基于 Micro_NR 水平的 PCoA 结果展示
说明:PCoA分析,横坐标表示一个主坐标,纵坐标表示另一个主坐标,百分比表示主坐标对样本差异的贡献值;图中的每个点表示一个样本,同一个组的样本使用同一种颜色表示
结果目录:
标注样品名的PCoA图见 : result/05.Diversity/MicroNR/PCoA_group1/*/PCoA_lable.{png,pdf}
未标注样品名的PCoA图见 : result/05.Diversity/MicroNR/PCoA_group1/*/PCoA.{png,pdf}
未标示样品名称的带聚类圈的PCoA图见 : result/05.Diversity/MicroNR/PCoA_group1/*/PCoA_circleLable.{png,pdf}
标示样品名称的带聚类圈的PCoA图见 : result/05.Diversity/MicroNR/PCoA_group1/*/PCoA_circle.{png,pdf}
各个主坐标分析结果见 : result/05.Diversity/MicroNR/PCoA_group1/*/PCoA.xls
4.5.4.2 KEGG
图4.35 基于 KEGG 水平的 PCoA 结果展示
说明:功能水平PCoA分析,横坐标表示一个主坐标,纵坐标表示另一个主坐标,百分比表示主坐标对样本差异的贡献值;图中的每个点表示一个样本,同一个组的样本使用同一种颜色表示
结果目录:
标注样品名的PCoA图见 : result/05.Diversity/KEGG/PCoA_group1/*/PCoA_lable.{png,pdf}
未标注样品名的PCoA图见 : result/05.Diversity/KEGG/PCoA_group1/*/PCoA.{png,pdf}
未标示样品名称的带聚类圈的PCoA图见 : result/05.Diversity/KEGG/PCoA_group1/*/PCoA_circleLable.{png,pdf}
标示样品名称的带聚类圈的PCoA图见 : result/05.Diversity/KEGG/PCoA_group1/*/PCoA_circle.{png,pdf}
各个主坐标分析结果见 : result/05.Diversity/KEGG/PCoA_group1/*/PCoA.xls
4.5.4.3 eggNOG
图4.36 基于 eggNOG 水平的 PCoA 结果展示
说明:功能水平PCoA分析,横坐标表示一个主坐标,纵坐标表示另一个主坐标,百分比表示主坐标对样本差异的贡献值;图中的每个点表示一个样本,同一个组的样本使用同一种颜色表示
结果目录:
标注样品名的PCoA图见 : result/05.Diversity/eggNOG/PCoA_group1/*/PCoA_lable.{png,pdf}
未标注样品名的PCoA图见 : result/05.Diversity/eggNOG/PCoA_group1/*/PCoA.{png,pdf}
未标示样品名称的带聚类圈的PCoA图见 : result/05.Diversity/eggNOG/PCoA_group1/*/PCoA_circleLable.{png,pdf}
标示样品名称的带聚类圈的PCoA图见 : result/05.Diversity/eggNOG/PCoA_group1/*/PCoA_circle.{png,pdf}
各个主坐标分析结果见 : result/05.Diversity/eggNOG/PCoA_group1/*/PCoA.xls
4.5.4.4 CAZy
图4.37 基于 CAZy 水平的 PCoA 结果展示
说明:功能水平PCoA分析,横坐标表示一个主坐标,纵坐标表示另一个主坐标,百分比表示主坐标对样本差异的贡献值;图中的每个点表示一个样本,同一个组的样本使用同一种颜色表示
结果目录:
标注样品名的PCoA图见 : result/05.Diversity/CAZy/PCoA_group1/*/PCoA_lable.{png,pdf}
未标注样品名的PCoA图见 : result/05.Diversity/CAZy/PCoA_group1/*/PCoA.{png,pdf}
未标示样品名称的带聚类圈的PCoA图见 : result/05.Diversity/CAZy/PCoA_group1/*/PCoA_circleLable.{png,pdf}
标示样品名称的带聚类圈的PCoA图见 : result/05.Diversity/CAZy/PCoA_group1/*/PCoA_circle.{png,pdf}
各个主坐标分析结果见 : result/05.Diversity/CAZy/PCoA_group1/*/PCoA.xls
4.5.4.5 VFDB
图4.38 基于 VFDB 水平的 PCoA 结果展示
说明:功能水平PCoA分析,横坐标表示一个主坐标,纵坐标表示另一个主坐标,百分比表示主坐标对样本差异的贡献值;图中的每个点表示一个样本,同一个组的样本使用同一种颜色表示
结果目录:
标注样品名的PCoA图见 : result/05.Diversity/VFDB/PCoA_group1/*/PCoA_lable.{png,pdf}
未标注样品名的PCoA图见 : result/05.Diversity/VFDB/PCoA_group1/*/PCoA.{png,pdf}
未标示样品名称的带聚类圈的PCoA图见 : result/05.Diversity/VFDB/PCoA_group1/*/PCoA_circleLable.{png,pdf}
标示样品名称的带聚类圈的PCoA图见 : result/05.Diversity/VFDB/PCoA_group1/*/PCoA_circle.{png,pdf}
各个主坐标分析结果见 : result/05.Diversity/VFDB/PCoA_group1/*/PCoA.xls
4.5.4.6 PHI
图4.39 基于 PHI 水平的 PCoA 结果展示
说明:功能水平PCoA分析,横坐标表示一个主坐标,纵坐标表示另一个主坐标,百分比表示主坐标对样本差异的贡献值;图中的每个点表示一个样本,同一个组的样本使用同一种颜色表示
结果目录:
标注样品名的PCoA图见 : result/05.Diversity/PHI/PCoA_group1/*/PCoA_lable.{png,pdf}
未标注样品名的PCoA图见 : result/05.Diversity/PHI/PCoA_group1/*/PCoA.{png,pdf}
未标示样品名称的带聚类圈的PCoA图见 : result/05.Diversity/PHI/PCoA_group1/*/PCoA_circleLable.{png,pdf}
标示样品名称的带聚类圈的PCoA图见 : result/05.Diversity/PHI/PCoA_group1/*/PCoA_circle.{png,pdf}
各个主坐标分析结果见 : result/05.Diversity/PHI/PCoA_group1/*/PCoA.xls
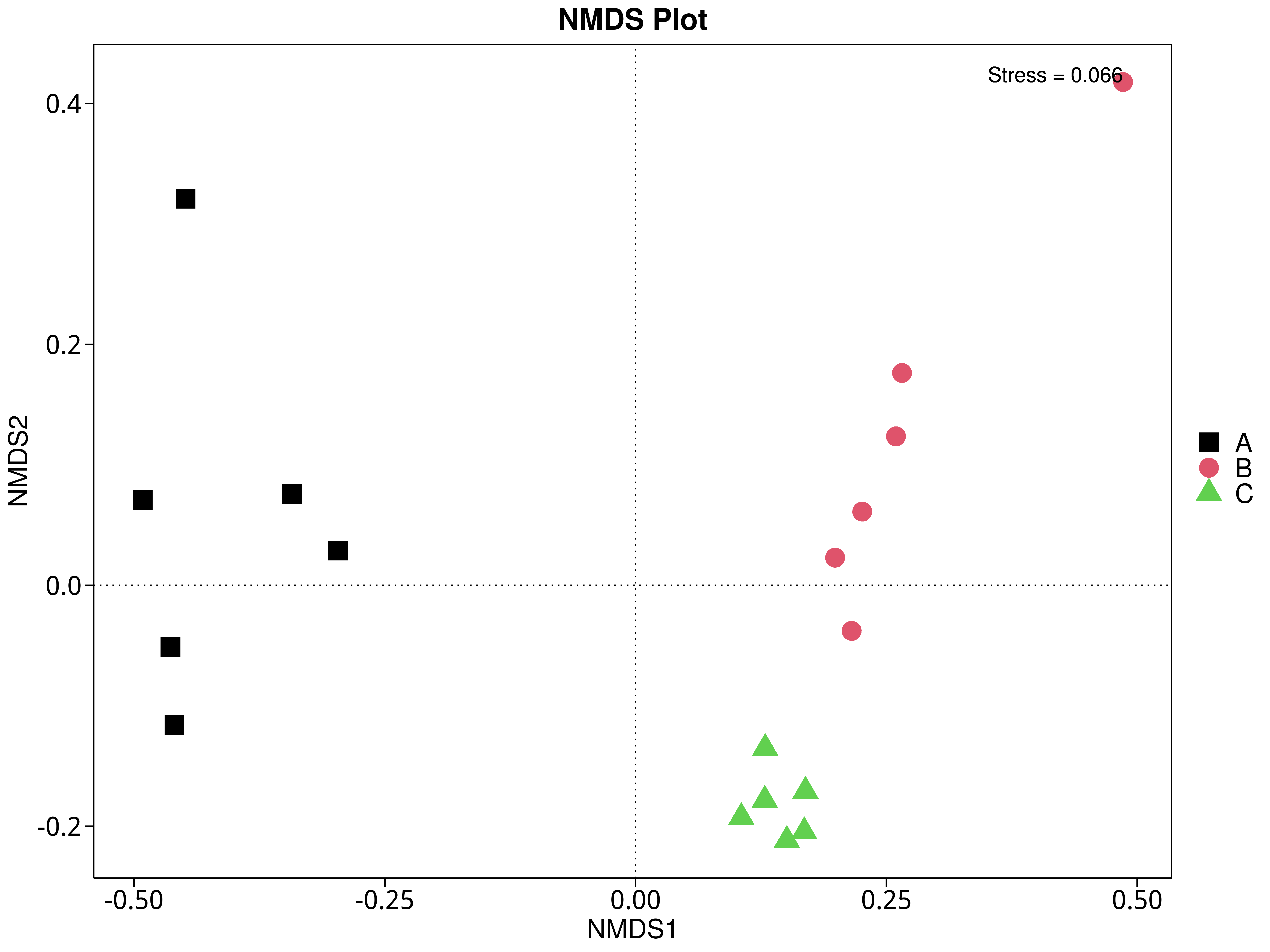
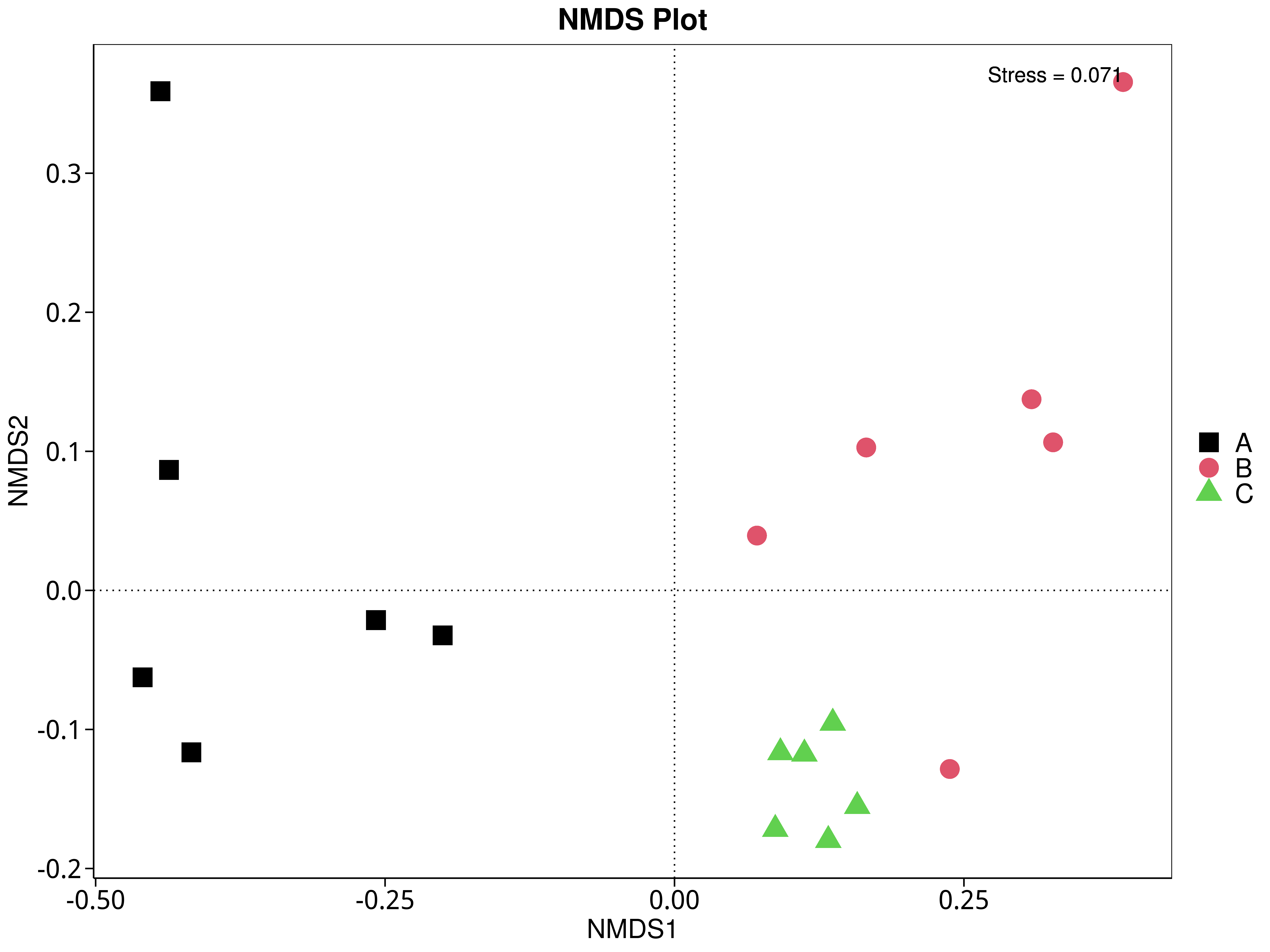
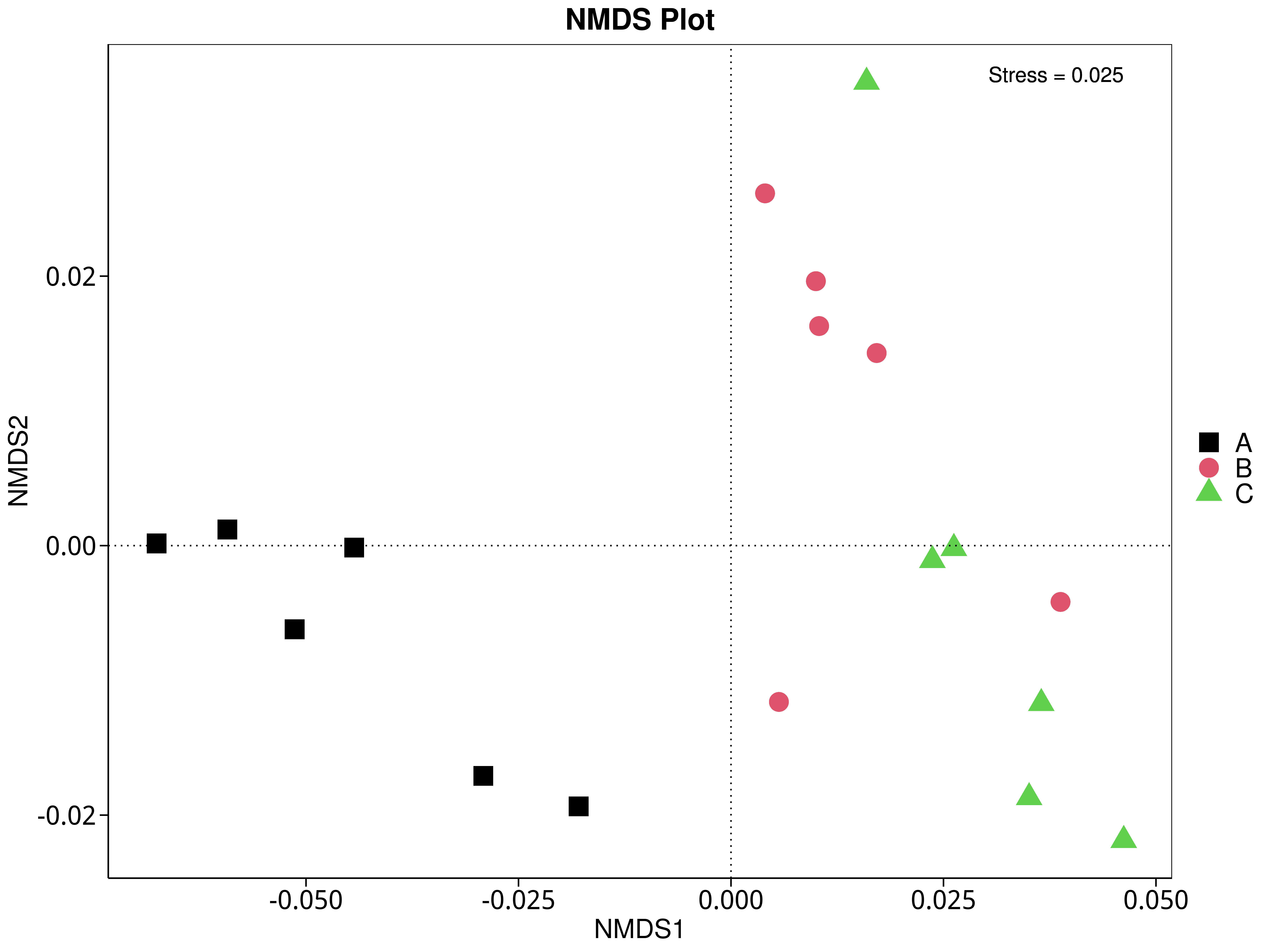
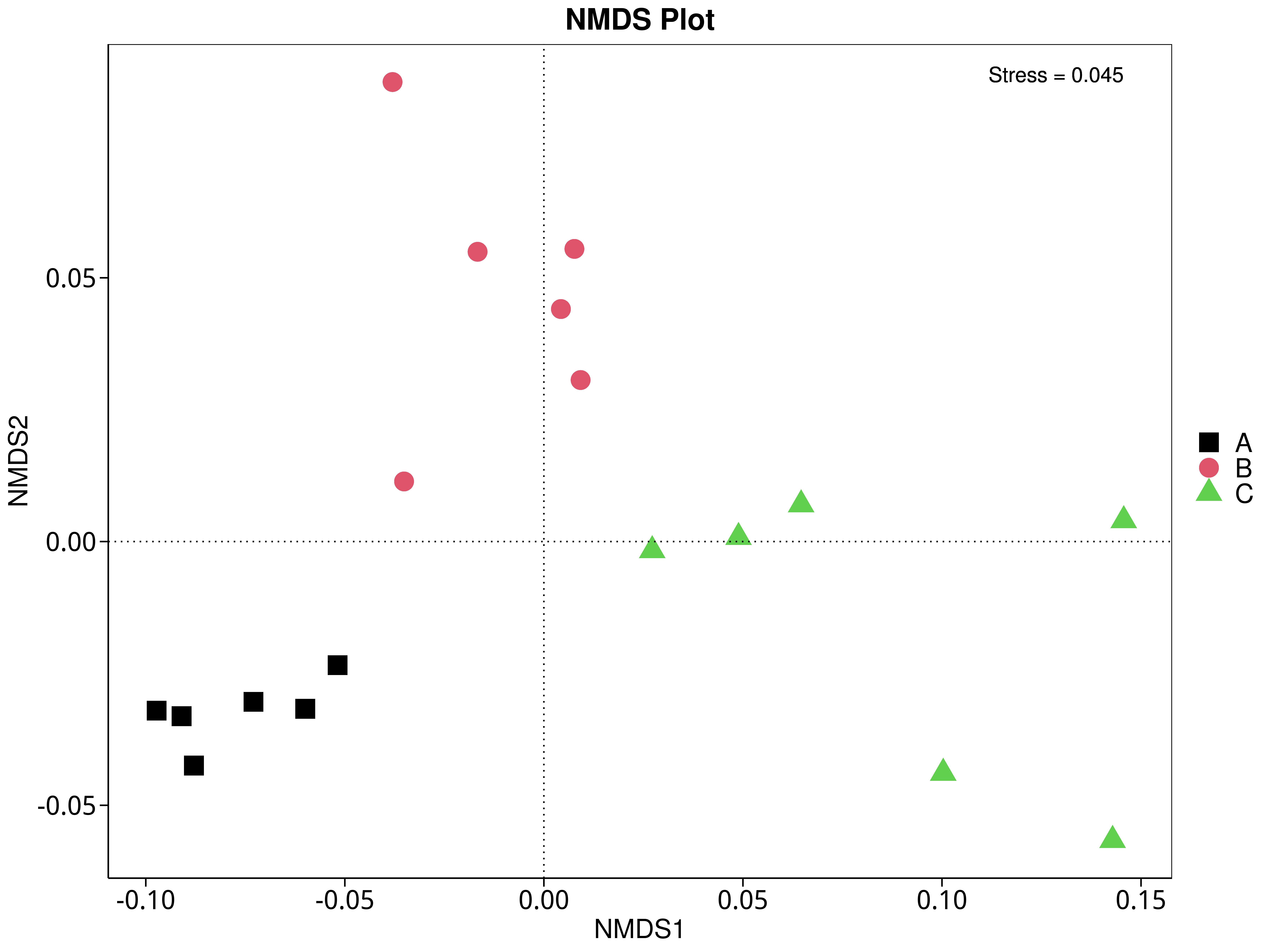
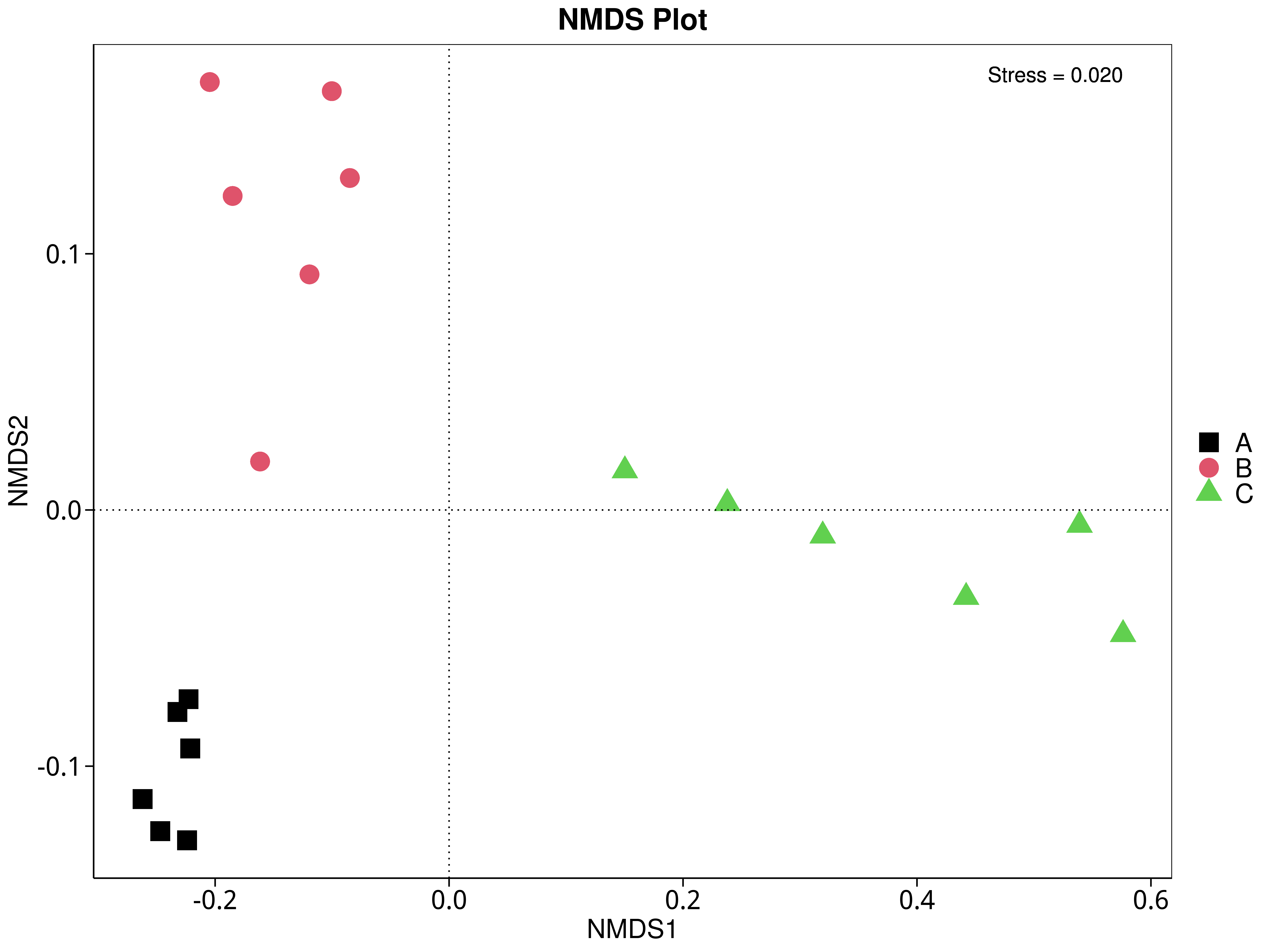
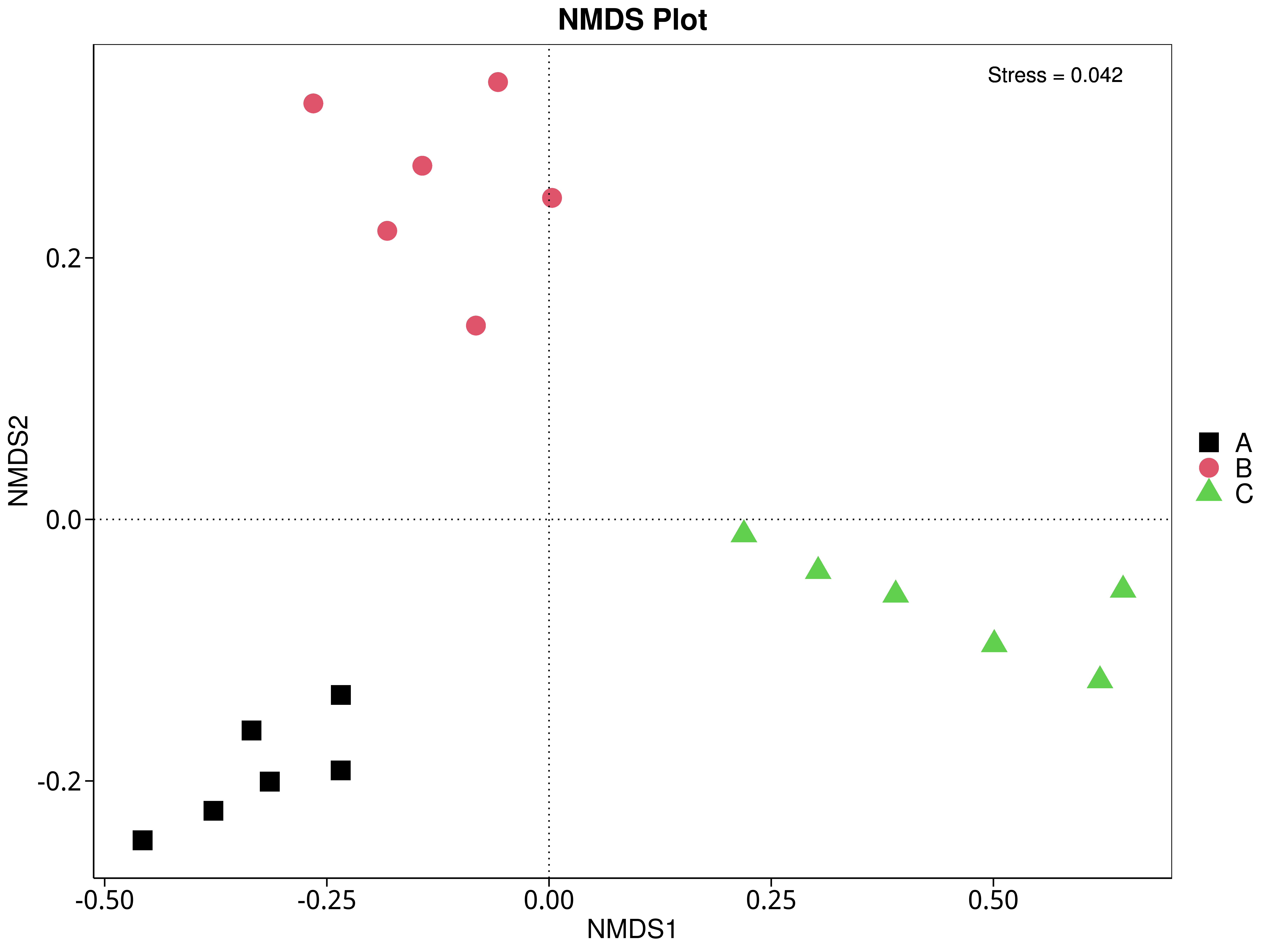
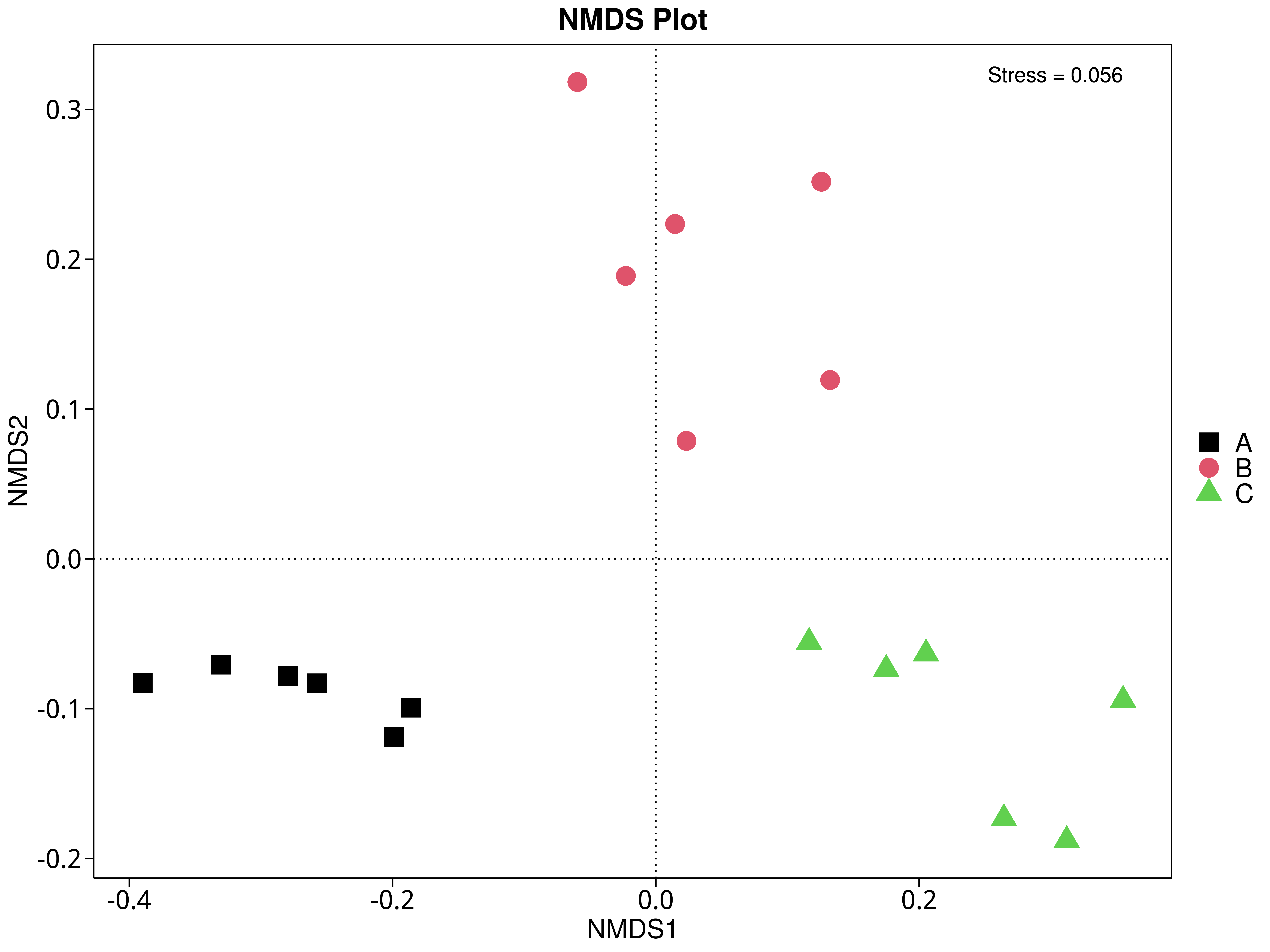
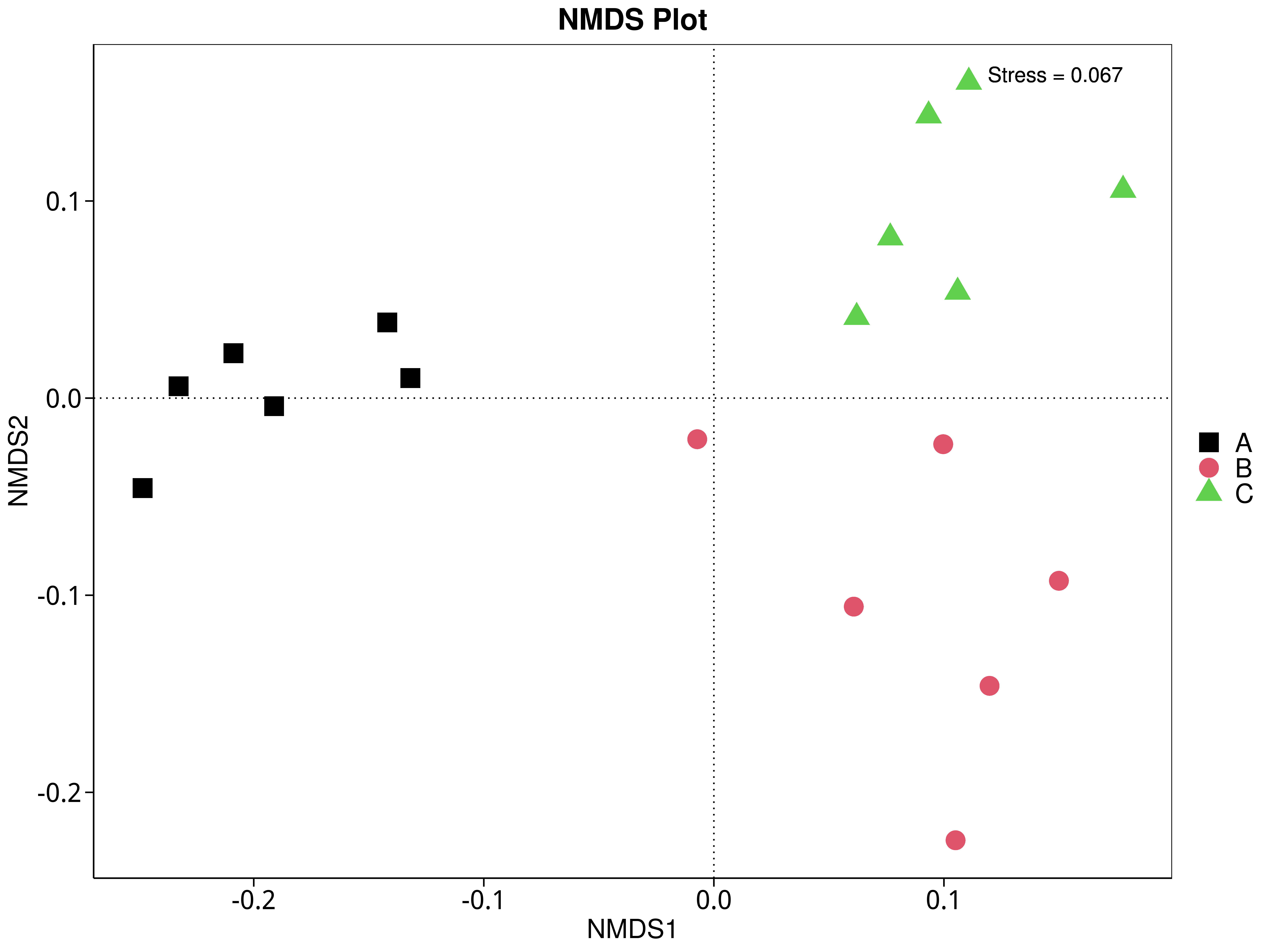
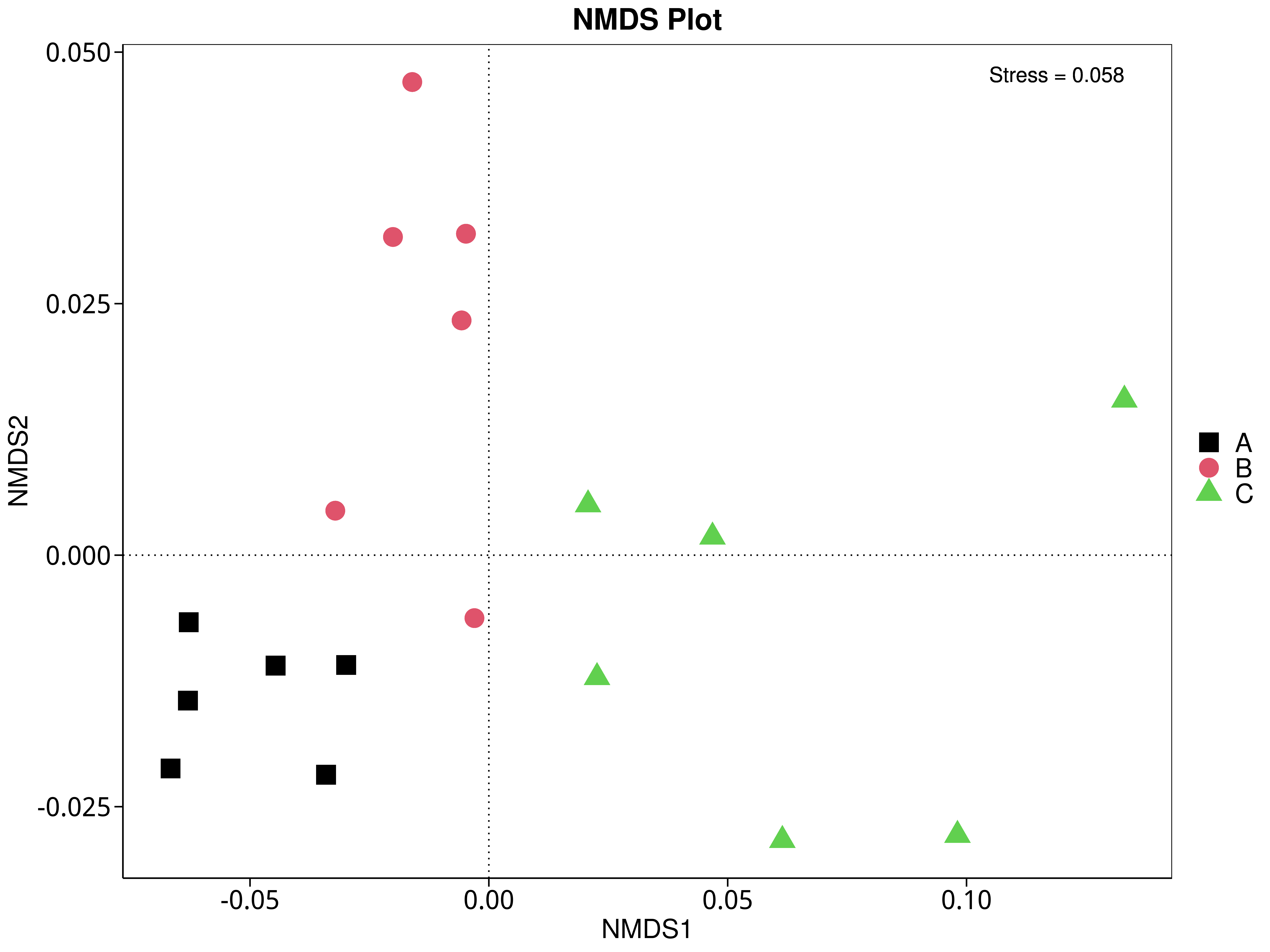
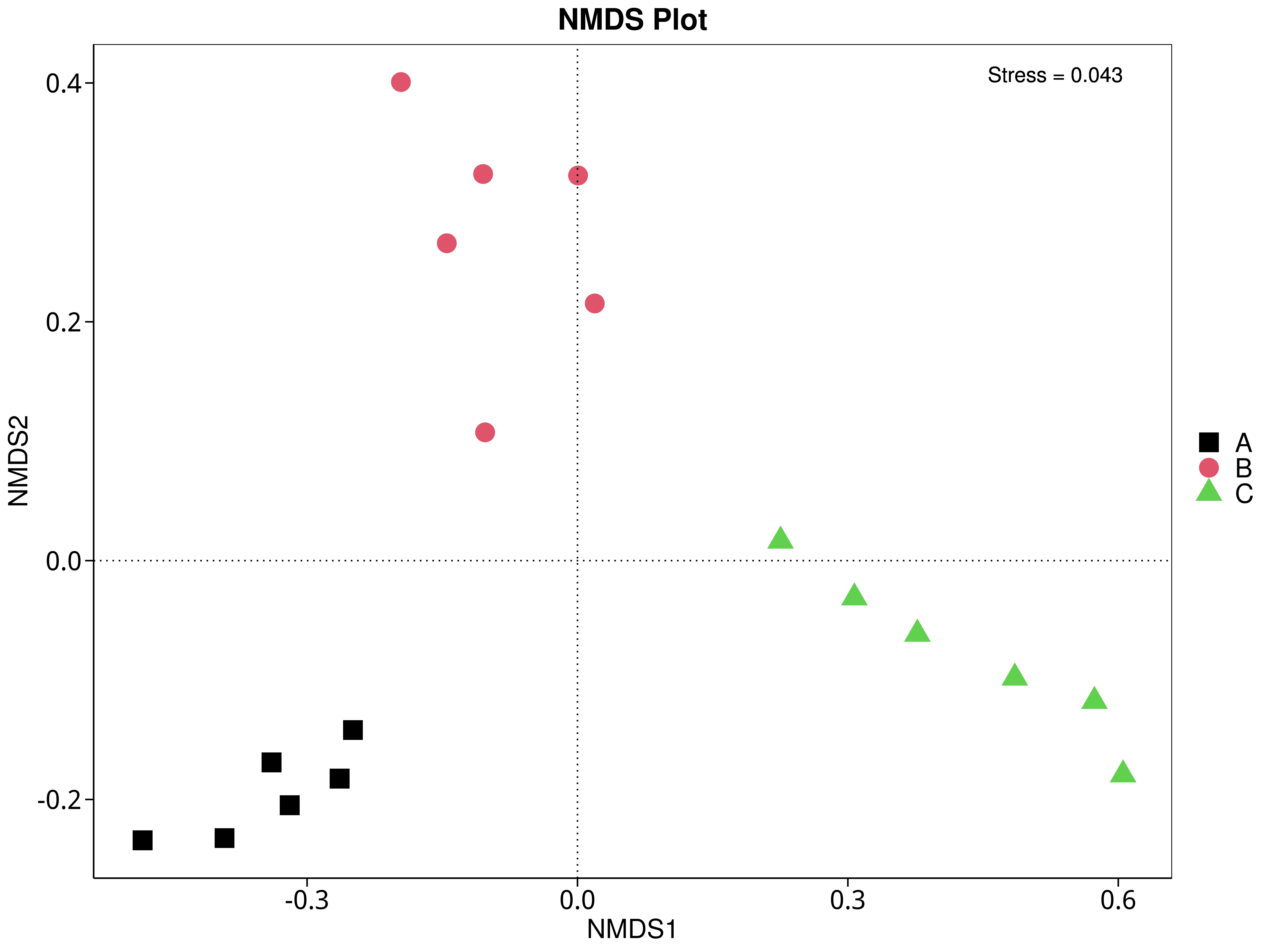
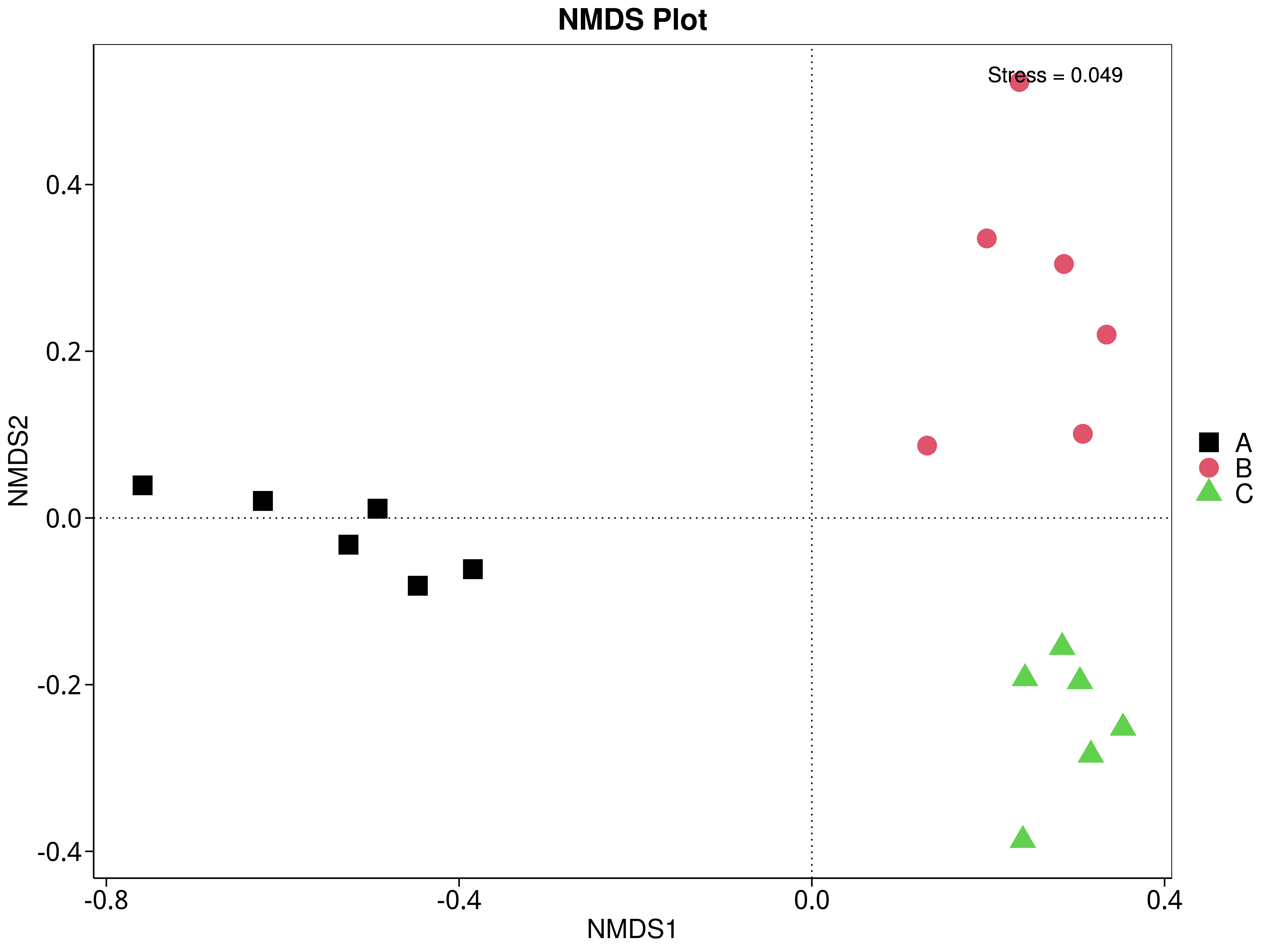
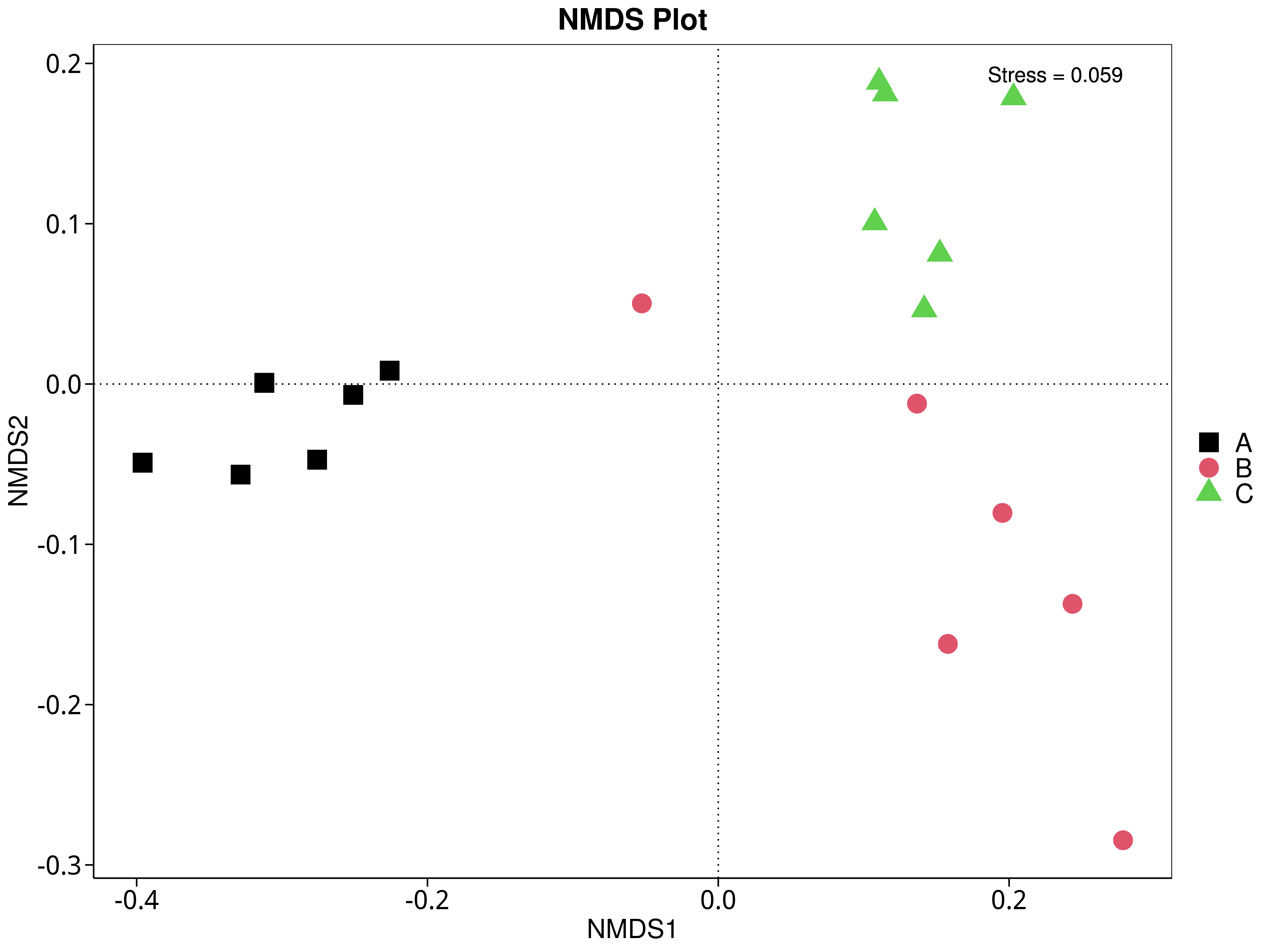
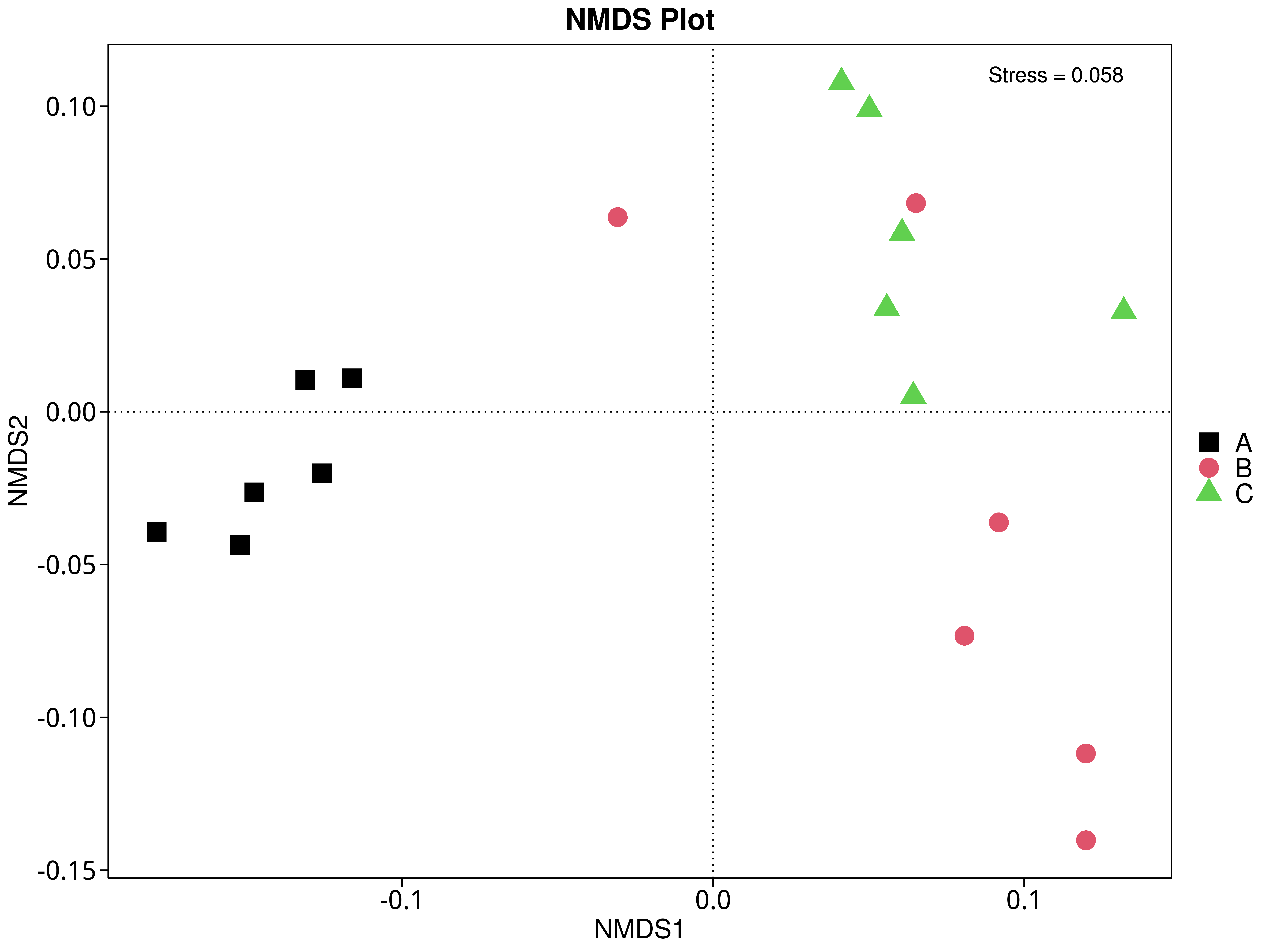
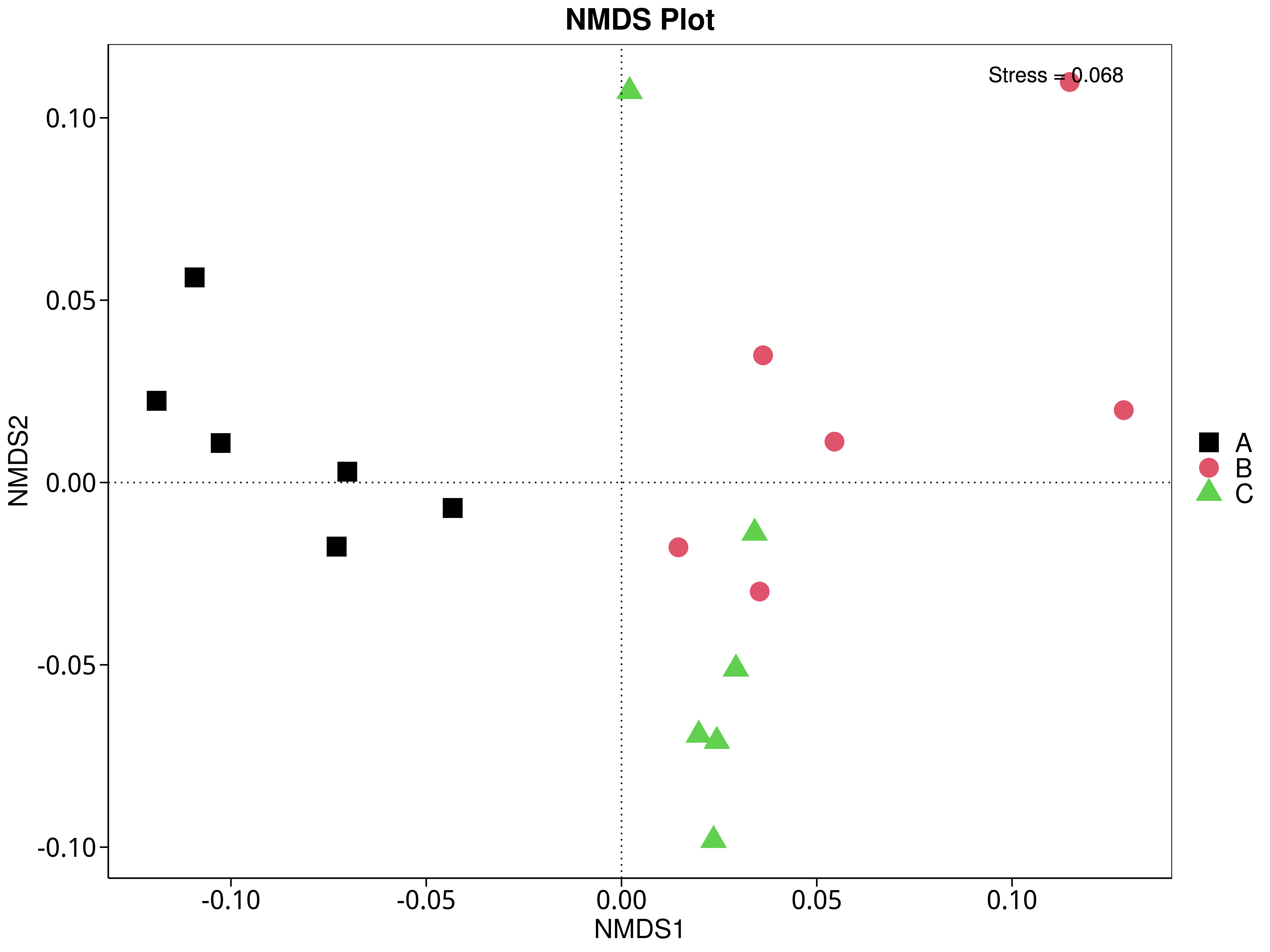
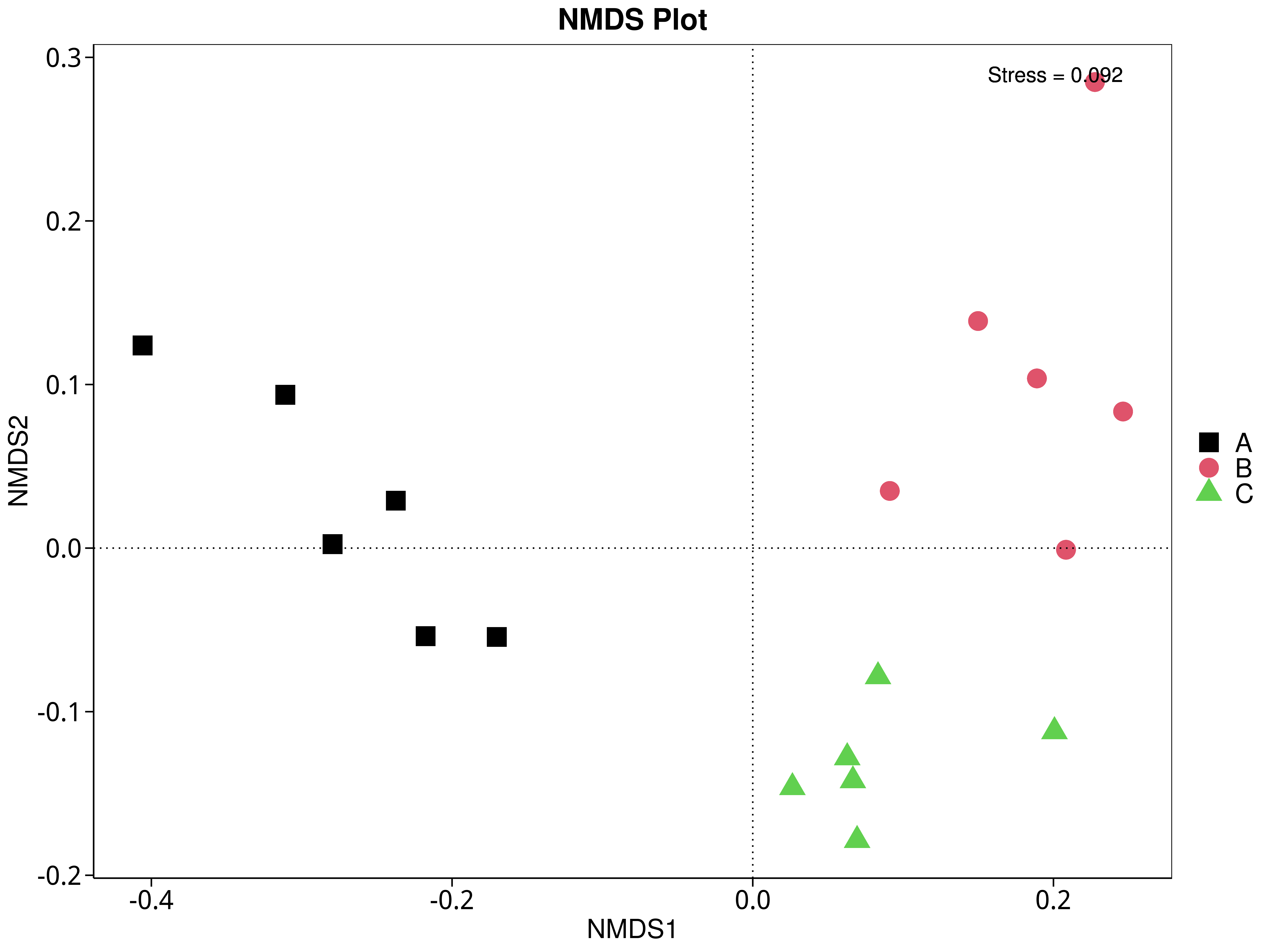
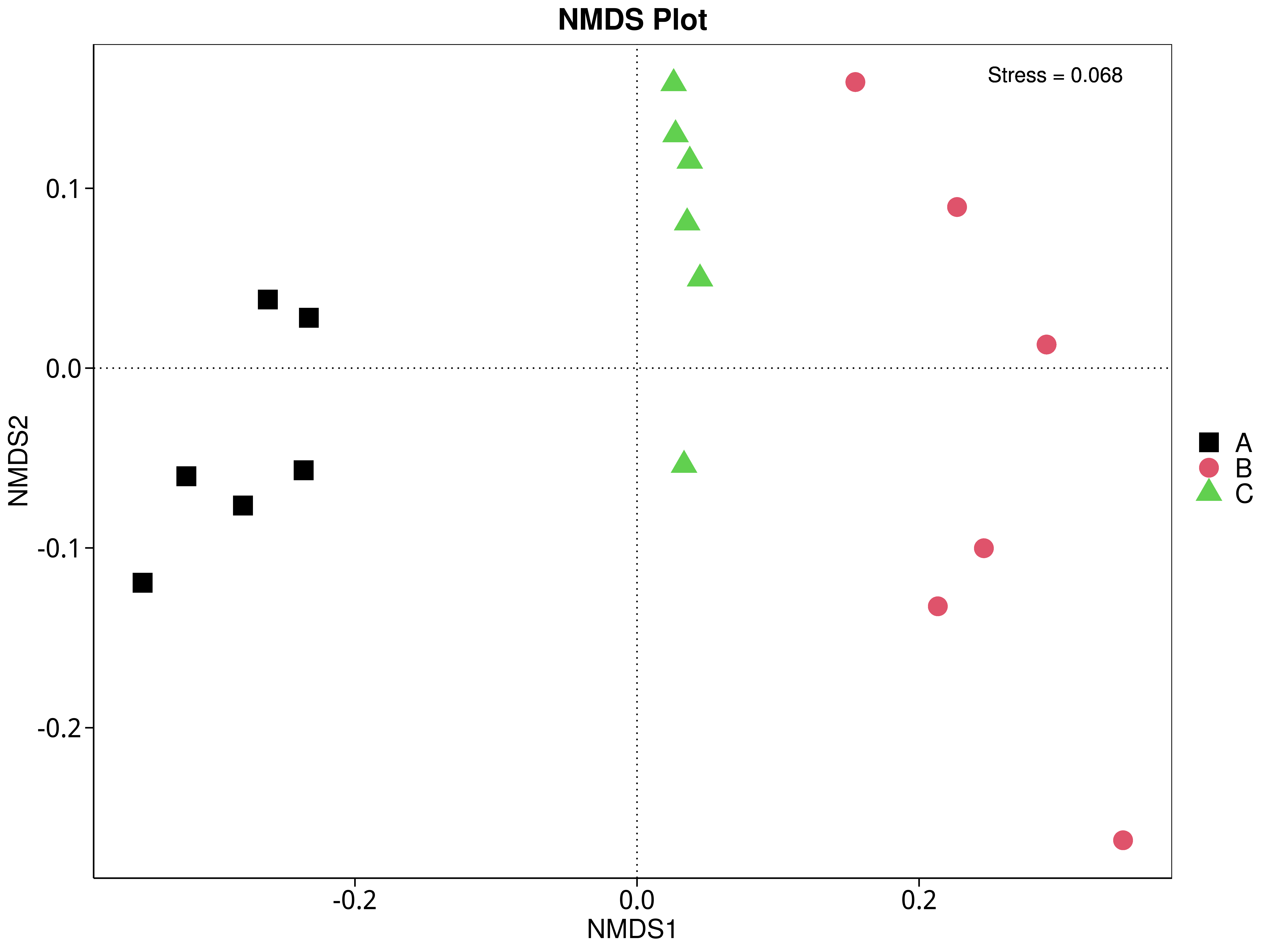
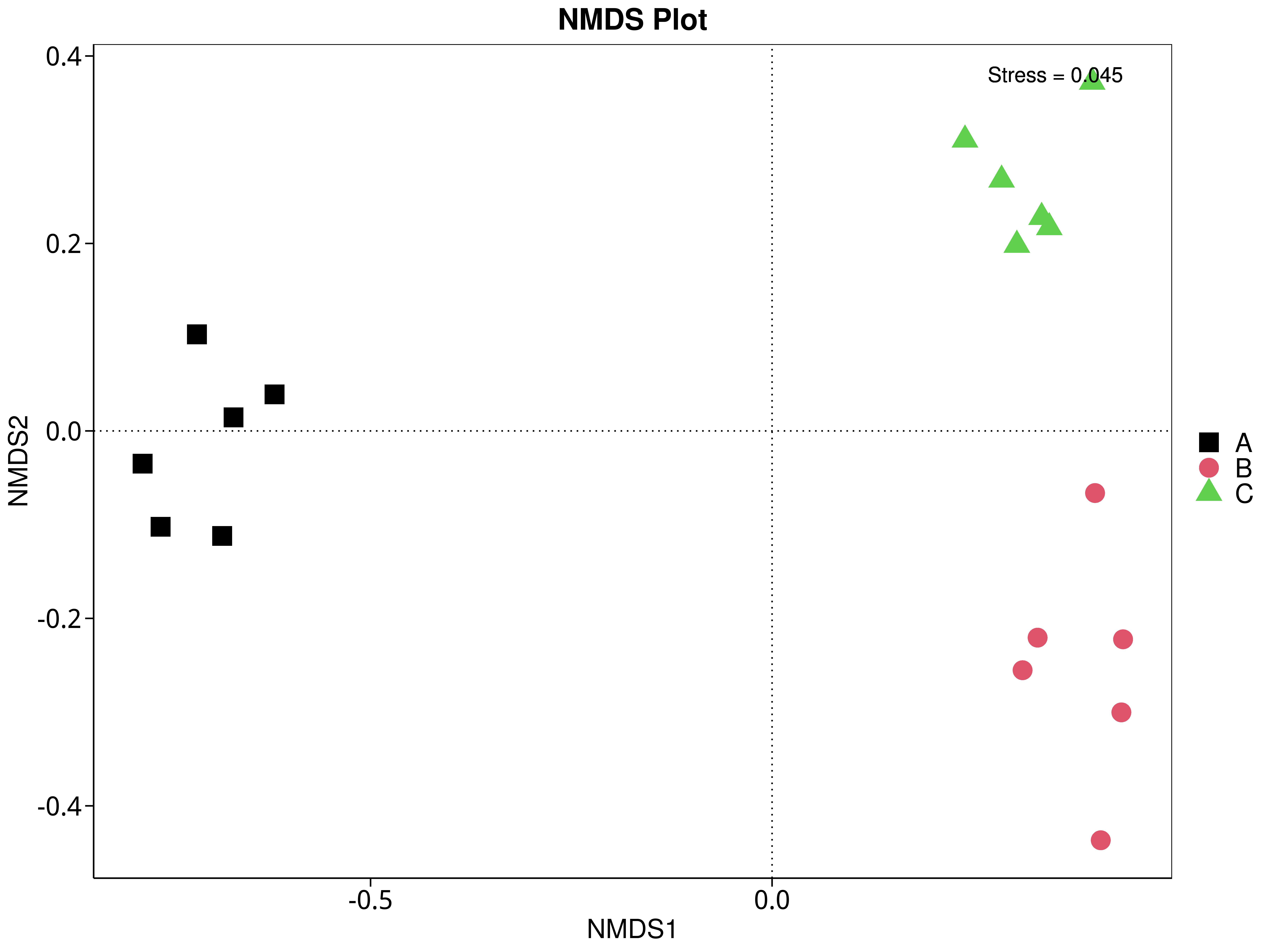
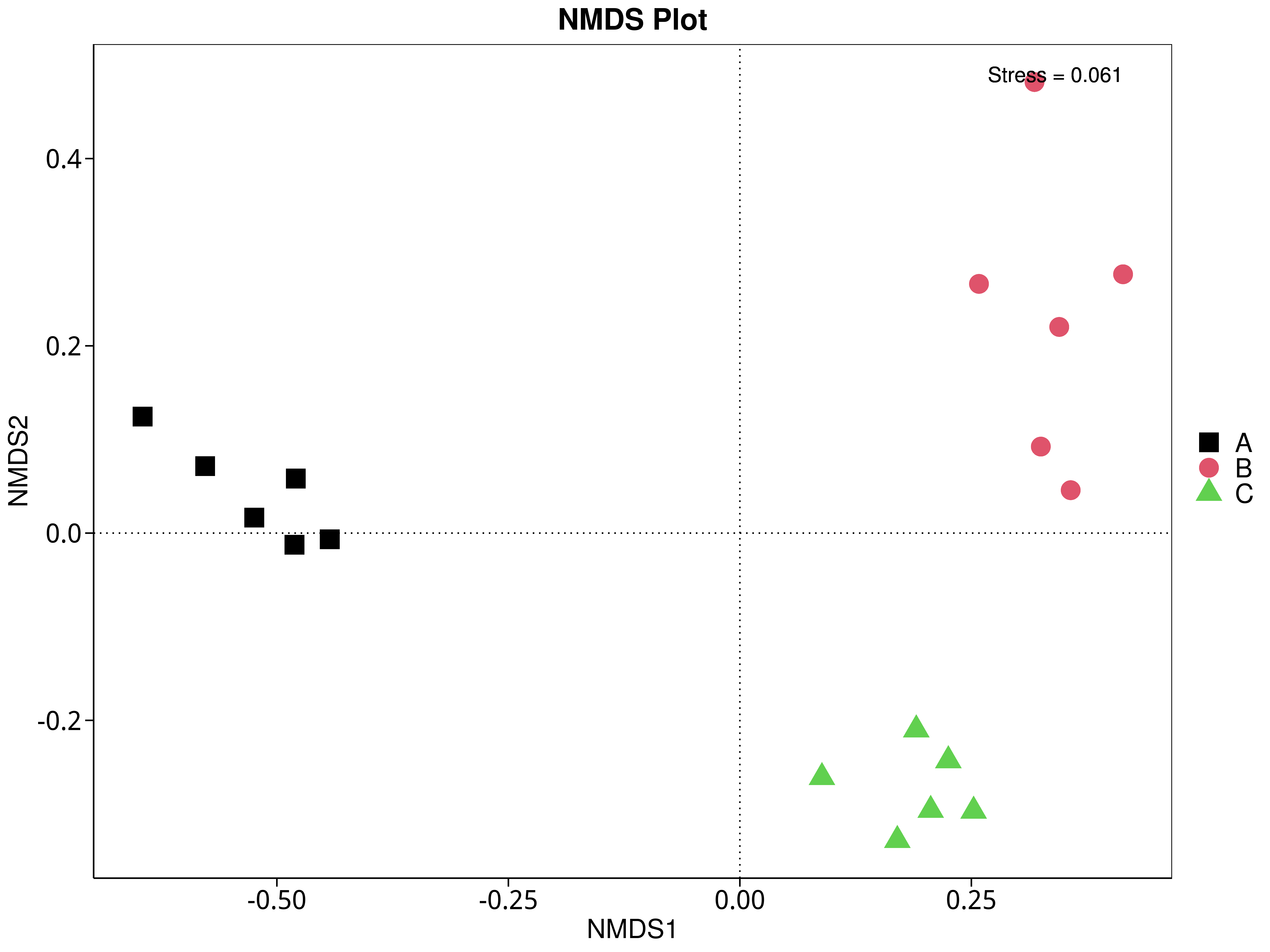
4.5.5 非度量多维尺度(NMDS)分析
NMDS是非线性模型,其目的是为了克服线性模型的缺点,更好地反映生态学数据的非线性结构(Legendre, 1998),应用NMDS分析,根据样本中包含的物种或功能信息,以点的形式反映在多维空间上,而不同样本间的差异程度则是通过点与点间的距离体现,能够反映样本的组间或组内差异。基于不同分类层级的物种或功能丰度表得到Bray-Curtis 距离矩阵,我们进行了 NMDS分析。基于物种和功能的NMDS分析结果展示如下:
4.5.5.1 Micro_NR
图4.40 基于Micro_NR水平的NMDS结果展示
说明:图中的每个点表示一个样本,点与点之间的距离表示差异程度,同一个组的样本使用同一种颜色表示。Stress小于0.2时,说明NMDS可以准确反映样本间的差异程度
结果目录:
标注样品名的NMDS图见 : result/05.Diversity/MicroNR/NMDS_group1/*/NMDS_lable.{png,pdf}
未标注样品名的NMDS图见 : result/05.Diversity/MicroNR/NMDS_group1/*/NMDS.{png,pdf}
未标示样品名称的带聚类圈的NMDS图见 : result/05.Diversity/MicroNR/NMDS_group1/*/NMDS_circle.{png,pdf}
标示样品名称的带聚类圈的NMDS图见 : result/05.Diversity/MicroNR/NMDS_group1/*/NMDS_circleLable.{png,pdf}
Stress 值分析结果见 : result/05.Diversity/MicroNR/NMDS_group1/*/NMDS_scores.xls
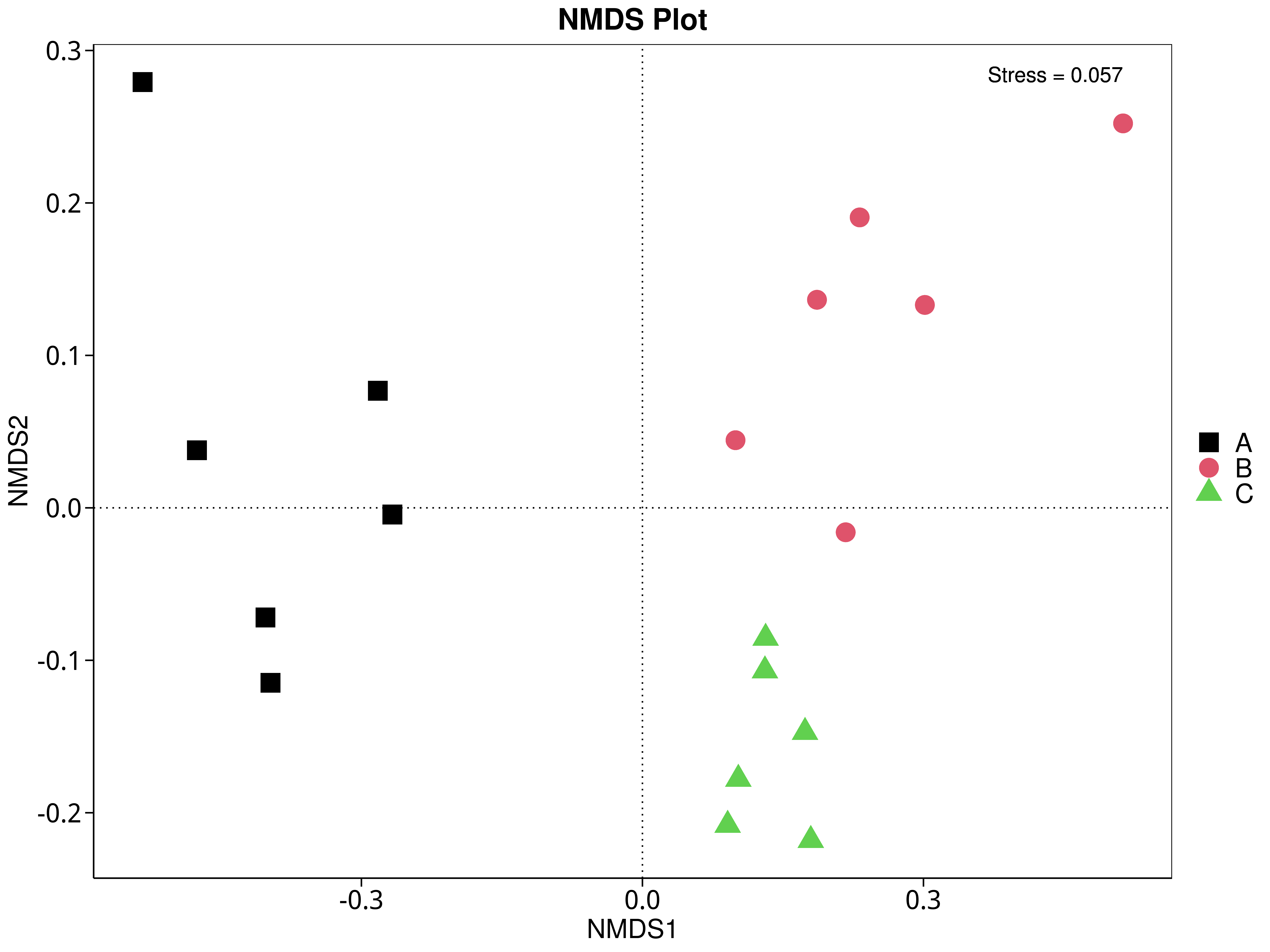
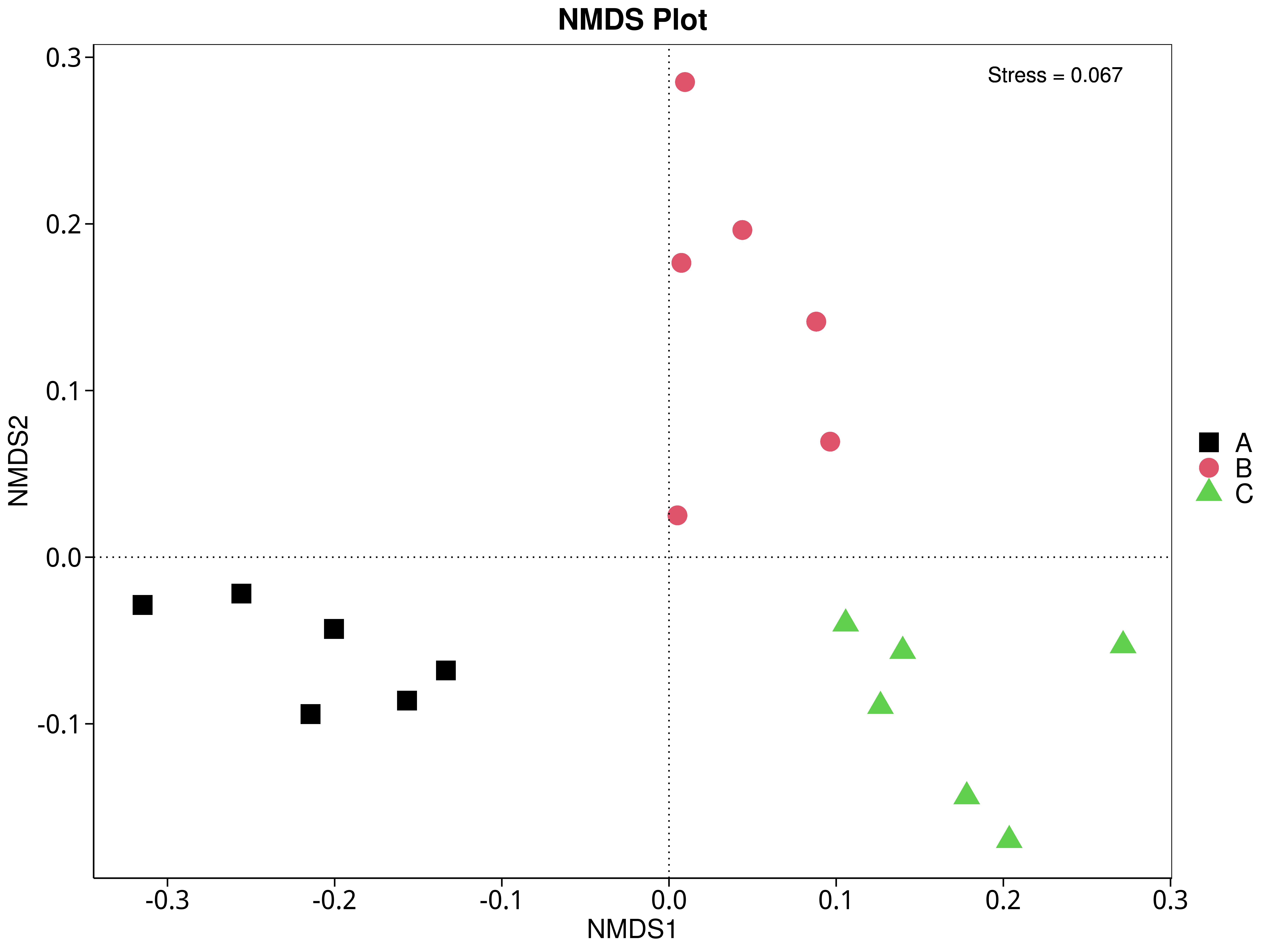
4.5.5.2 KEGG
图4.41 基于 KEGG 水平的 NMDS 结果展示
说明:图中的每个点表示一个样本,点与点之间的距离表示差异程度,同一个组的样本使用同一种颜色表示。Stress小于0.2时,说明NMDS可以准确反映样本间的差异程度
结果目录:
标注样品名的NMDS图见 : result/05.Diversity/KEGG/NMDS_group1/*/NMDS_lable.{png,pdf}
未标注样品名的NMDS图见 : result/05.Diversity/KEGG/NMDS_group1/*/NMDS.{png,pdf}
未标示样品名称的带聚类圈的NMDS图见 : result/05.Diversity/KEGG/NMDS_group1/*/NMDS_circle.{png,pdf}
标示样品名称的带聚类圈的NMDS图见 : result/05.Diversity/KEGG/NMDS_group1/*/NMDS_circleLable.{png,pdf}
Stress 值分析结果见 : result/05.Diversity/KEGG/NMDS_group1/*/NMDS_scores.xls
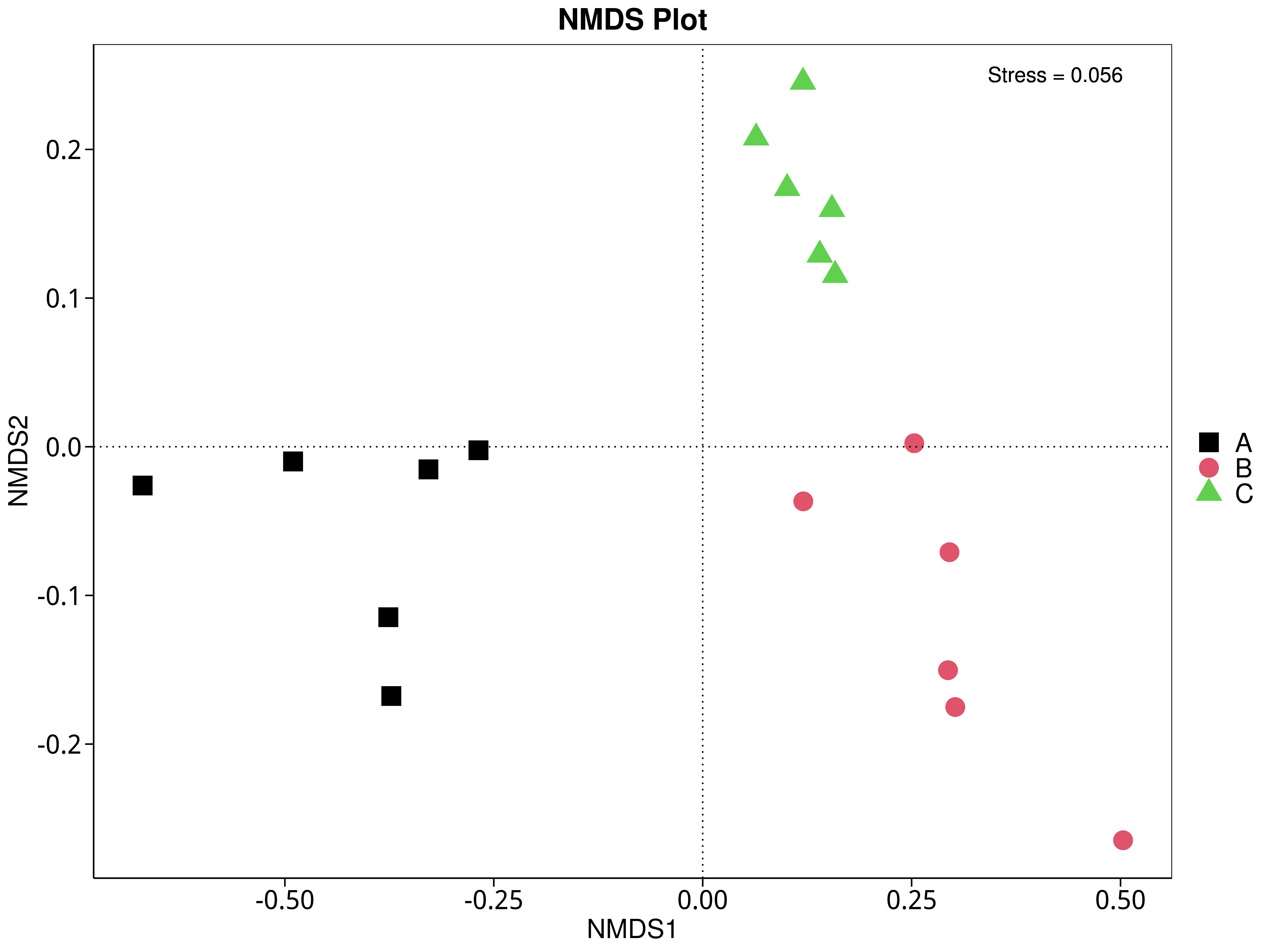
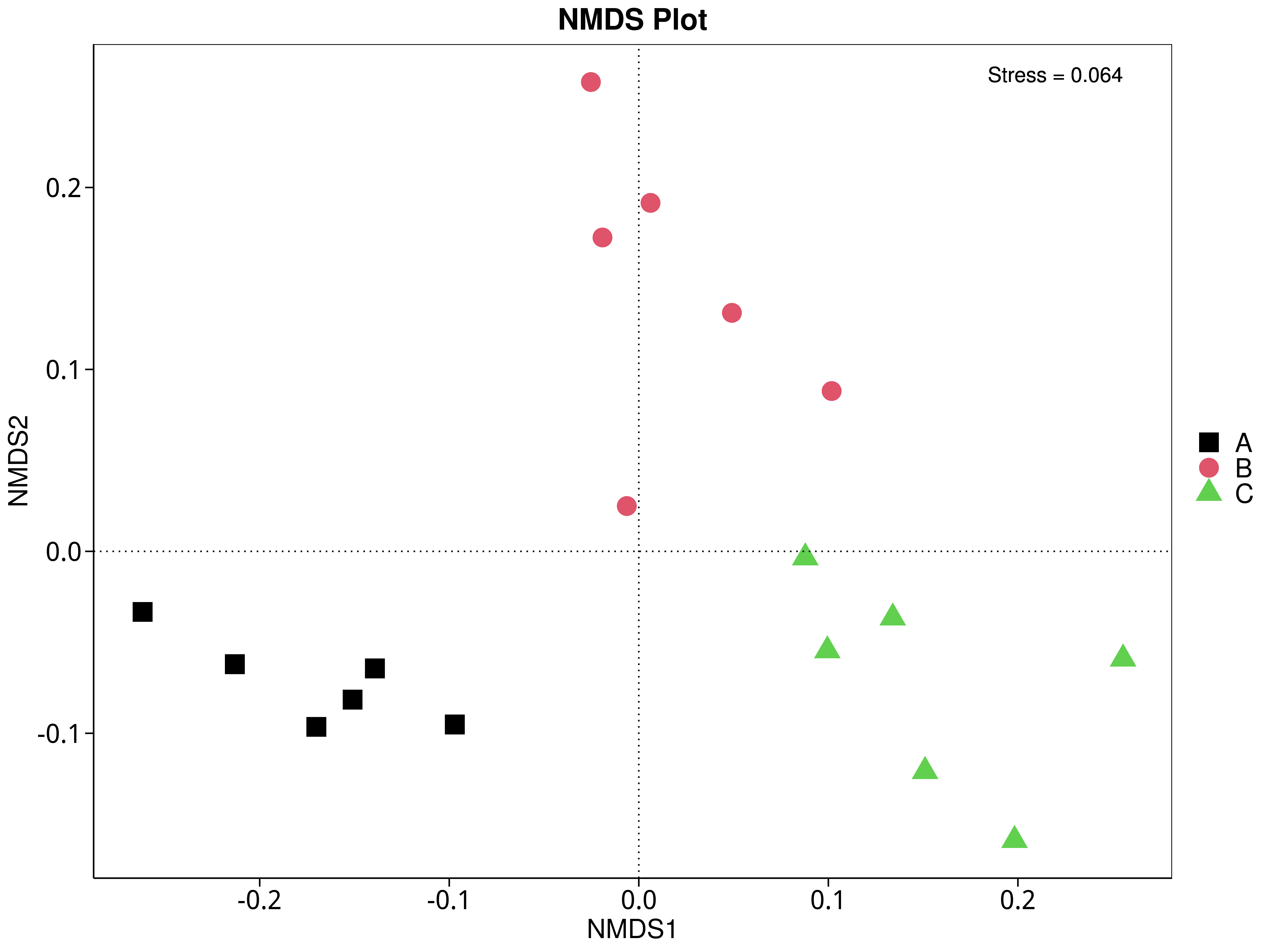
4.5.5.3 eggNOG
图4.42 基于 eggNOG 水平的 NMDS 结果展示
说明:图中的每个点表示一个样本,点与点之间的距离表示差异程度,同一个组的样本使用同一种颜色表示。Stress小于0.2时,说明NMDS可以准确反映样本间的差异程度
结果目录:
标注样品名的NMDS图见 : result/05.Diversity/eggNOG/NMDS_group1/*/NMDS_lable.{png,pdf}
未标注样品名的NMDS图见 : result/05.Diversity/eggNOG/NMDS_group1/*/NMDS.{png,pdf}
未标示样品名称的带聚类圈的NMDS图见 : result/05.Diversity/eggNOG/NMDS_group1/*/NMDS_circle.{png,pdf}
标示样品名称的带聚类圈的NMDS图见 : result/05.Diversity/eggNOG/NMDS_group1/*/NMDS_circleLable.{png,pdf}
Stress 值分析结果见 : result/05.Diversity/eggNOG/NMDS_group1/*/NMDS_scores.xls
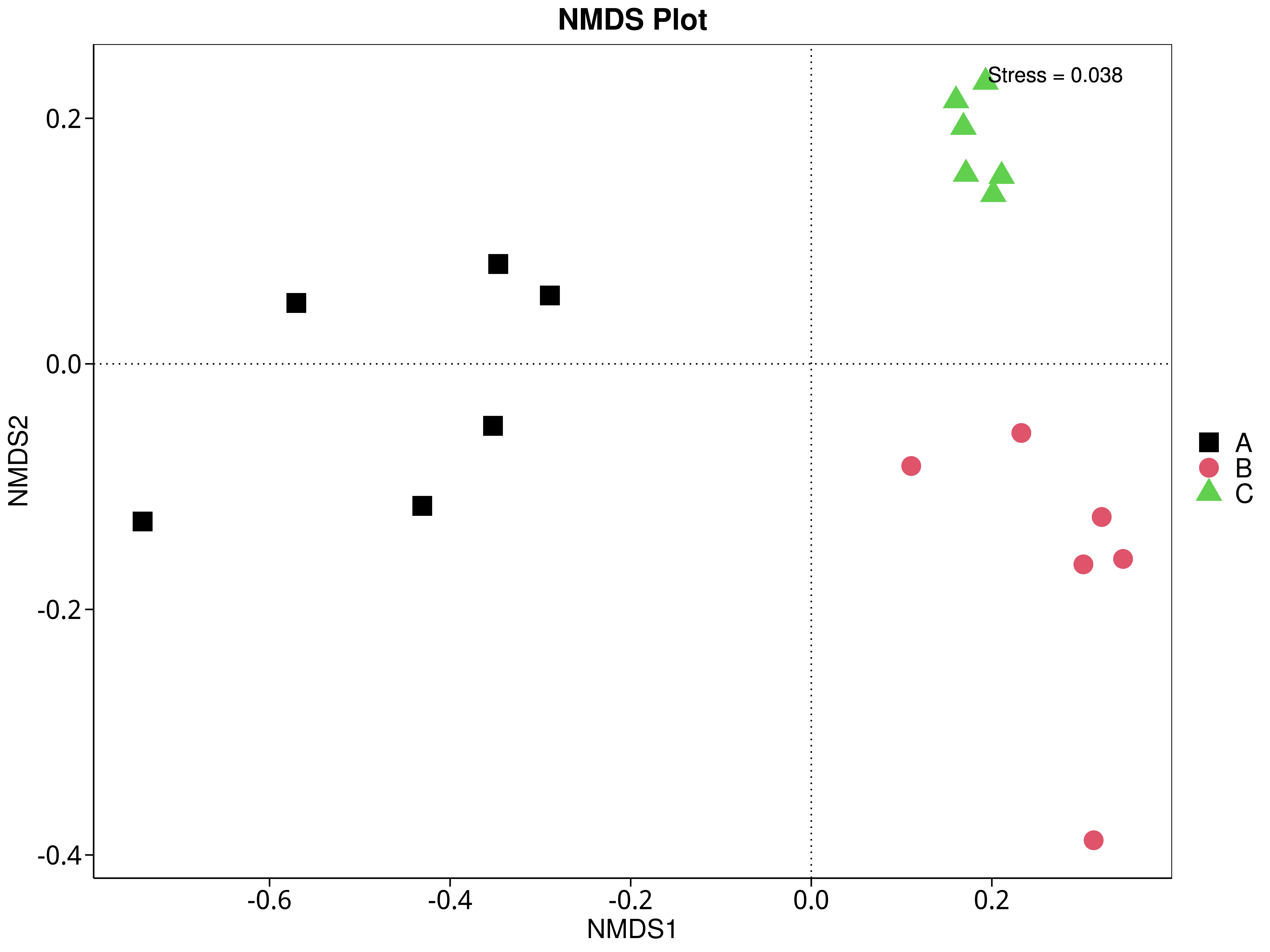
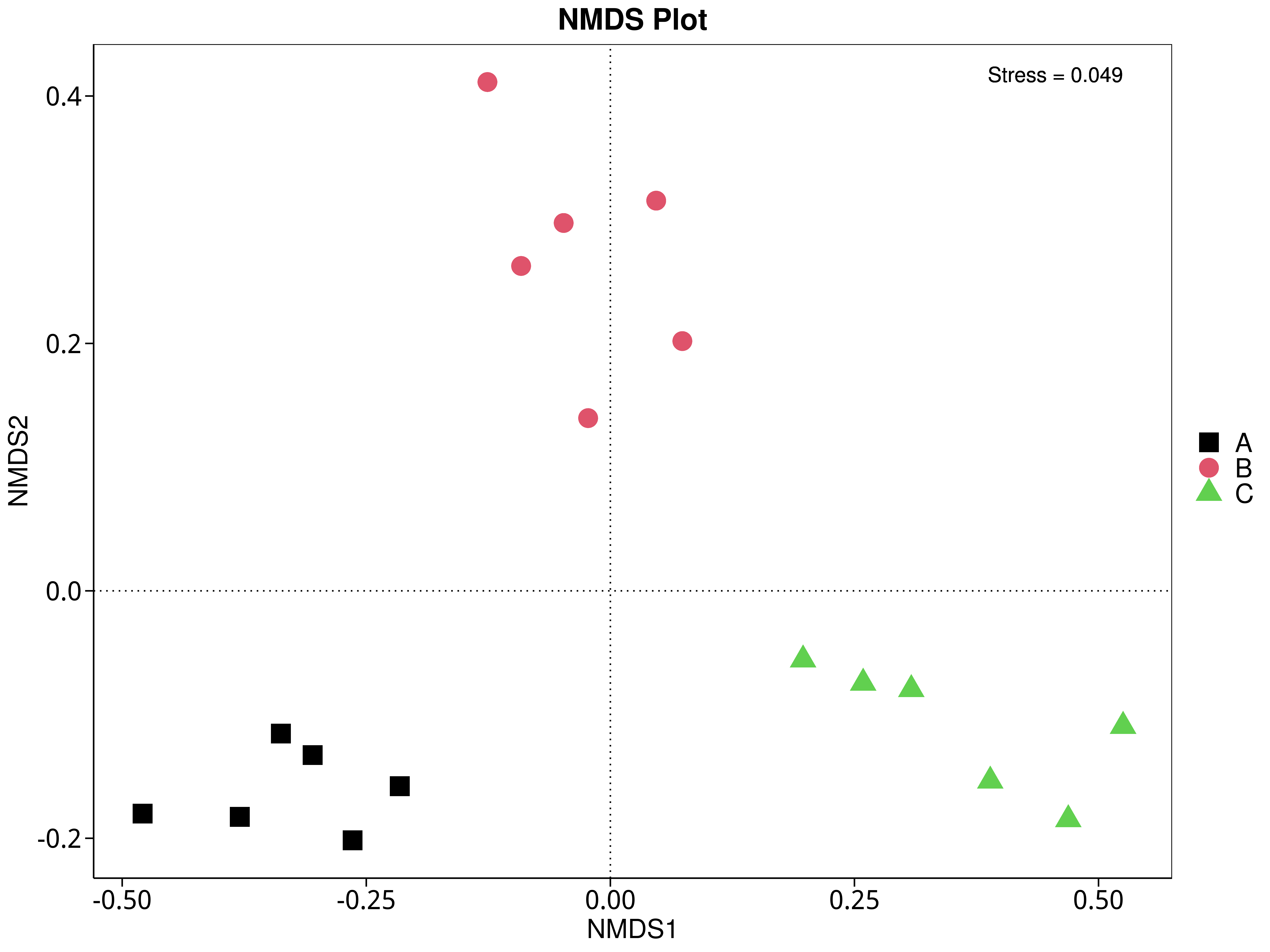
4.5.5.4 CAZy
图4.43 基于 CAZy 水平的 NMDS 结果展示
说明:图中的每个点表示一个样本,点与点之间的距离表示差异程度,同一个组的样本使用同一种颜色表示。Stress小于0.2时,说明NMDS可以准确反映样本间的差异程度
结果目录:
标注样品名的NMDS图见 : result/05.Diversity/CAZy/NMDS_group1/*/NMDS_lable.{png,pdf}
未标注样品名的NMDS图见 : result/05.Diversity/CAZy/NMDS_group1/*/NMDS.{png,pdf}
未标示样品名称的带聚类圈的NMDS图见 : result/05.Diversity/CAZy/NMDS_group1/*/NMDS_circle.{png,pdf}
标示样品名称的带聚类圈的NMDS图见 : result/05.Diversity/CAZy/NMDS_group1/*/NMDS_circleLable.{png,pdf}
Stress 值分析结果见 : result/05.Diversity/CAZy/NMDS_group1/*/NMDS_scores.xls
4.5.5.5 VFDB
图4.44 基于 VFDB 水平的 NMDS 结果展示
说明:图中的每个点表示一个样本,点与点之间的距离表示差异程度,同一个组的样本使用同一种颜色表示。Stress小于0.2时,说明NMDS可以准确反映样本间的差异程度
结果目录:
标注样品名的NMDS图见 : result/05.Diversity/VFDB/NMDS_group1/*/NMDS_lable.{png,pdf}
未标注样品名的NMDS图见 : result/05.Diversity/VFDB/NMDS_group1/*/NMDS.{png,pdf}
未标示样品名称的带聚类圈的NMDS图见 : result/05.Diversity/VFDB/NMDS_group1/*/NMDS_circle.{png,pdf}
标示样品名称的带聚类圈的NMDS图见 : result/05.Diversity/VFDB/NMDS_group1/*/NMDS_circleLable.{png,pdf}
Stress 值分析结果见 : result/05.Diversity/VFDB/NMDS_group1/*/NMDS_scores.xls
4.5.5.6 PHI
图4.45 基于 PHI 水平的 NMDS 结果展示
说明:图中的每个点表示一个样本,点与点之间的距离表示差异程度,同一个组的样本使用同一种颜色表示。Stress小于0.2时,说明NMDS可以准确反映样本间的差异程度
结果目录:
标注样品名的NMDS图见 : result/05.Diversity/PHI/NMDS_group1/*/NMDS_lable.{png,pdf}
未标注样品名的NMDS图见 : result/05.Diversity/PHI/NMDS_group1/*/NMDS.{png,pdf}
未标示样品名称的带聚类圈的NMDS图见 : result/05.Diversity/PHI/NMDS_group1/*/NMDS_circle.{png,pdf}
标示样品名称的带聚类圈的NMDS图见 : result/05.Diversity/PHI/NMDS_group1/*/NMDS_circleLable.{png,pdf}
Stress 值分析结果见 : result/05.Diversity/PHI/NMDS_group1/*/NMDS_scores.xls
4.6 组间物种与功能差异统计检验分析
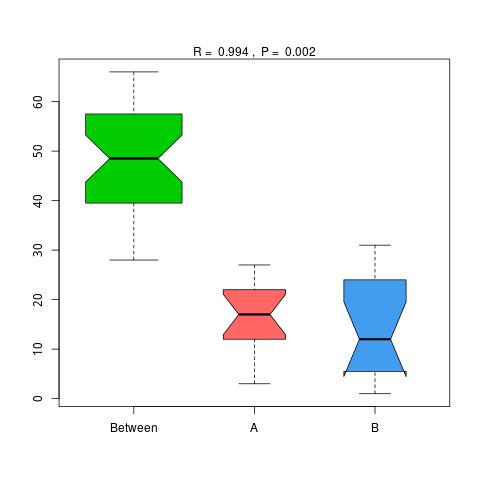
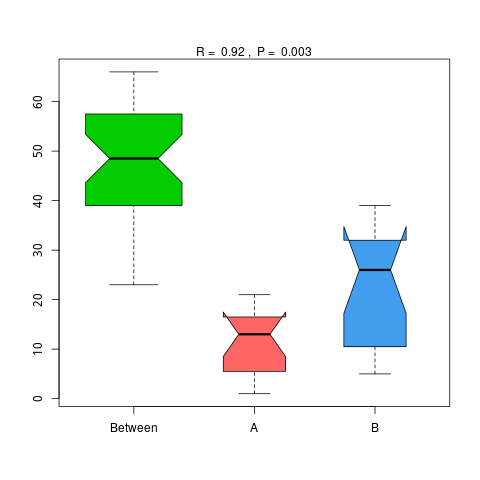
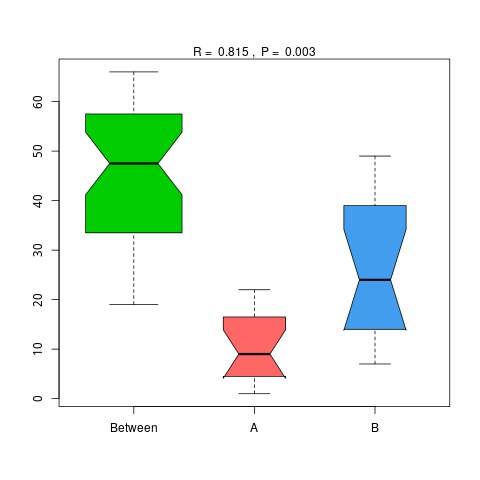
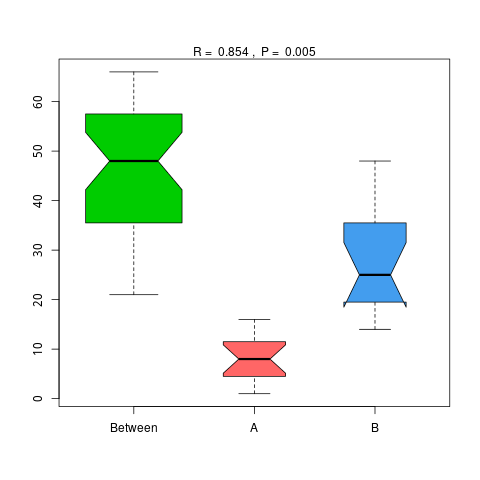
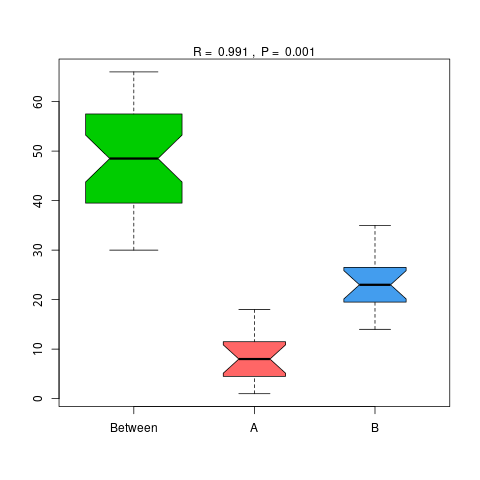
4.6.1 Anosim 分析
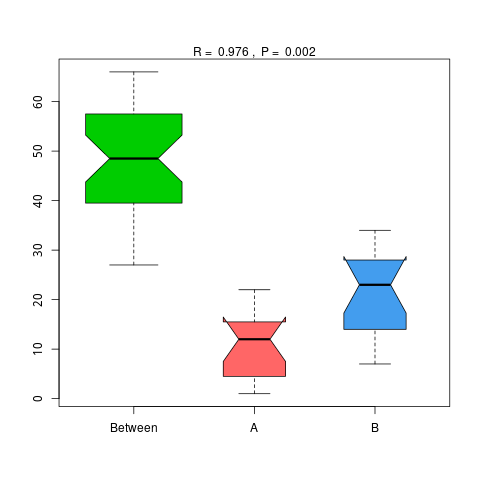
Anosim分析是一种非参数检验,用来检验组间的差异是否显著大于组内差异,从而判断分组是否有意义,详细计算过程可查看Anosim(https://www.rdocumentation.org/packages/vegan/versions/2.3-5/topics/anosim)。基于物种和功能水平的Anosim分析结果如下:
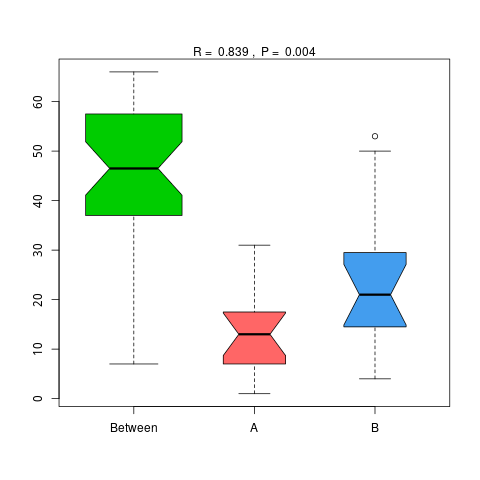
4.6.1.1 Micro_NR
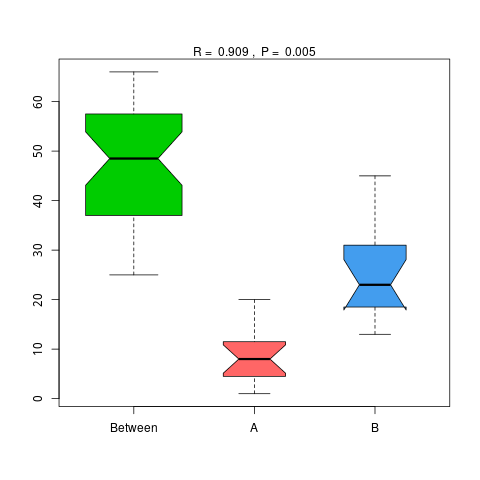
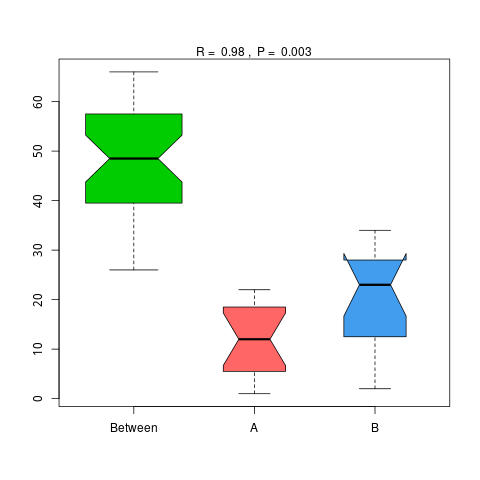
图4.46 基于 Micro_NR 水平的 Anosim 分析
说明:横向为分组信息,纵向为距离信息。Between为两组合并信息,between中位线高于另外两组中位线为分组信息较好。R-value介于(-1,1)之间,R-value大于0,说明组间差异大于组内差异。R-value小于0,说明组内差异大于组间差异,统计分析的可信度用 P-value 表示,P< 0.05 表示统计具有显著性。
结果目录:
Anosim分析结果见 : result/06.StatisticalTest/MicroNR/Anosim_group1/*/*.{pdf,png}
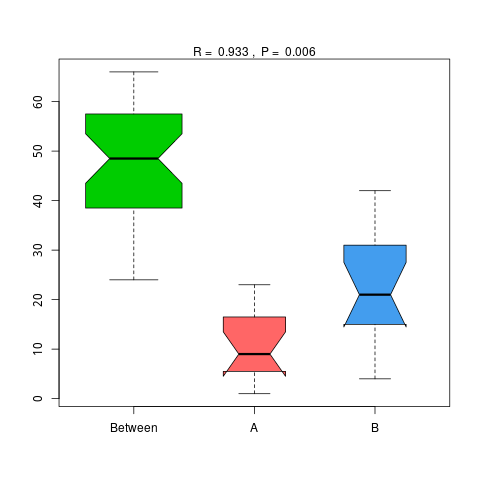
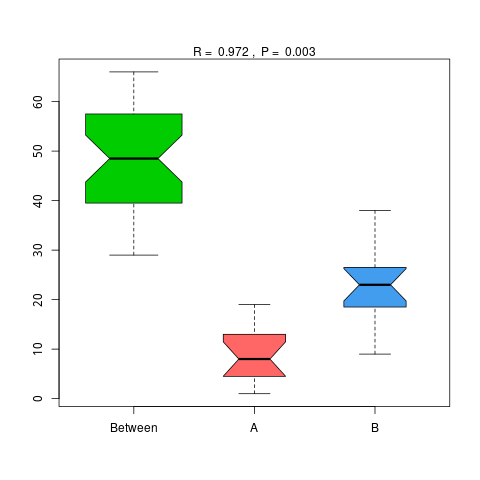
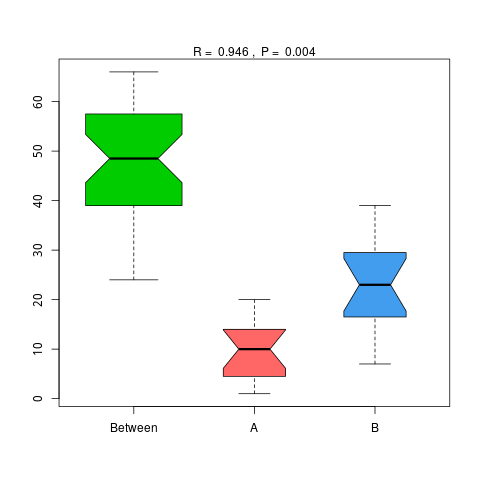
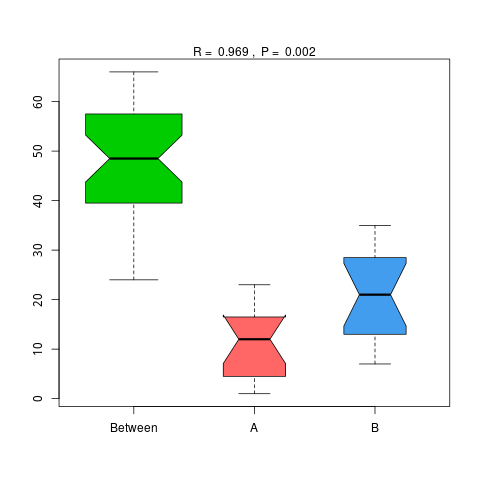
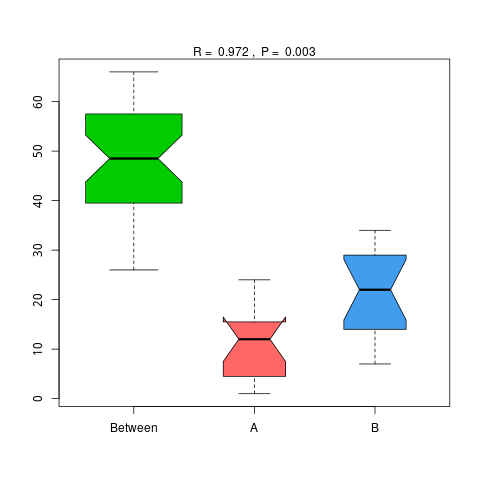
4.6.1.2 KEGG
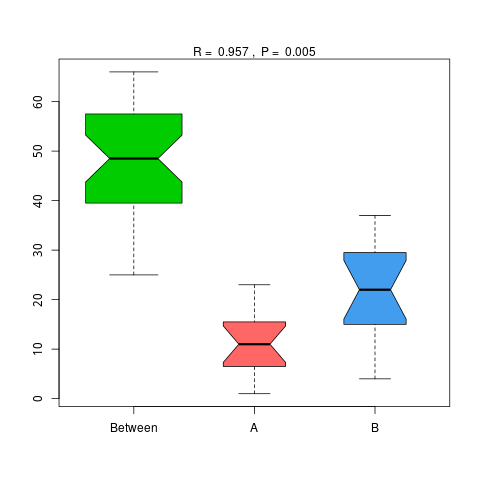
图4.47 基于 KEGG 水平的 Anosim 分析
说明:横向为分组信息,纵向为距离信息。Between为两组合并信息,between中位线高于另外两组中位线为分组信息较好。R-value介于(-1,1)之间,R-value大于0,说明组间差异大于组内差异。R-value小于0,说明组内差异大于组间差异,统计分析的可信度用 P-value 表示,P< 0.05 表示统计具有显著性。
结果目录:
Anosim分析结果见 : result/06.StatisticalTest/KEGG/Anosim_group1/*/*.{pdf,png}
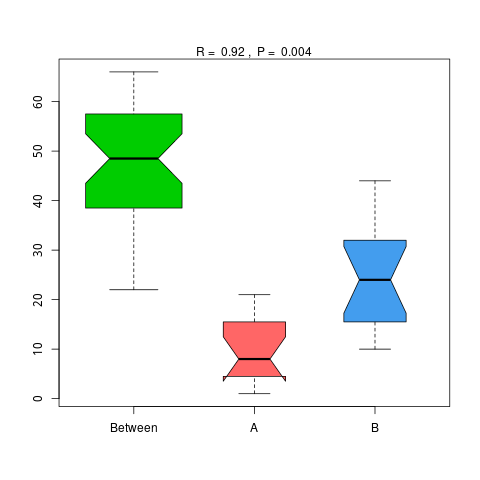
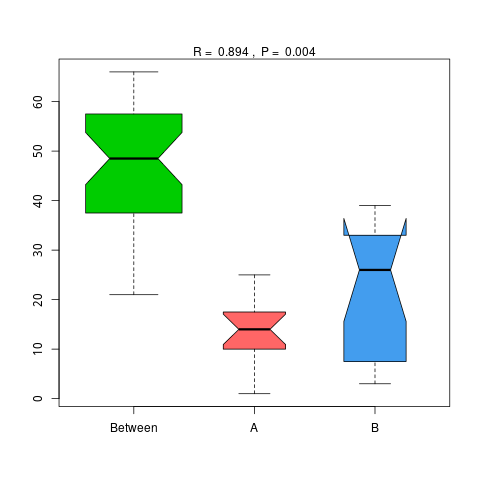
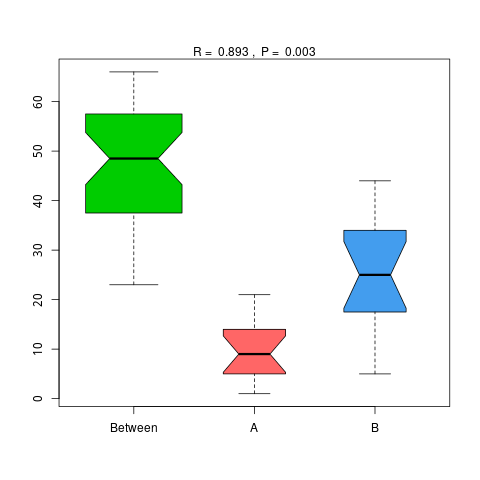
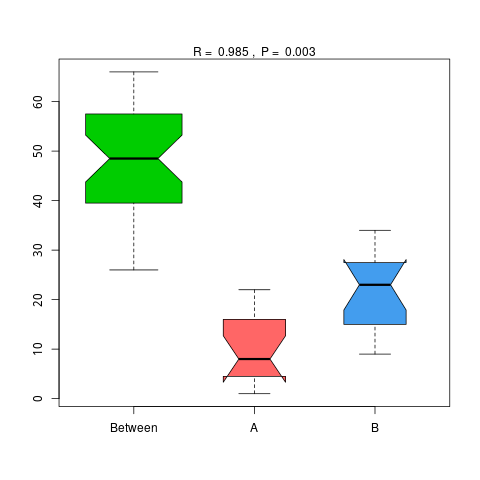
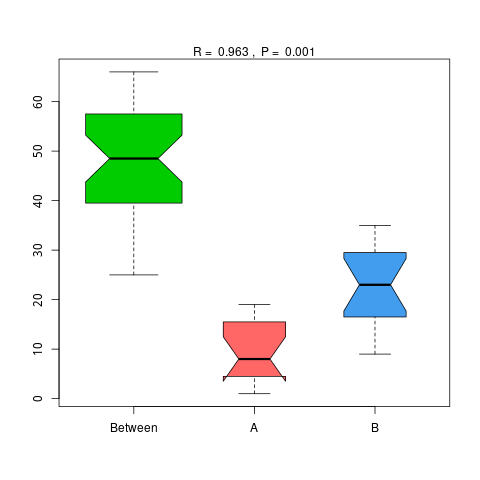
4.6.1.3 eggNOG
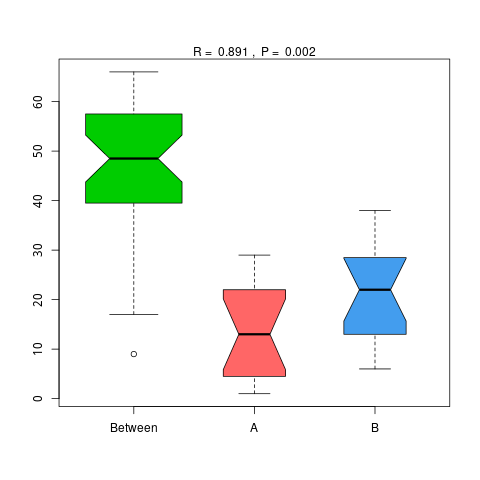
图4.48 基于 eggNOG 水平的 Anosim 分析
说明:横向为分组信息,纵向为距离信息。Between为两组合并信息,between中位线高于另外两组中位线为分组信息较好。R-value介于(-1,1)之间,R-value大于0,说明组间差异大于组内差异。R-value小于0,说明组内差异大于组间差异,统计分析的可信度用 P-value 表示,P< 0.05 表示统计具有显著性。
结果目录:
Anosim分析结果见 : result/06.StatisticalTest/eggNOG/Anosim_group1/*/*.{pdf,png}
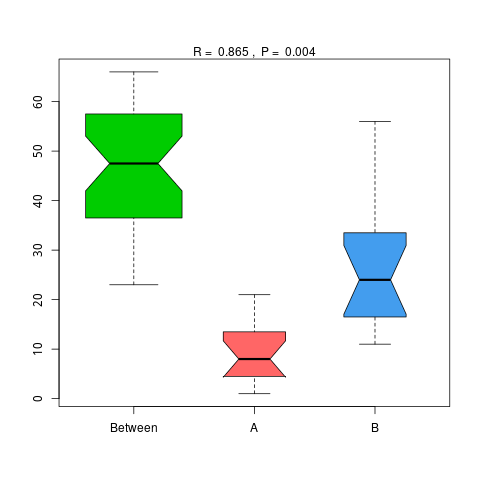
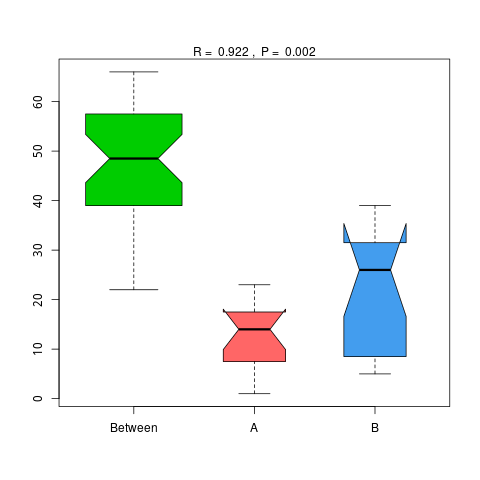
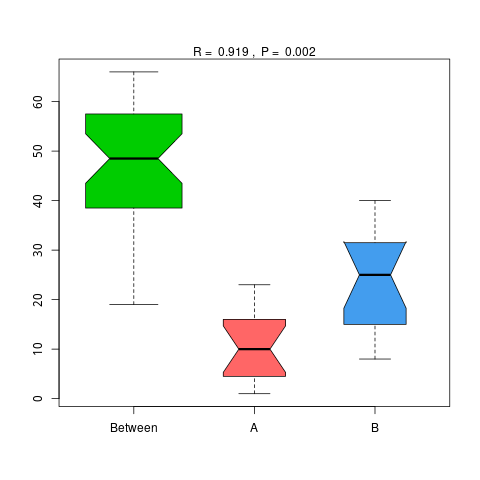
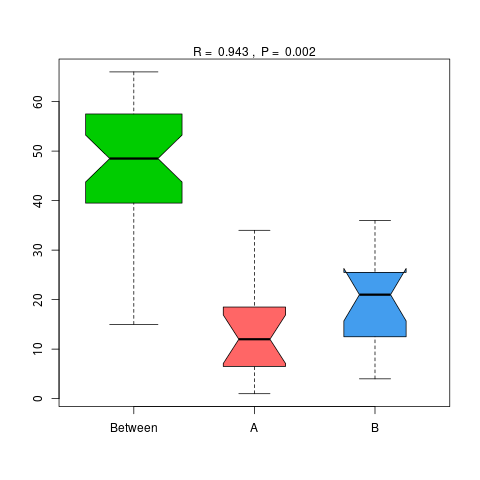
4.6.1.4 CAZy
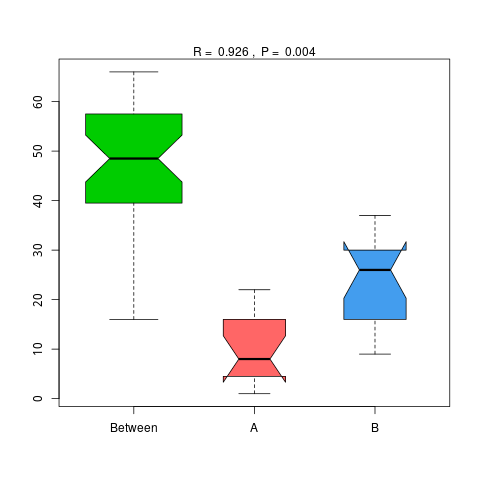
图4.49 基于 CAZy 水平的 Anosim 分析
说明:横向为分组信息,纵向为距离信息。Between为两组合并信息,between中位线高于另外两组中位线为分组信息较好。R-value介于(-1,1)之间,R-value大于0,说明组间差异大于组内差异。R-value小于0,说明组内差异大于组间差异,统计分析的可信度用 P-value 表示,P< 0.05 表示统计具有显著性。
结果目录:
Anosim分析结果见 : result/06.StatisticalTest/CAZy/Anosim_group1/*/*.{pdf,png}
4.6.1.5 VFDB
图4.50 基于 VFDB 水平的 Anosim 分析
说明:横向为分组信息,纵向为距离信息。Between为两组合并信息,between中位线高于另外两组中位线为分组信息较好。R-value介于(-1,1)之间,R-value大于0,说明组间差异大于组内差异。R-value小于0,说明组内差异大于组间差异,统计分析的可信度用 P-value 表示,P< 0.05 表示统计具有显著性。
结果目录:
Anosim分析结果见 : result/06.StatisticalTest/VFDB/Anosim_group1/*/*.{pdf,png}
4.6.1.6 PHI
图4.51 基于 PHI 水平的 Anosim 分析
说明:横向为分组信息,纵向为距离信息。Between为两组合并信息,between中位线高于另外两组中位线为分组信息较好。R-value介于(-1,1)之间,R-value大于0,说明组间差异大于组内差异。R-value小于0,说明组内差异大于组间差异,统计分析的可信度用 P-value 表示,P< 0.05 表示统计具有显著性。
结果目录:
Anosim分析结果见 : result/06.StatisticalTest/PHI/Anosim_group1/*/*.{pdf,png}
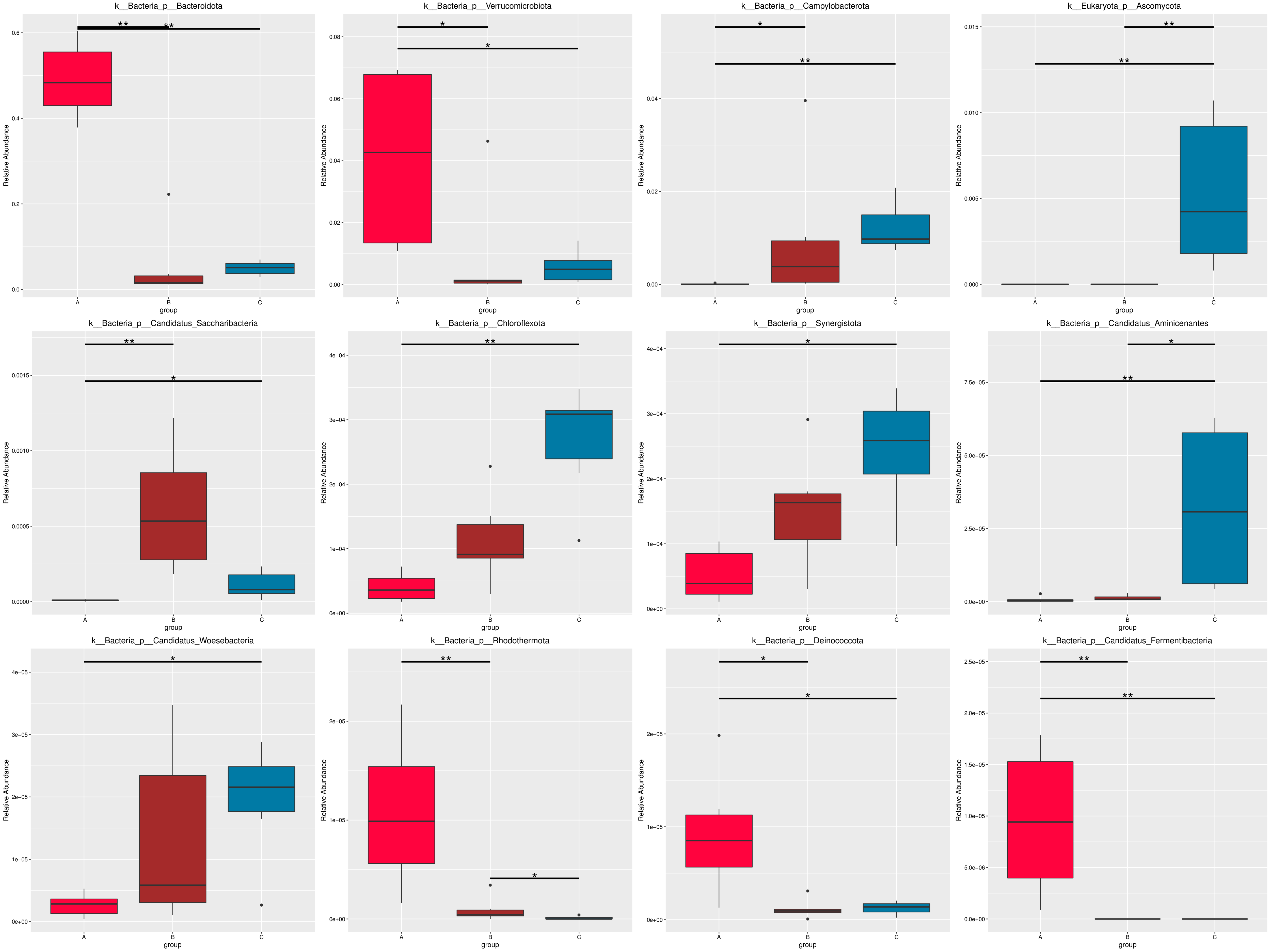
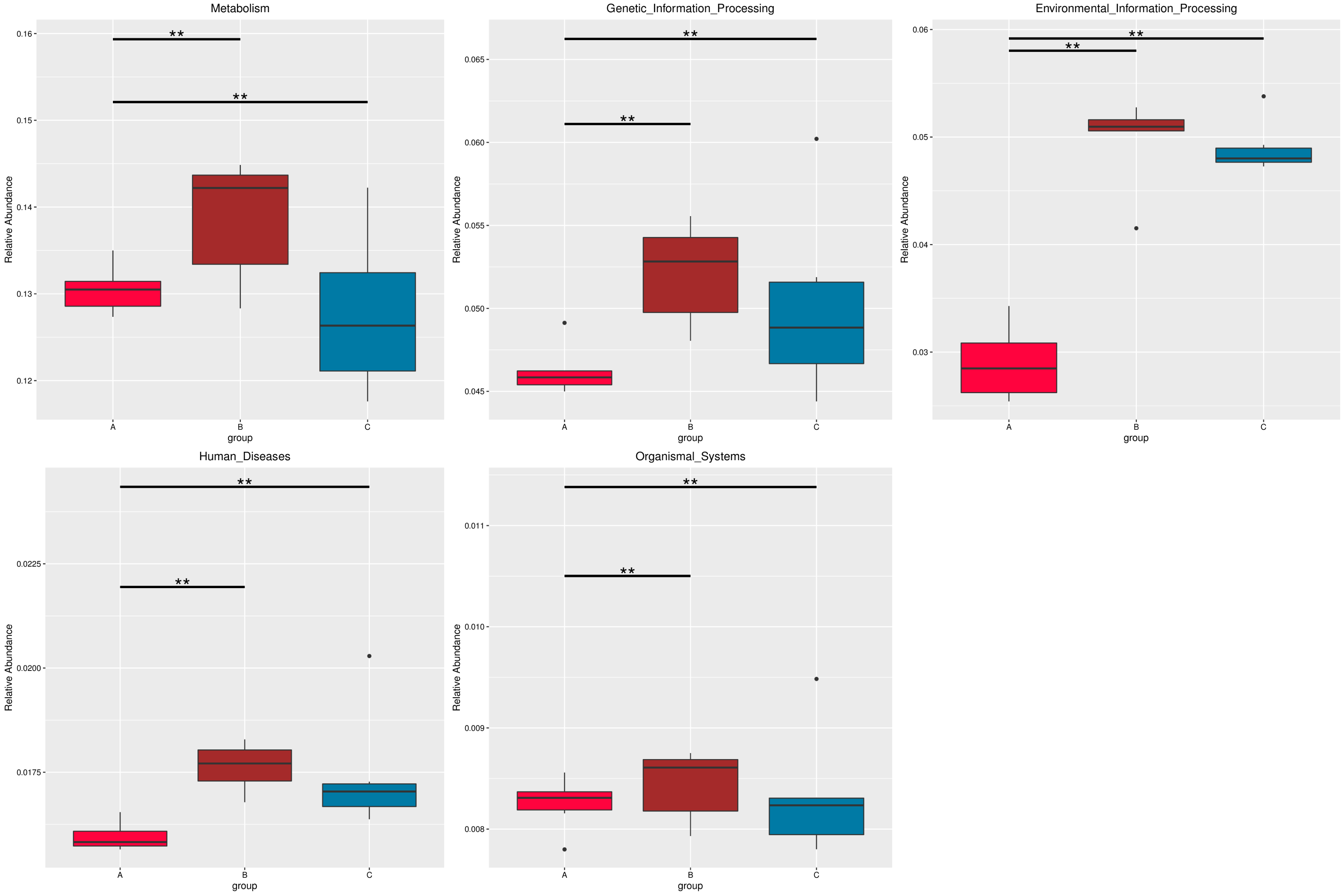
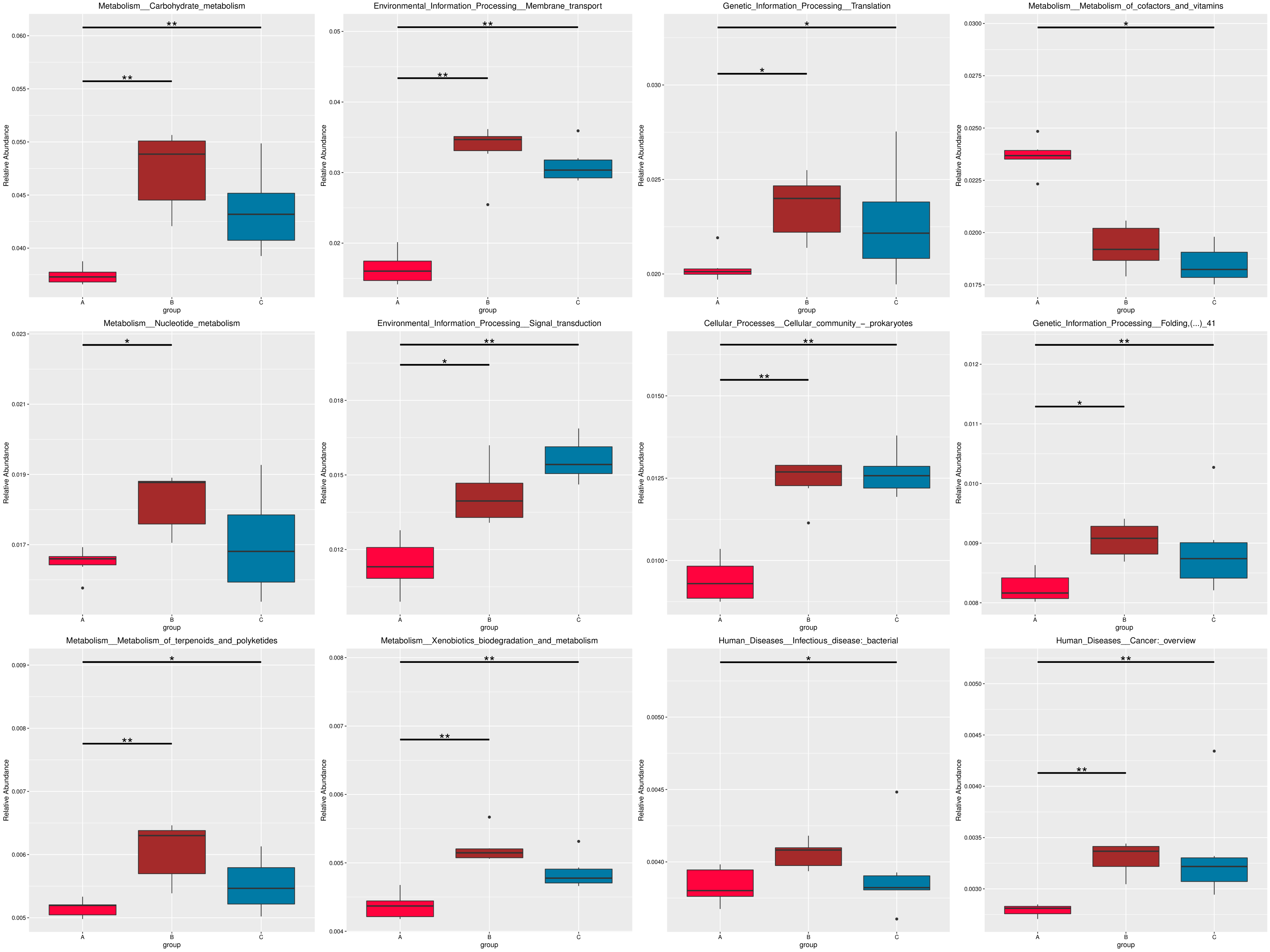
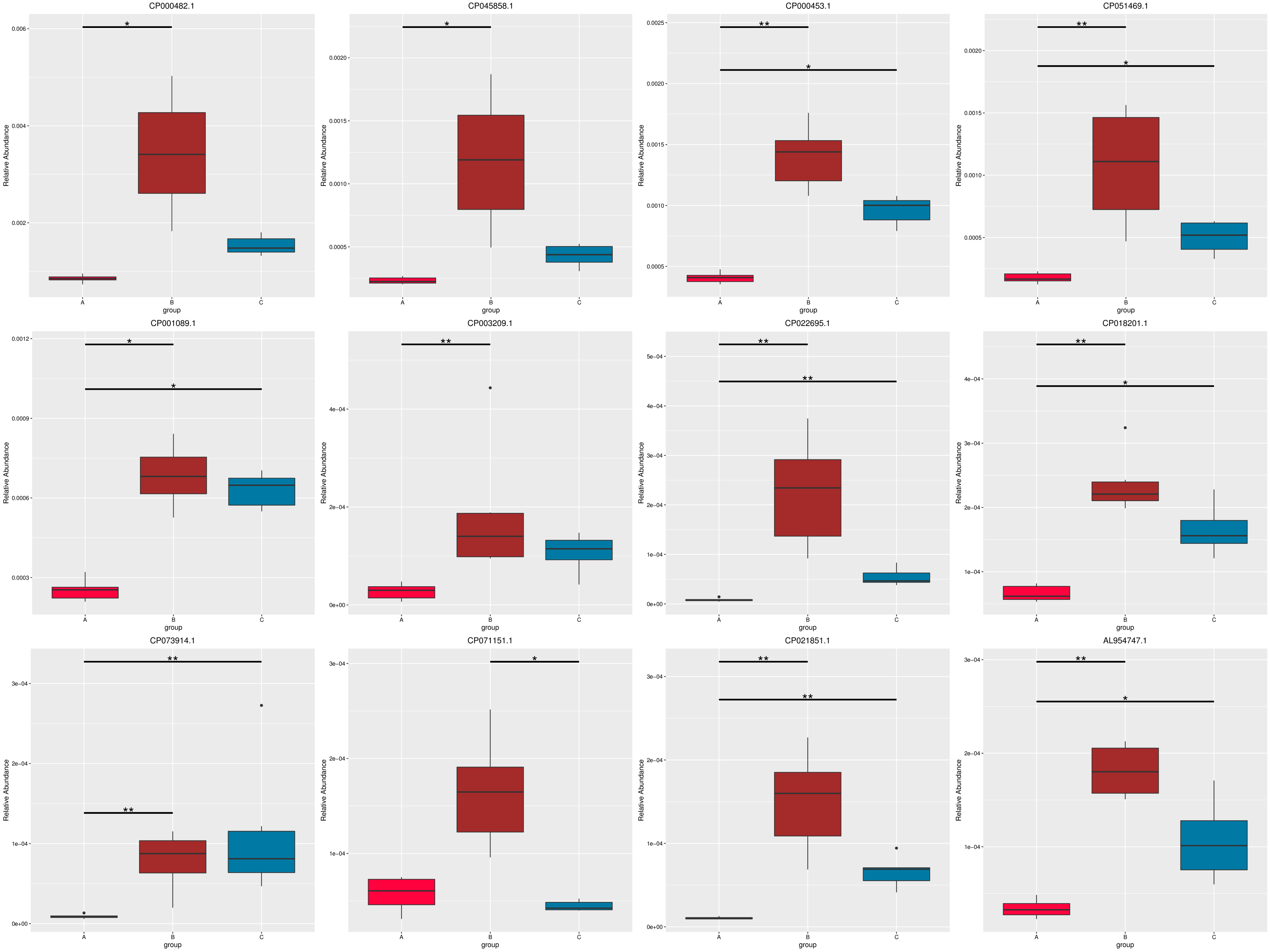
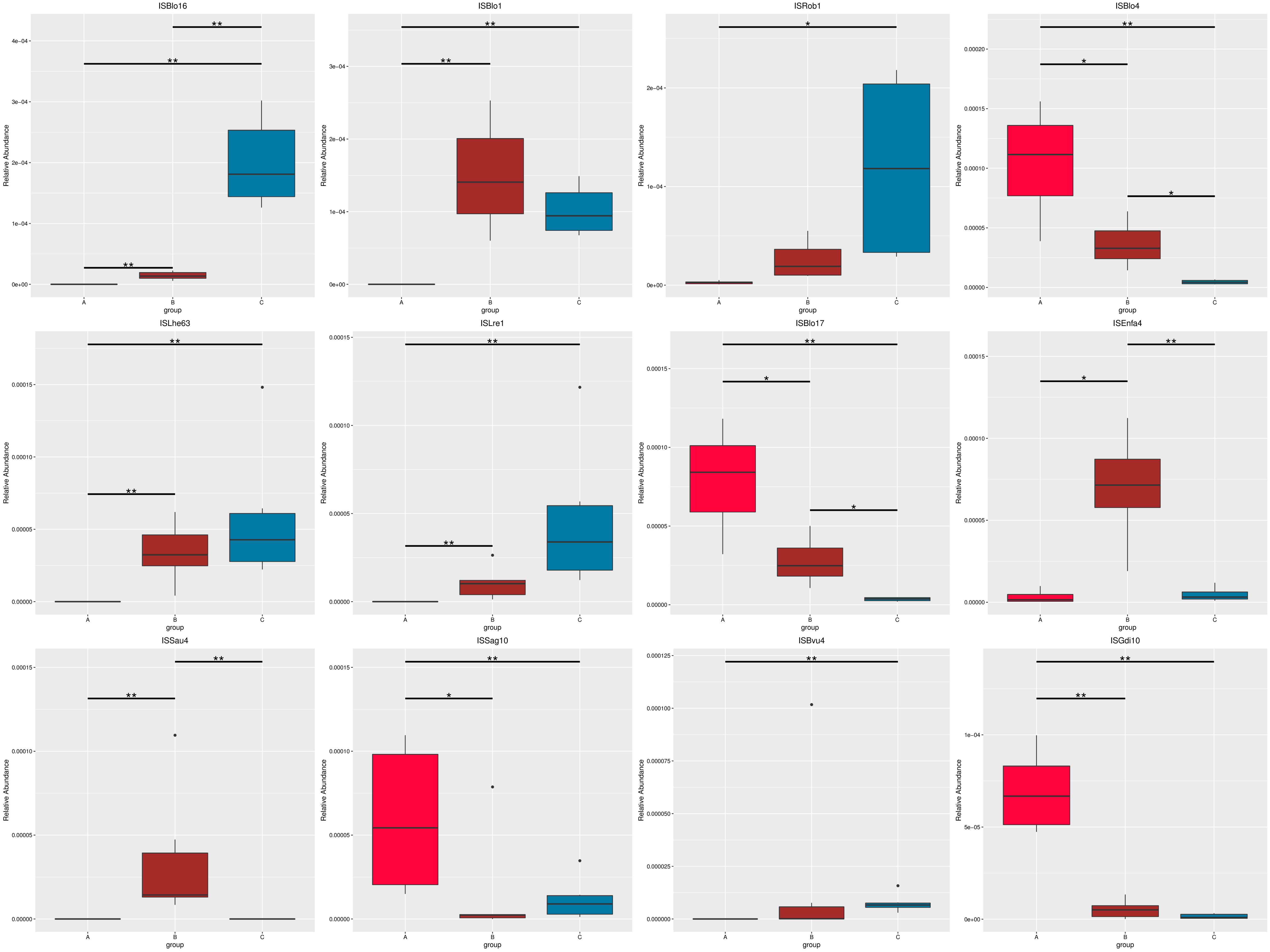
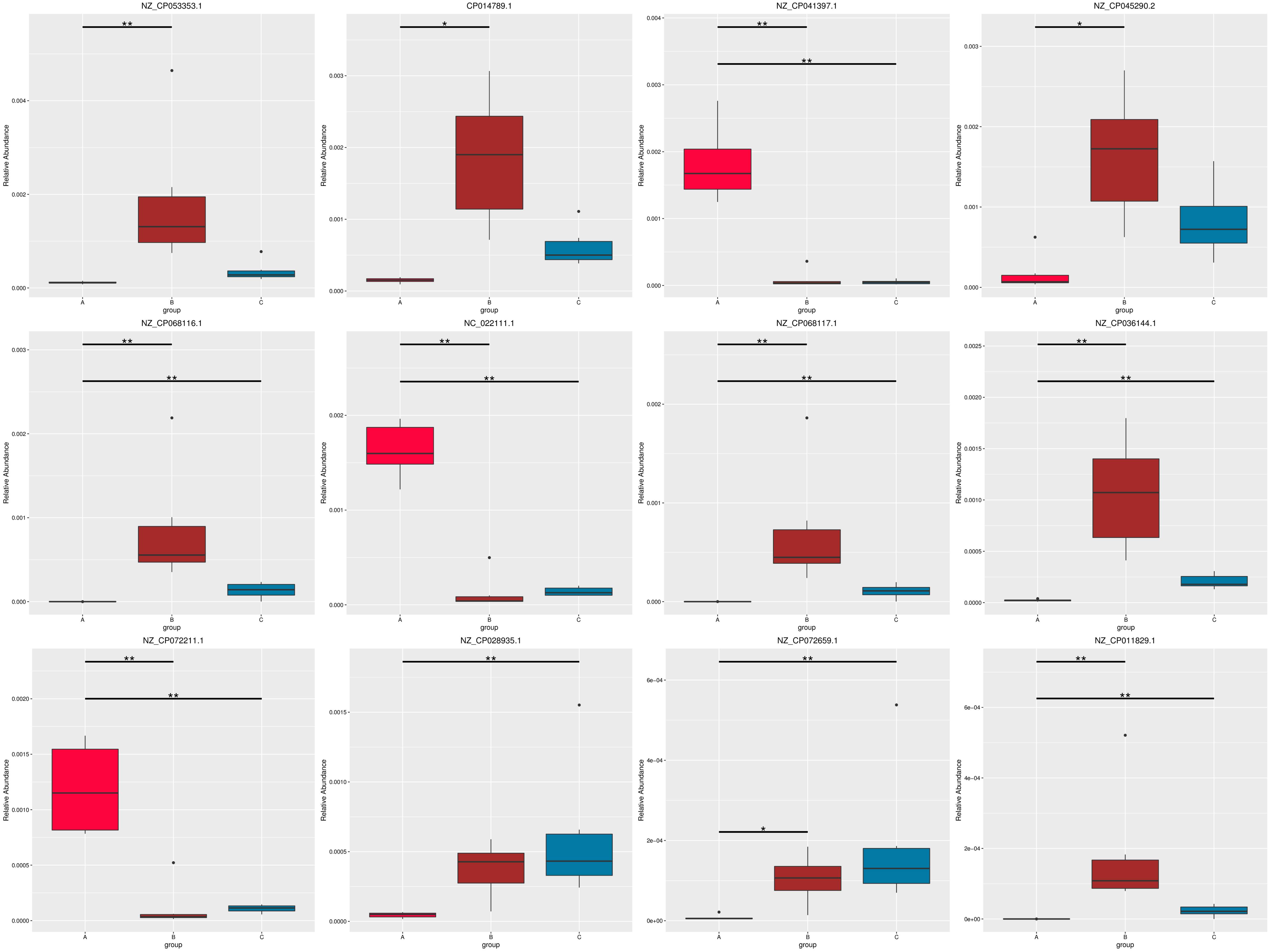
4.6.2 MetaGenomeSeq 分析
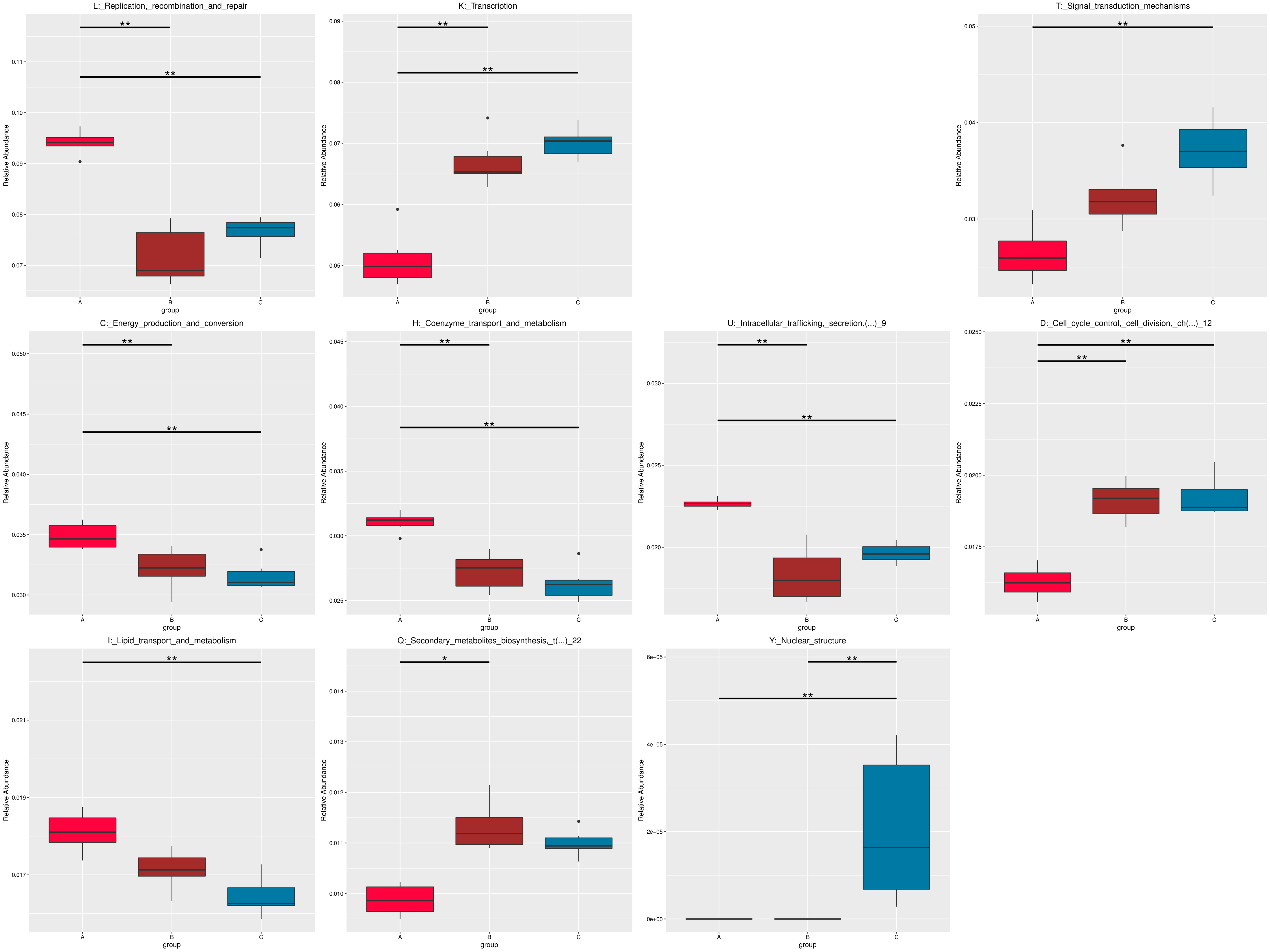
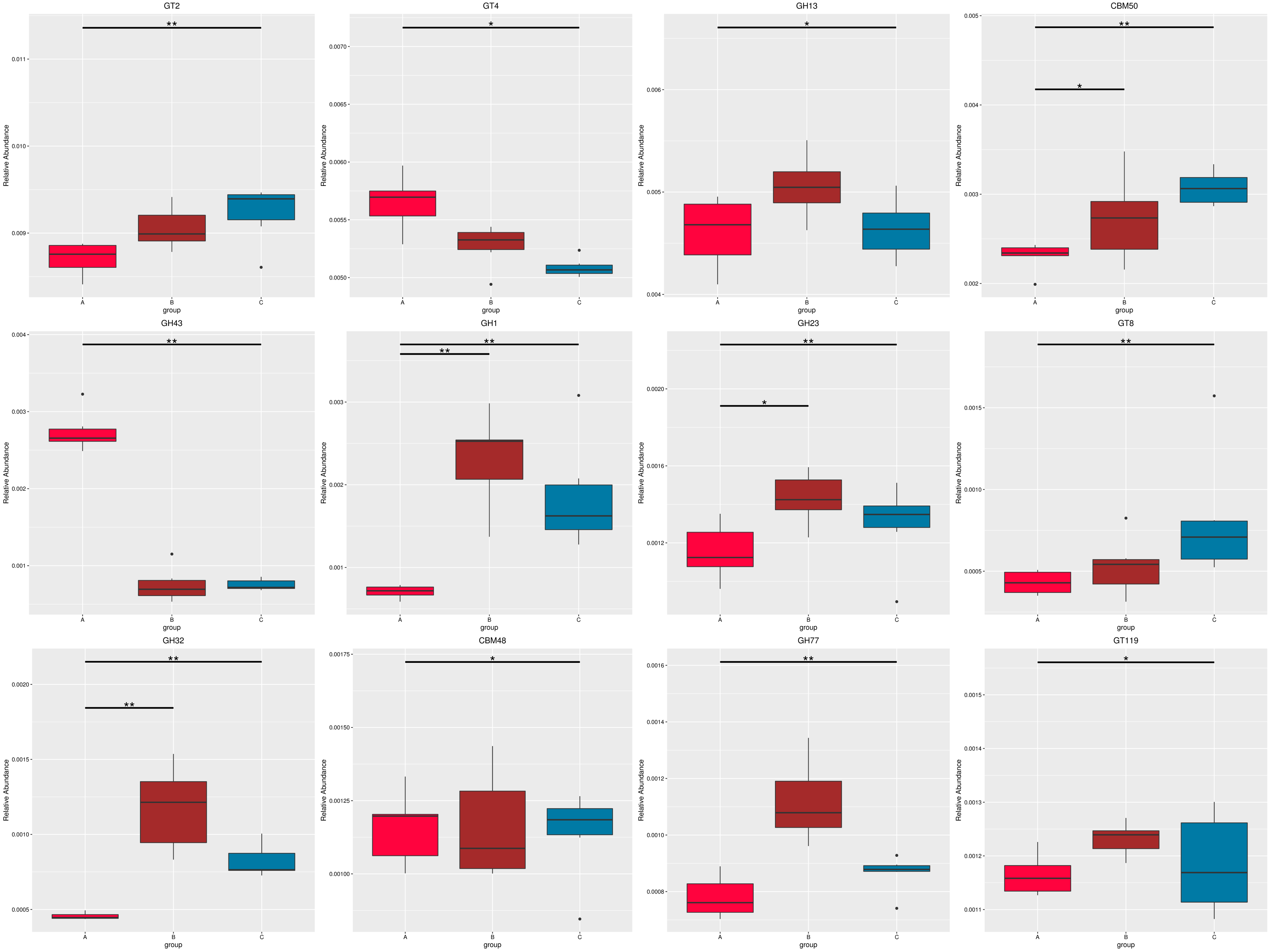
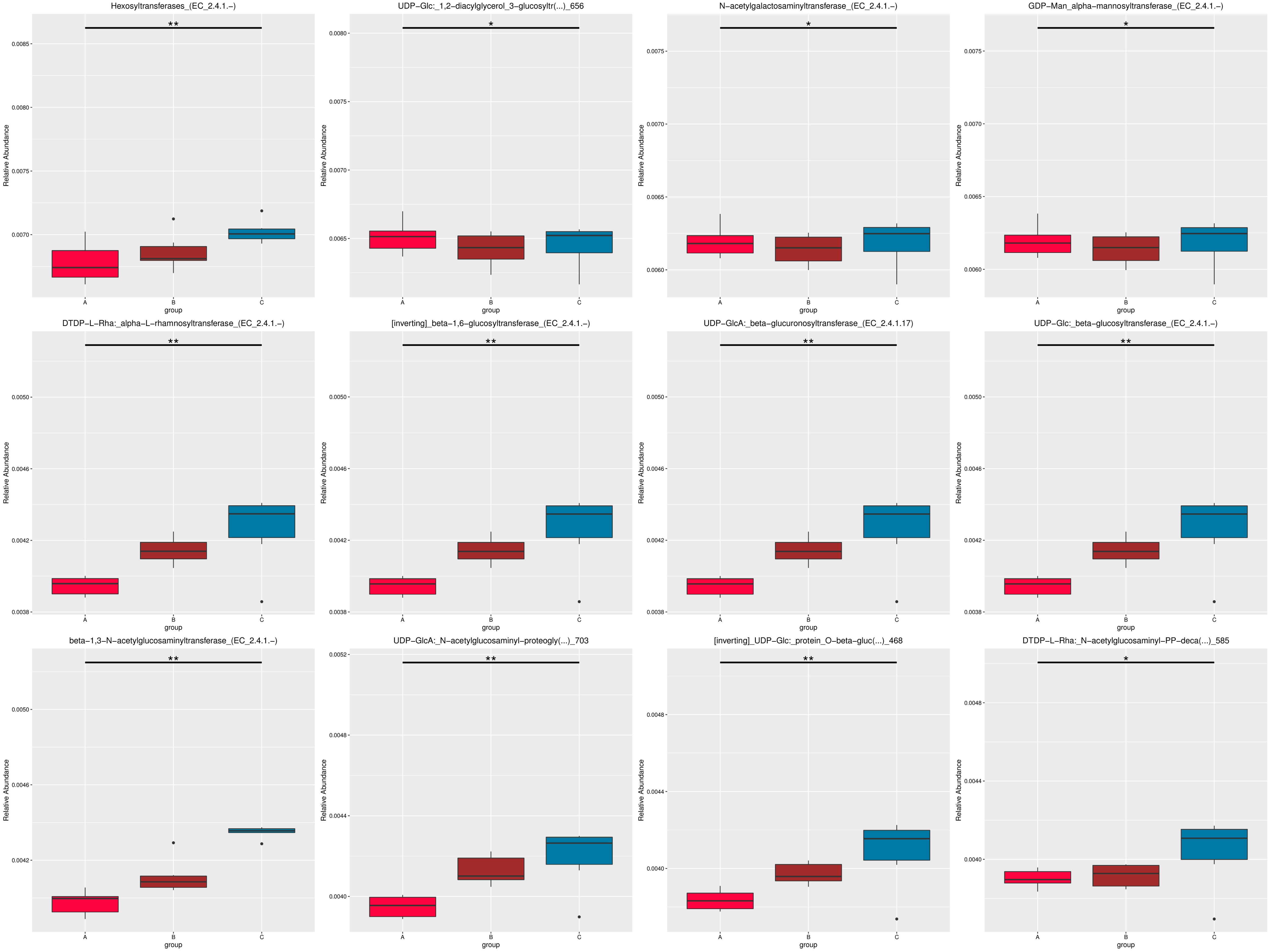
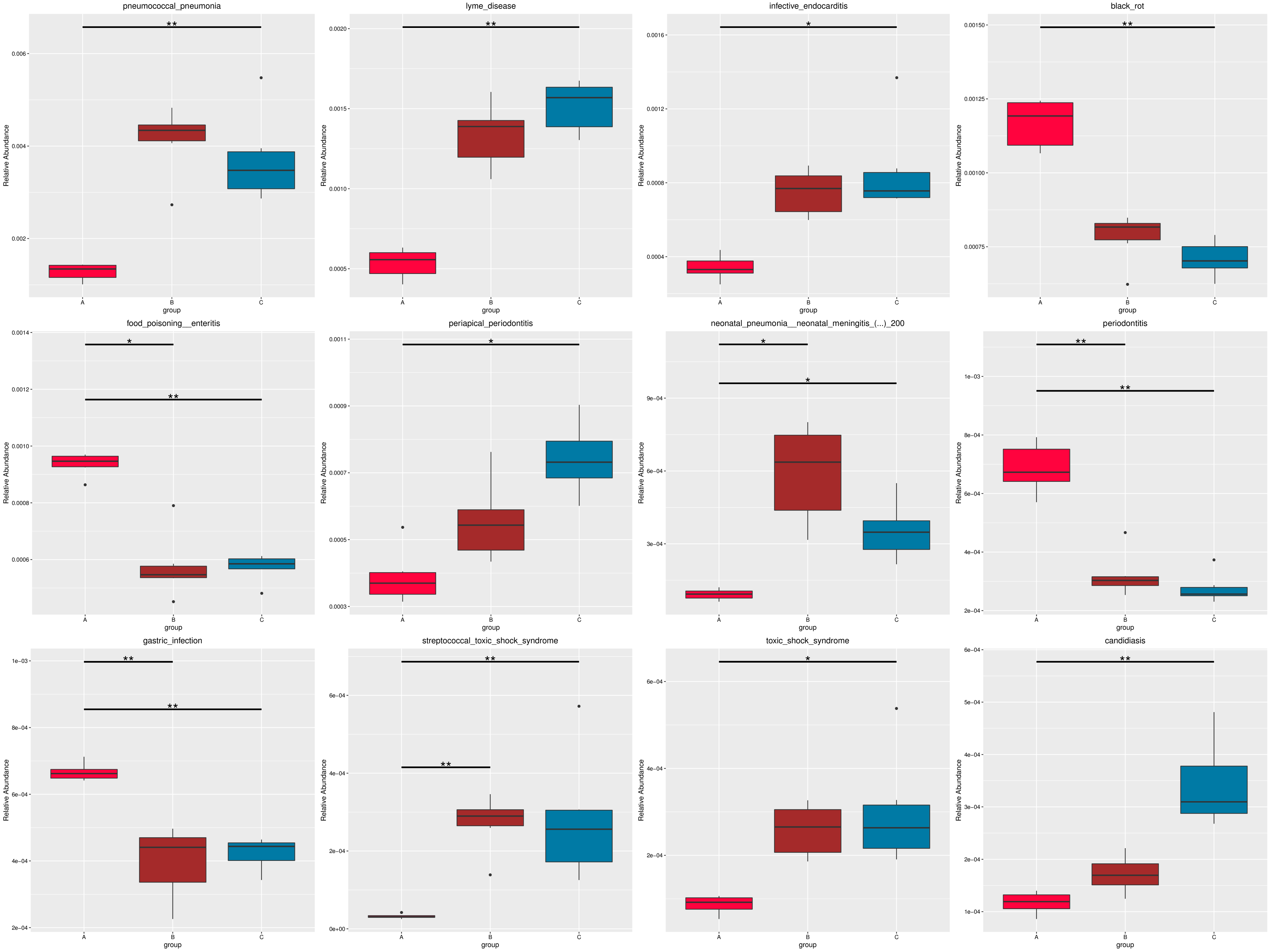
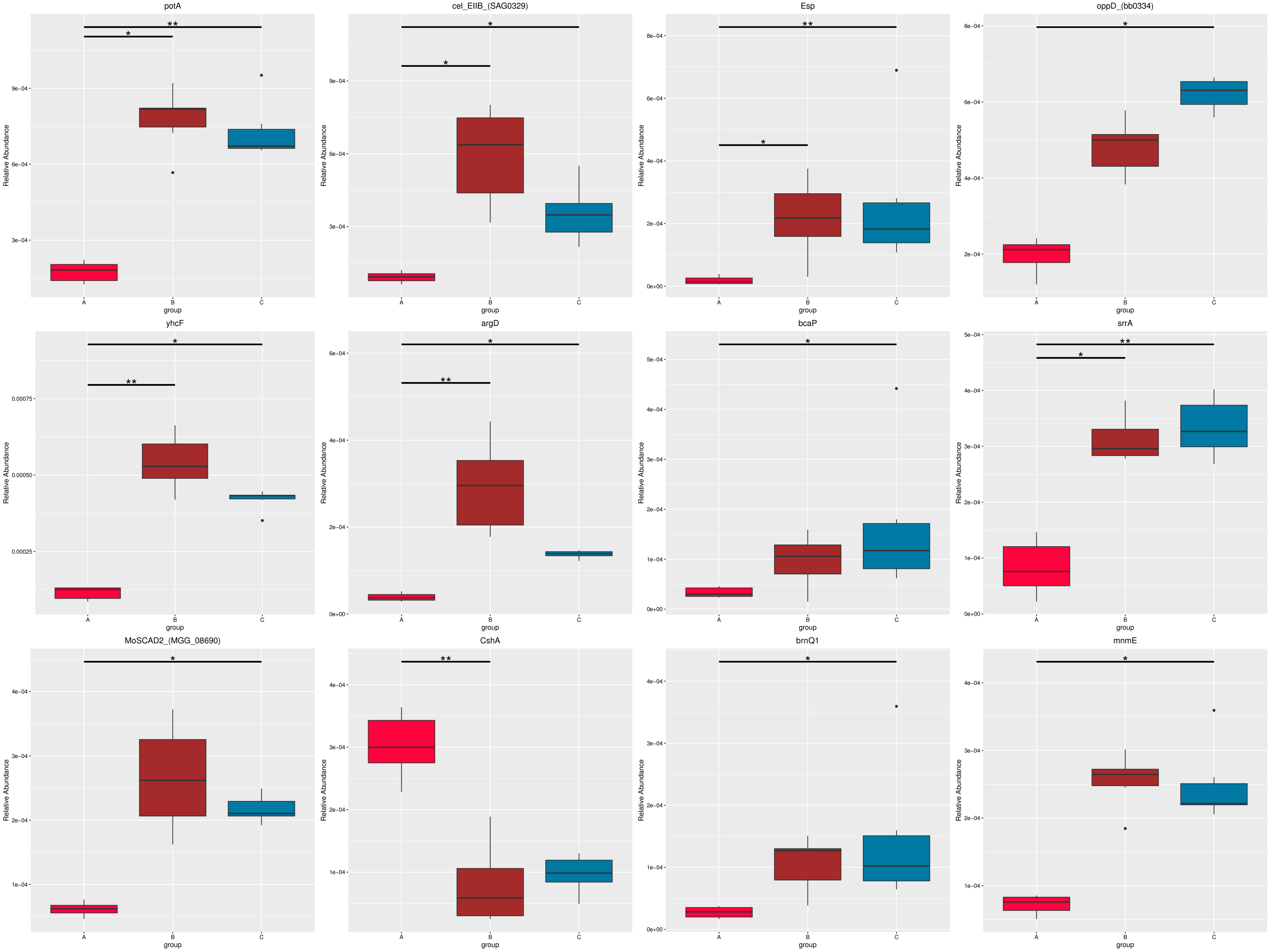
为了研究组间具有显著性差异的物种或功能,从不同层级的物种或功能丰度表出发,利用 MetaGenomeSeq方法对组间的物种或功能丰度数据进行假设检验得到 p 值,通过对 p 值的校正,得到 q 值;最后根据 q 值筛选具有显著性差异的物种或功能,并绘制差异物种或功能在组间的丰度分布箱图。
4.6.2.1 Micro_NR
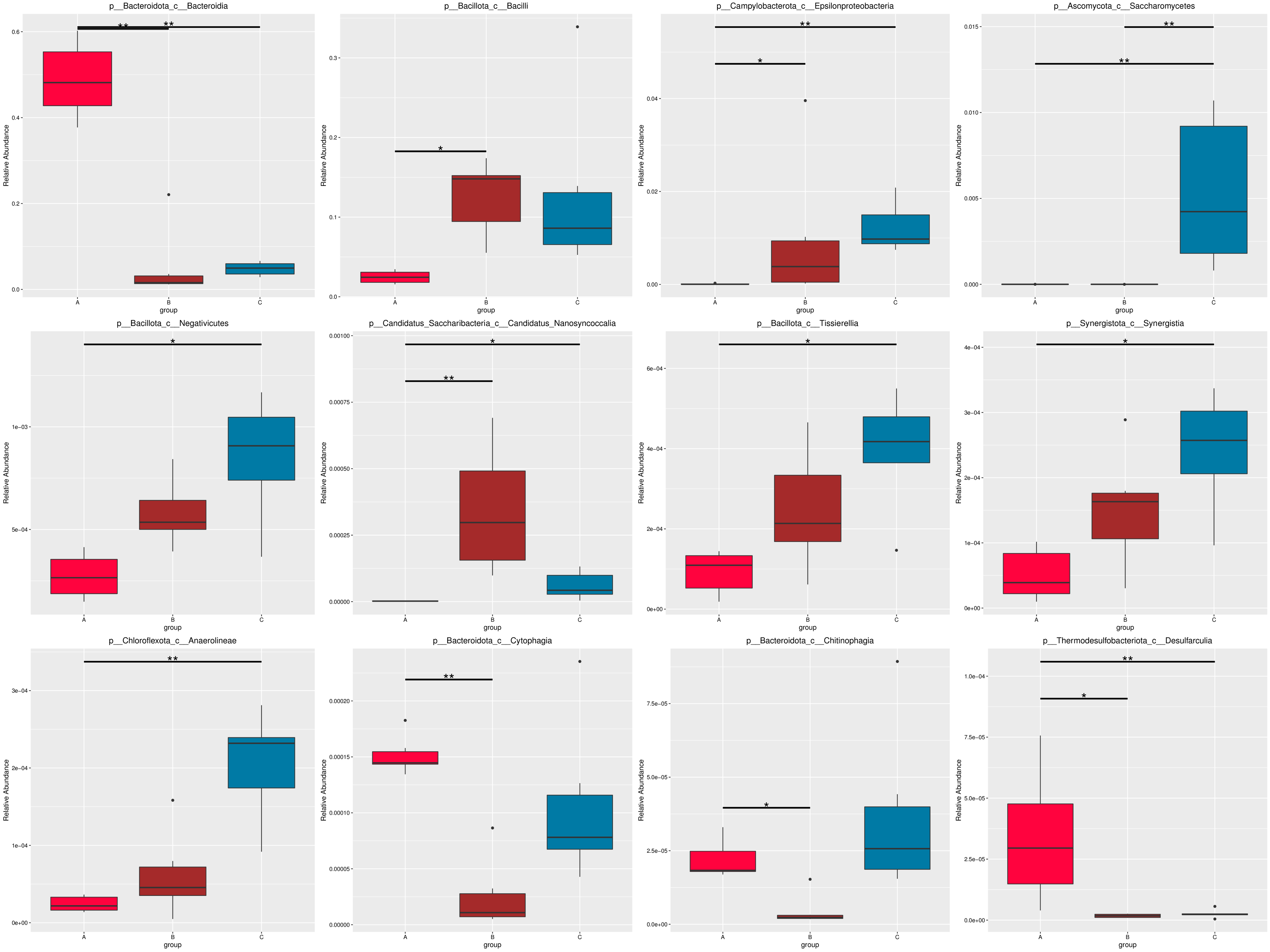
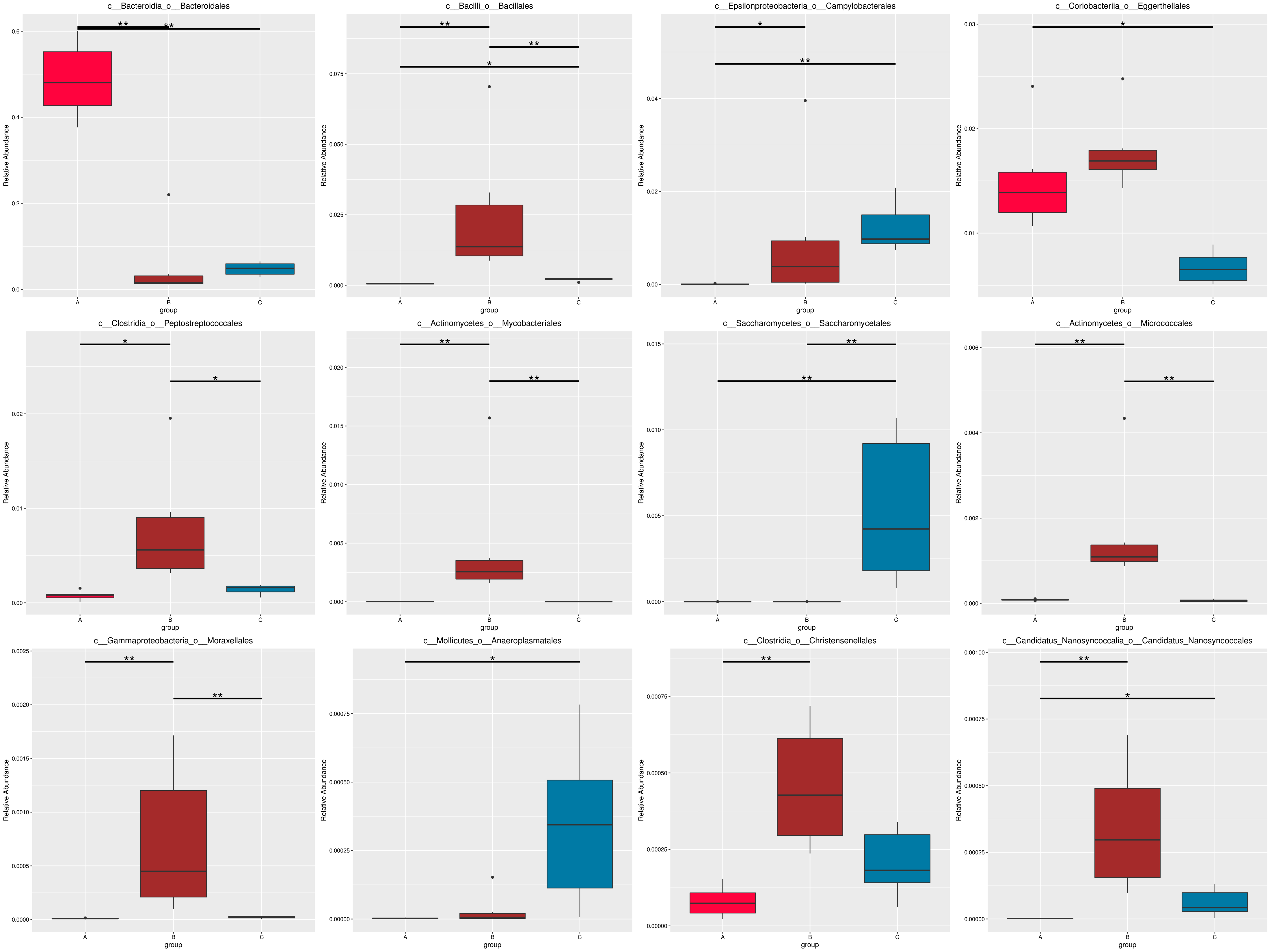
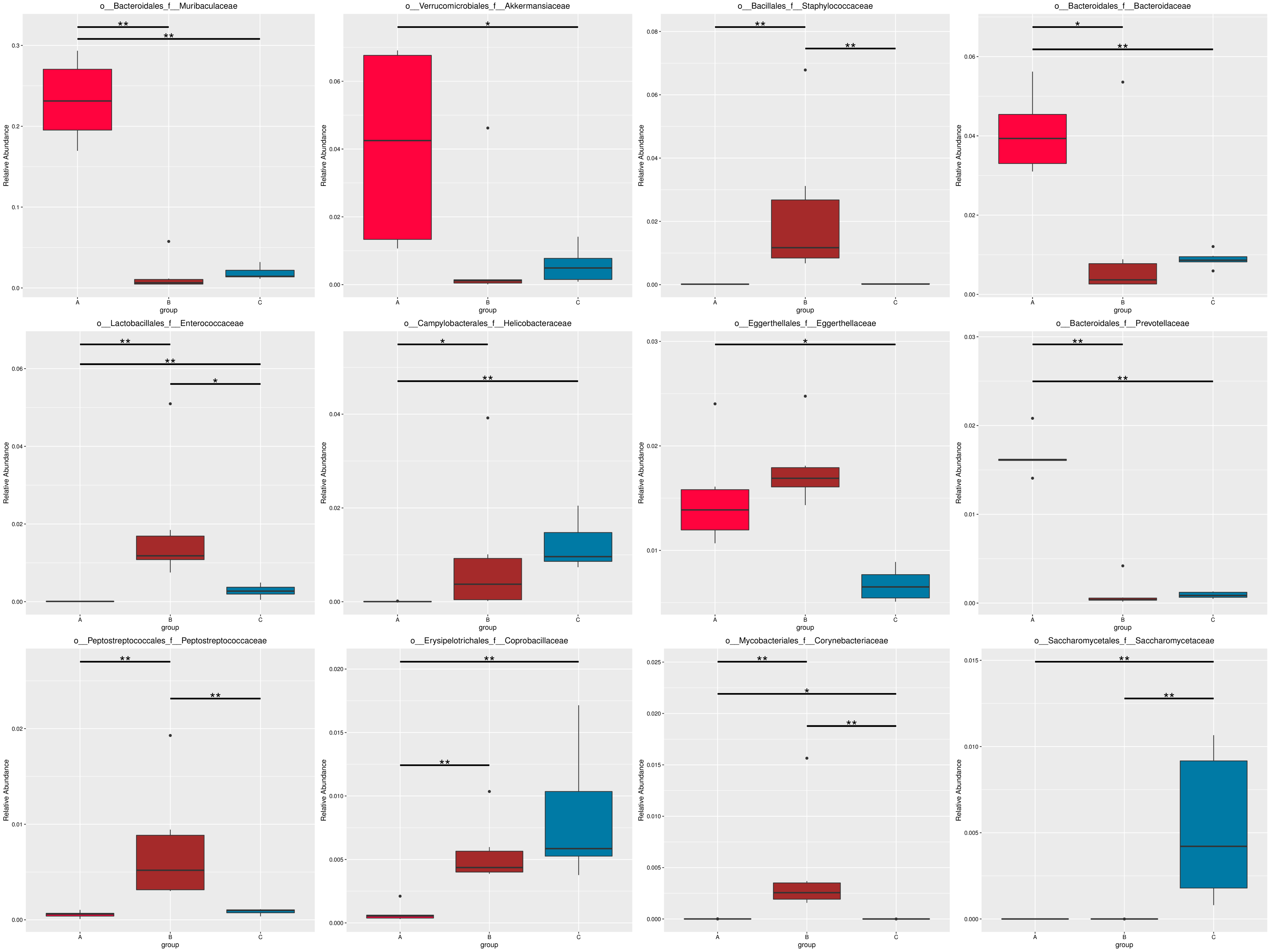
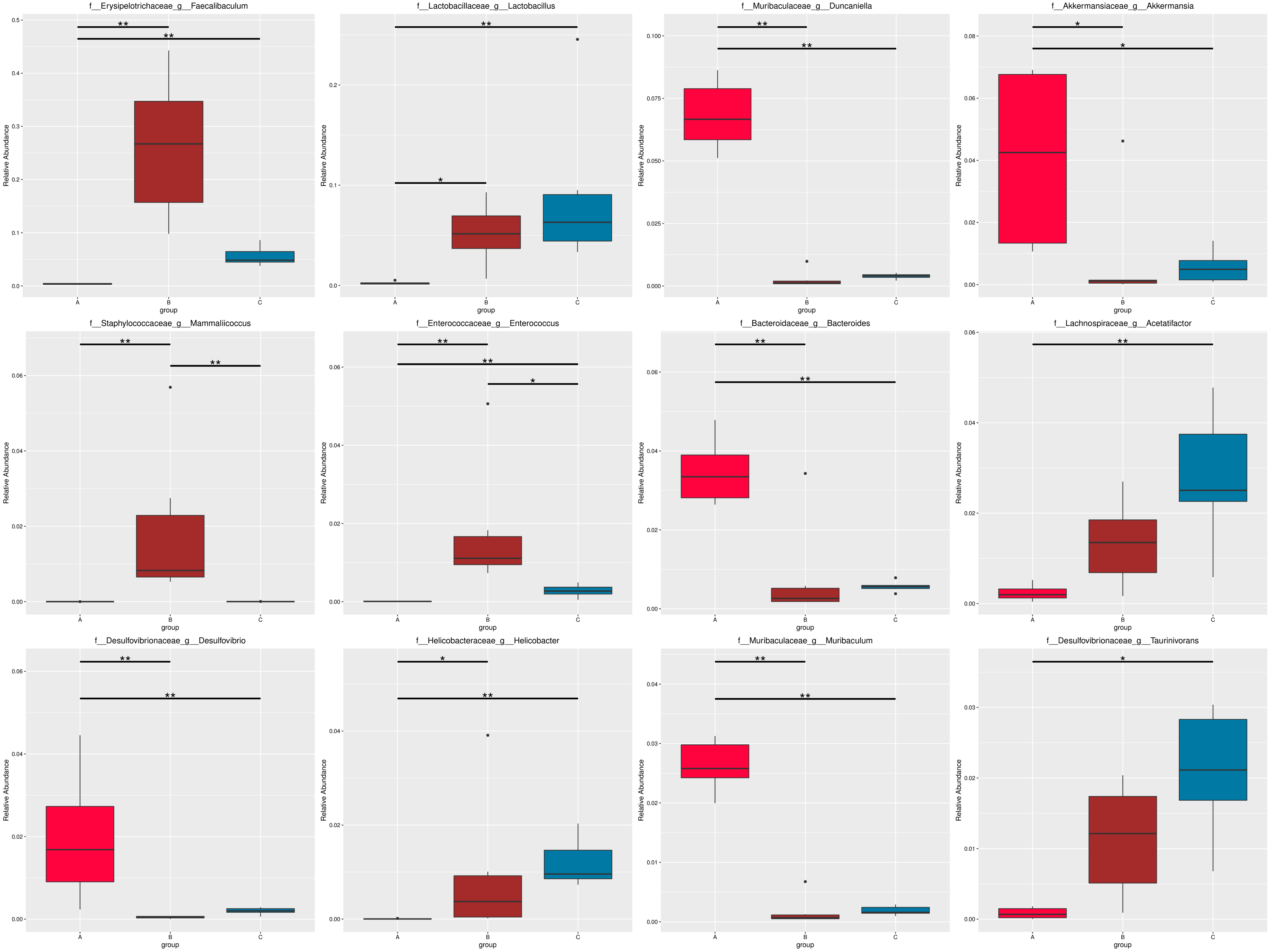
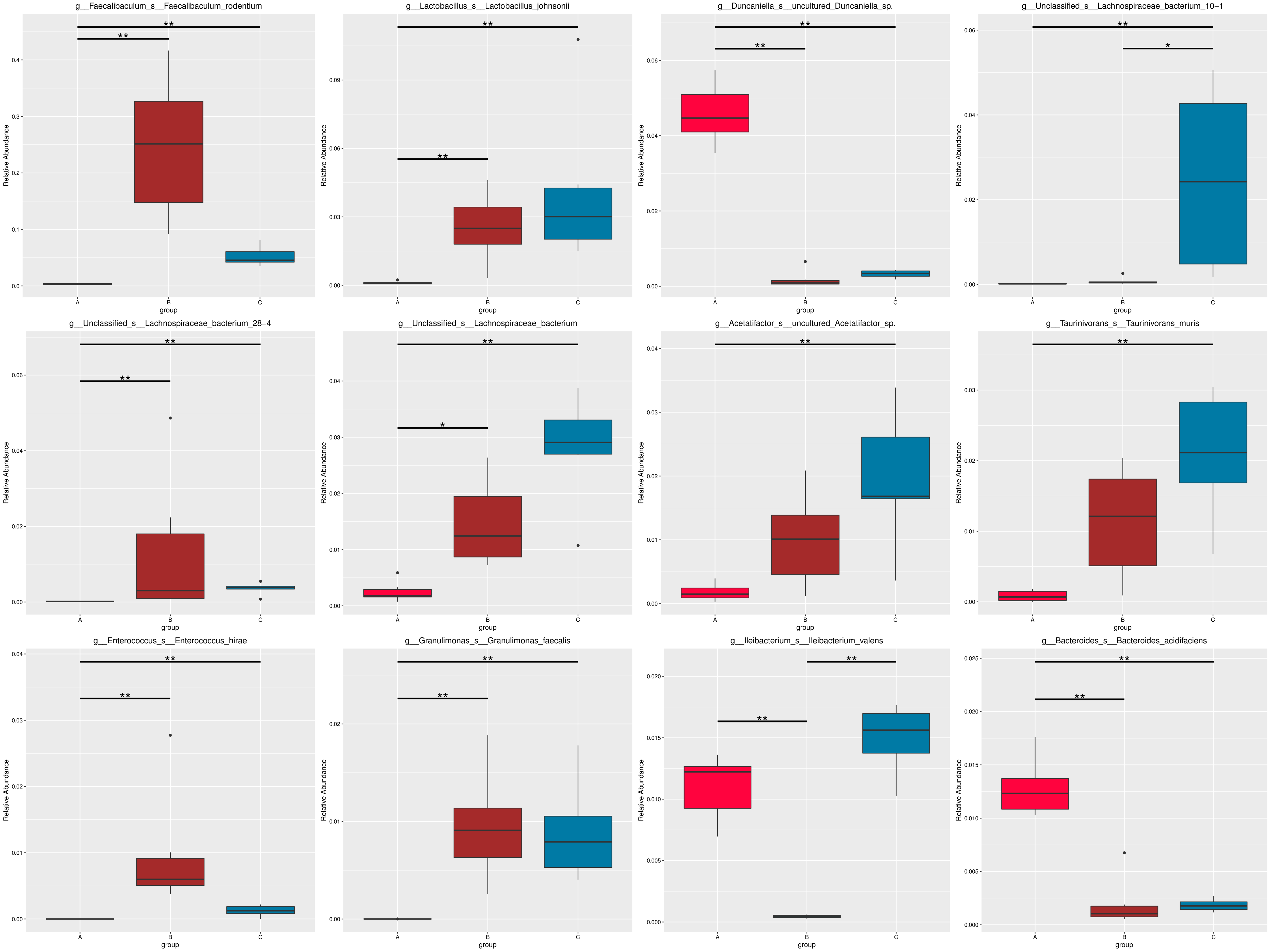
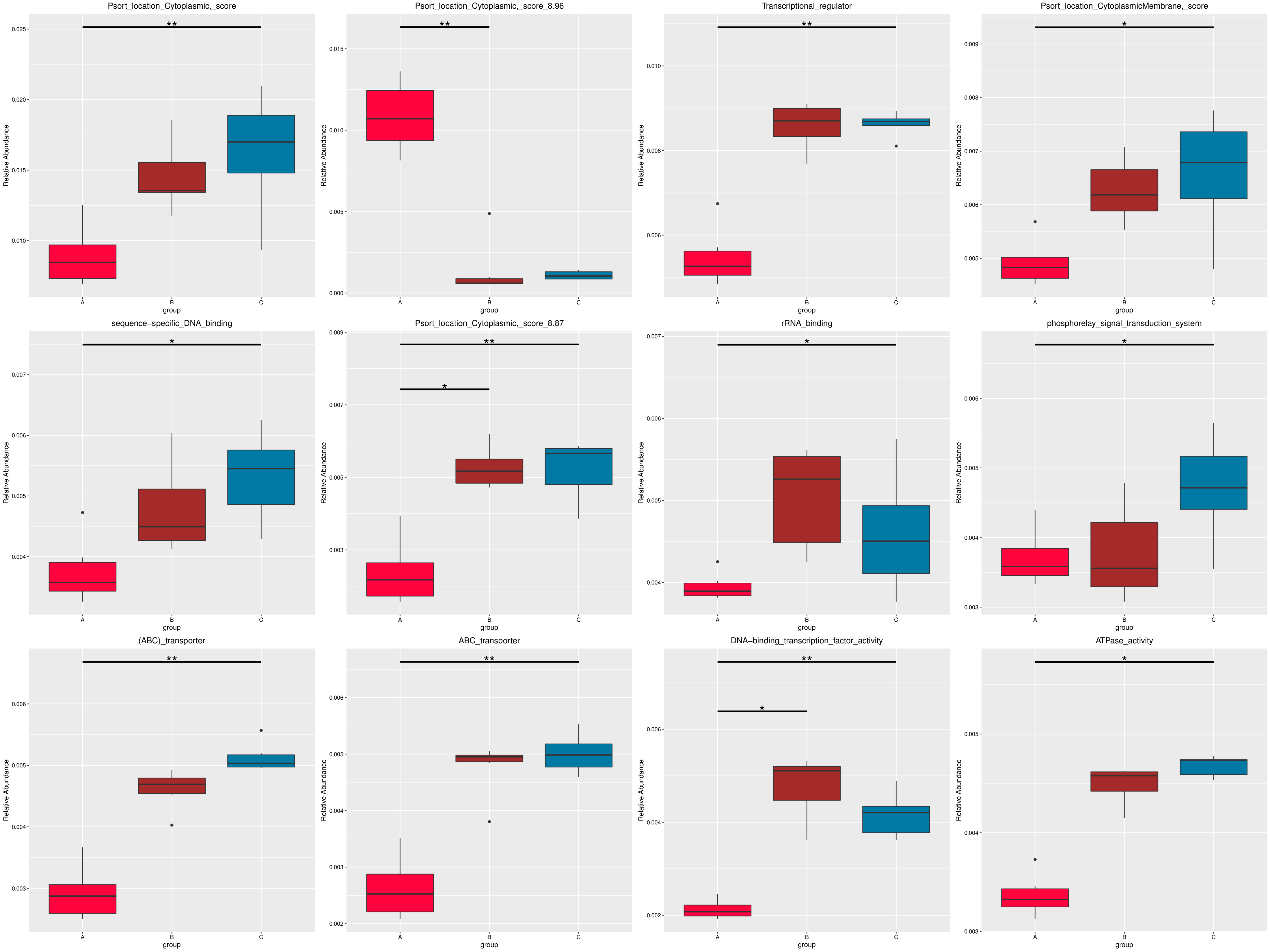
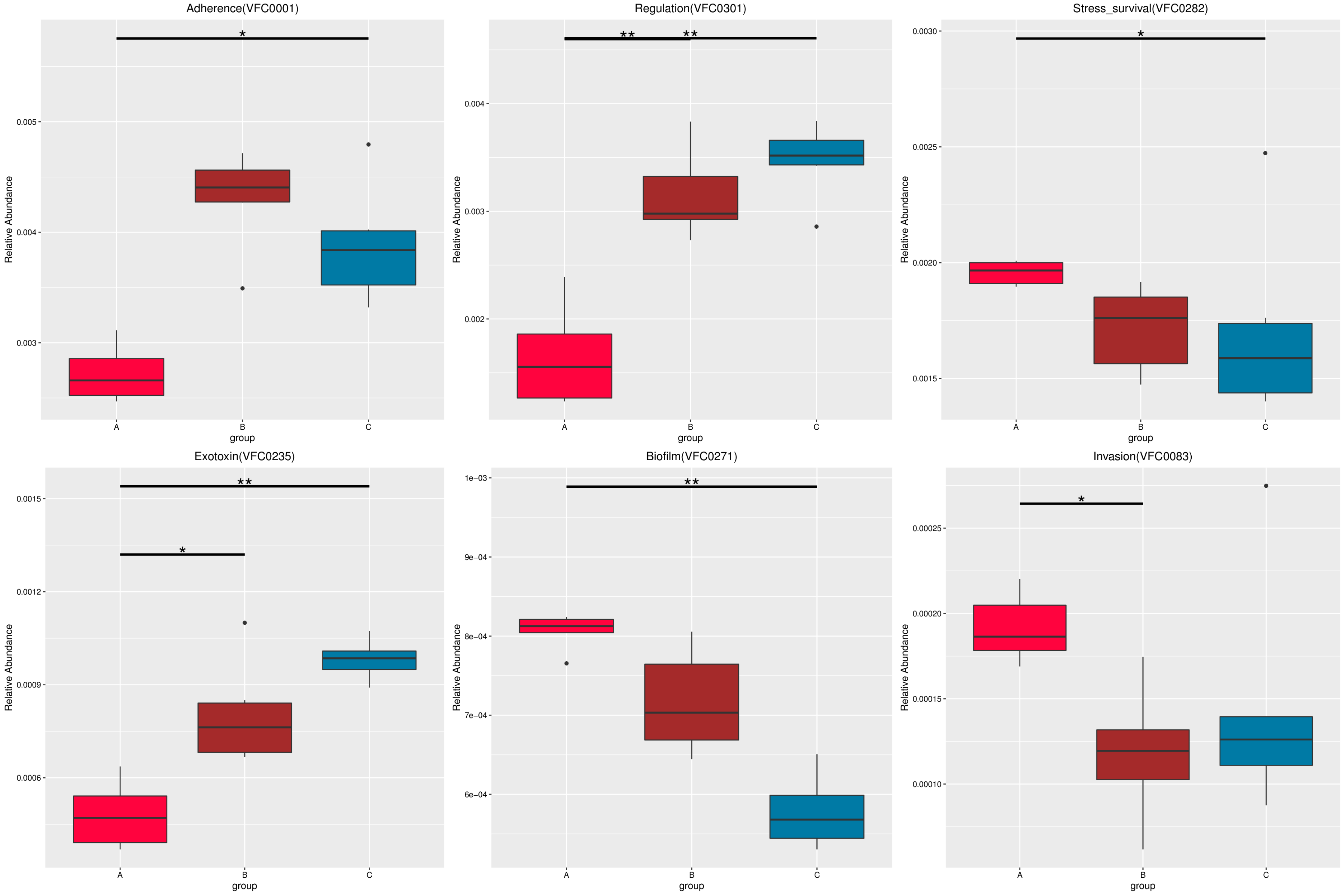
图4.52 基于 Micro_NR 水平的 MetaGenomeSeq 分析
图中,横向为样品分组;纵向为对应物种的相对丰度。横线代表具有显著性差异的两个分组,没有则表示此物种在两个分组间不存在差异。“*”表示两组间差异显著(q value < 0.05),“**”表示两组间差异极显著(q value < 0.01)。
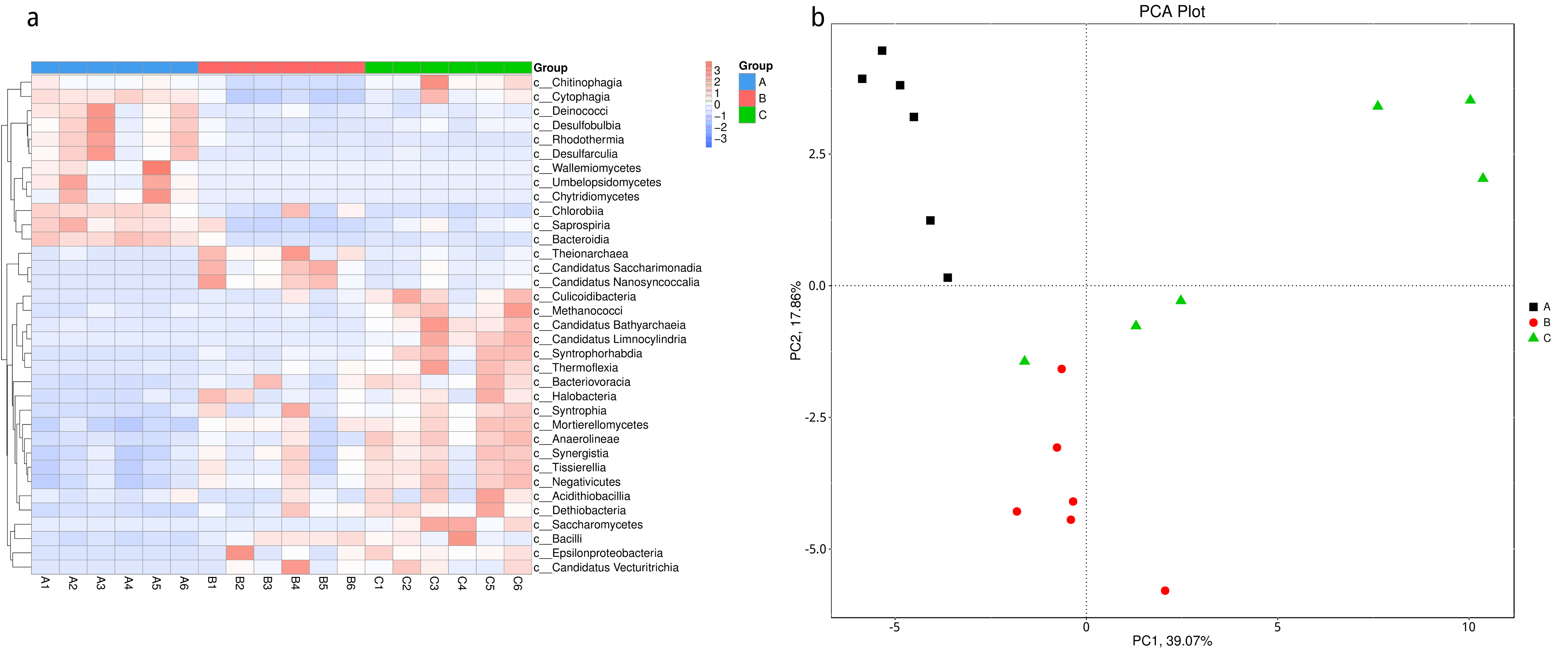
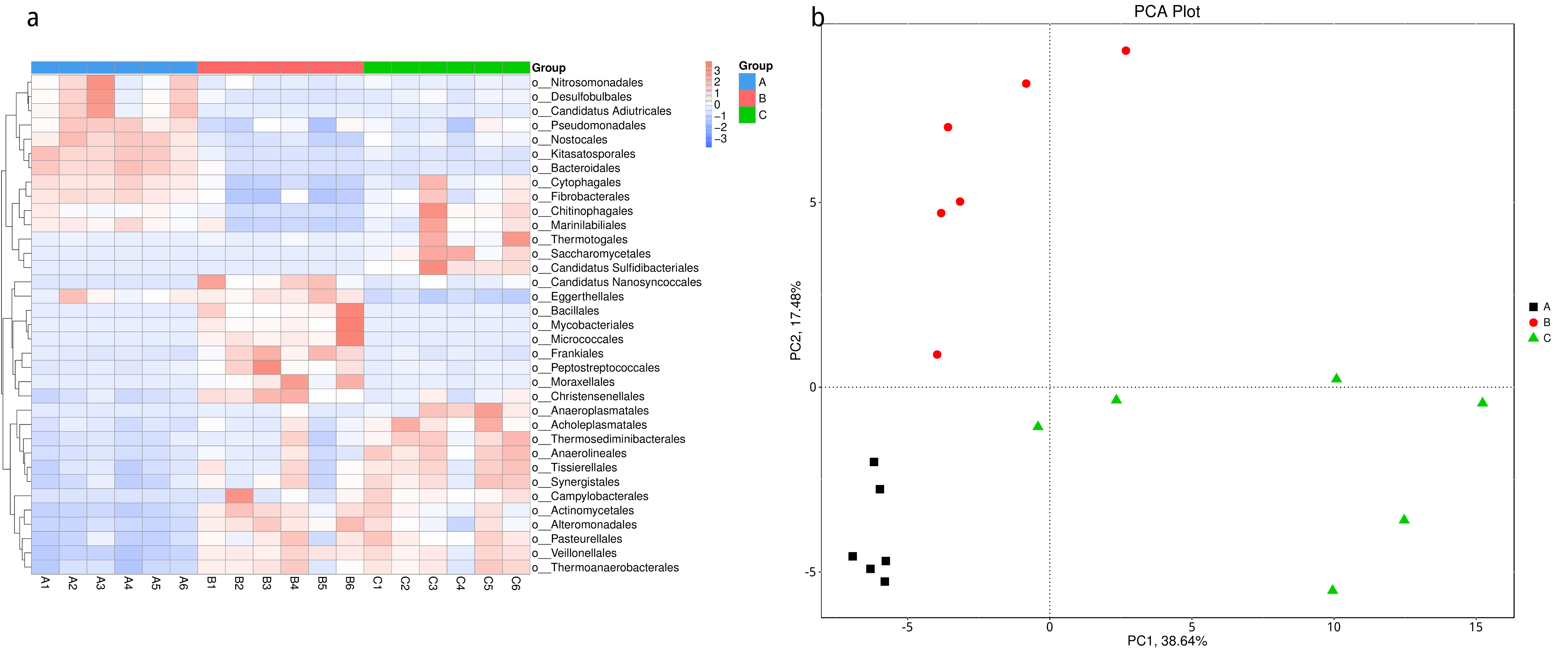
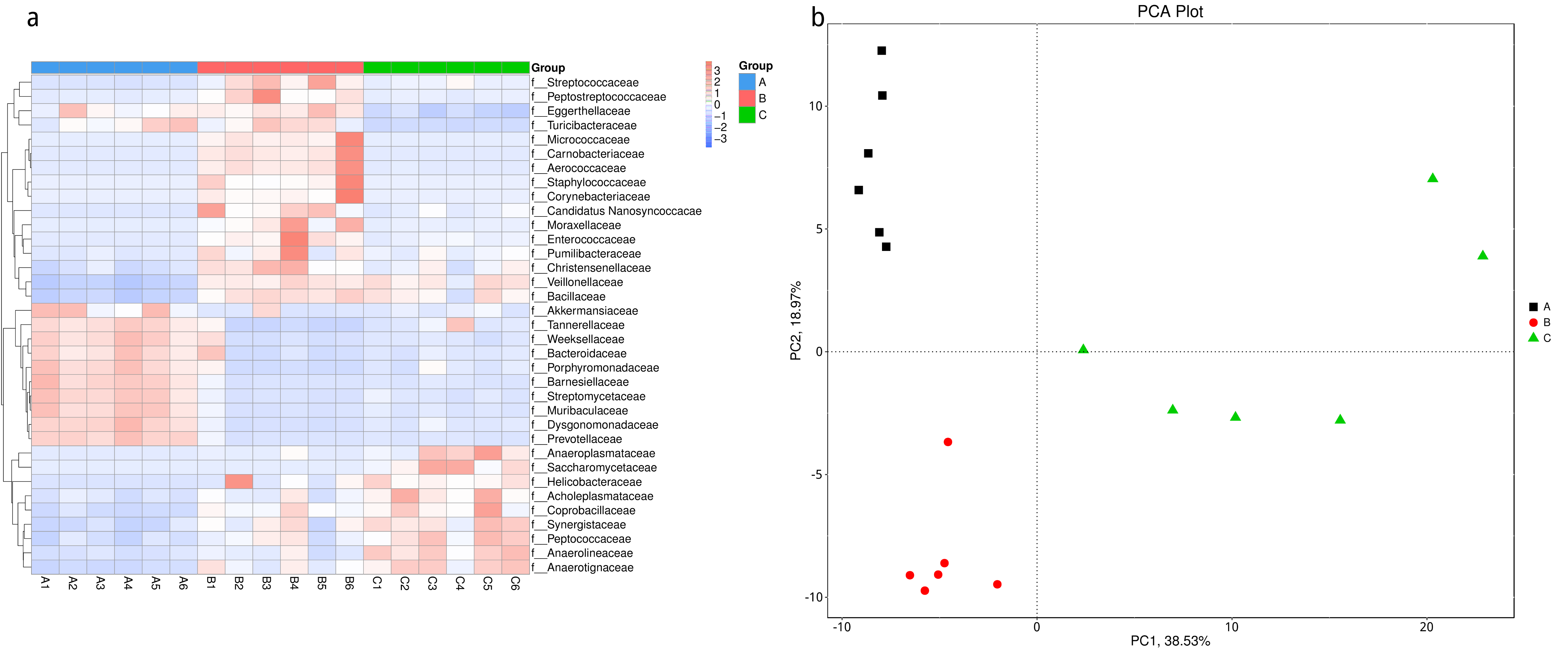
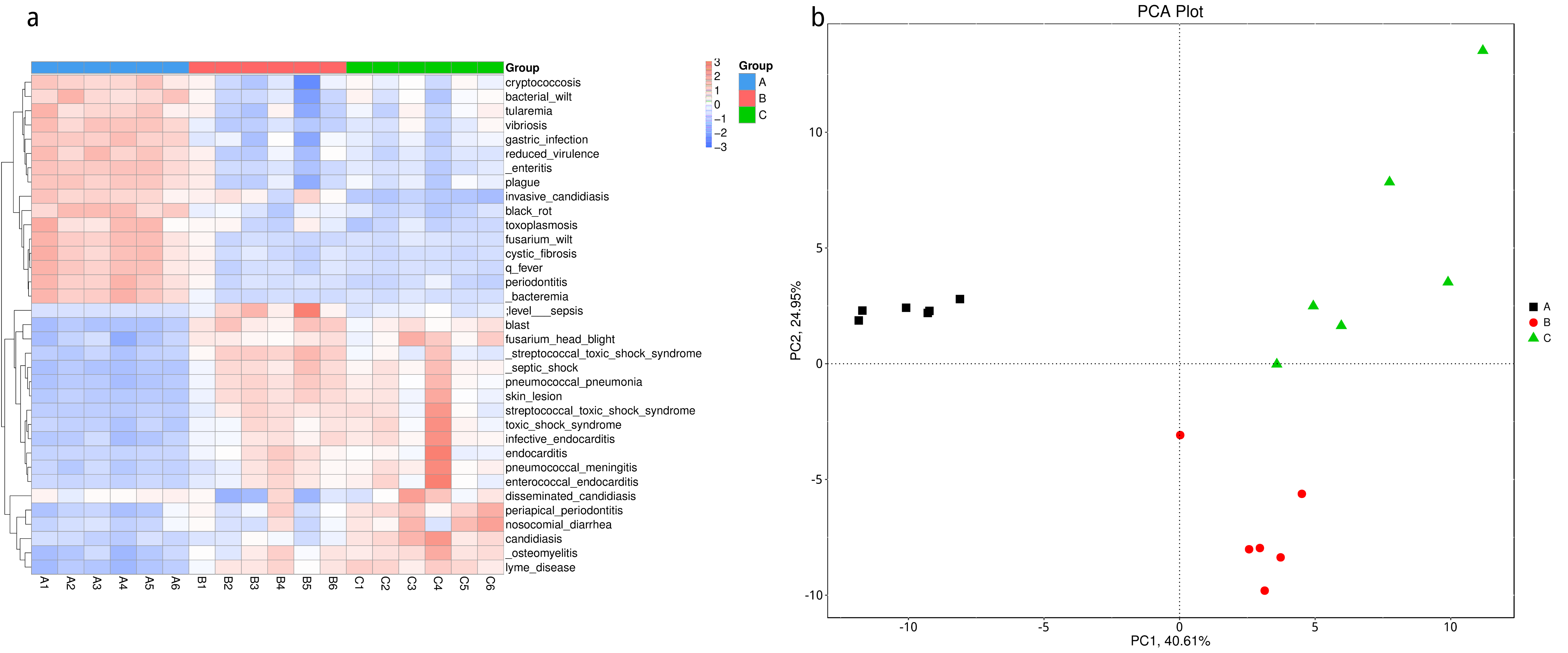
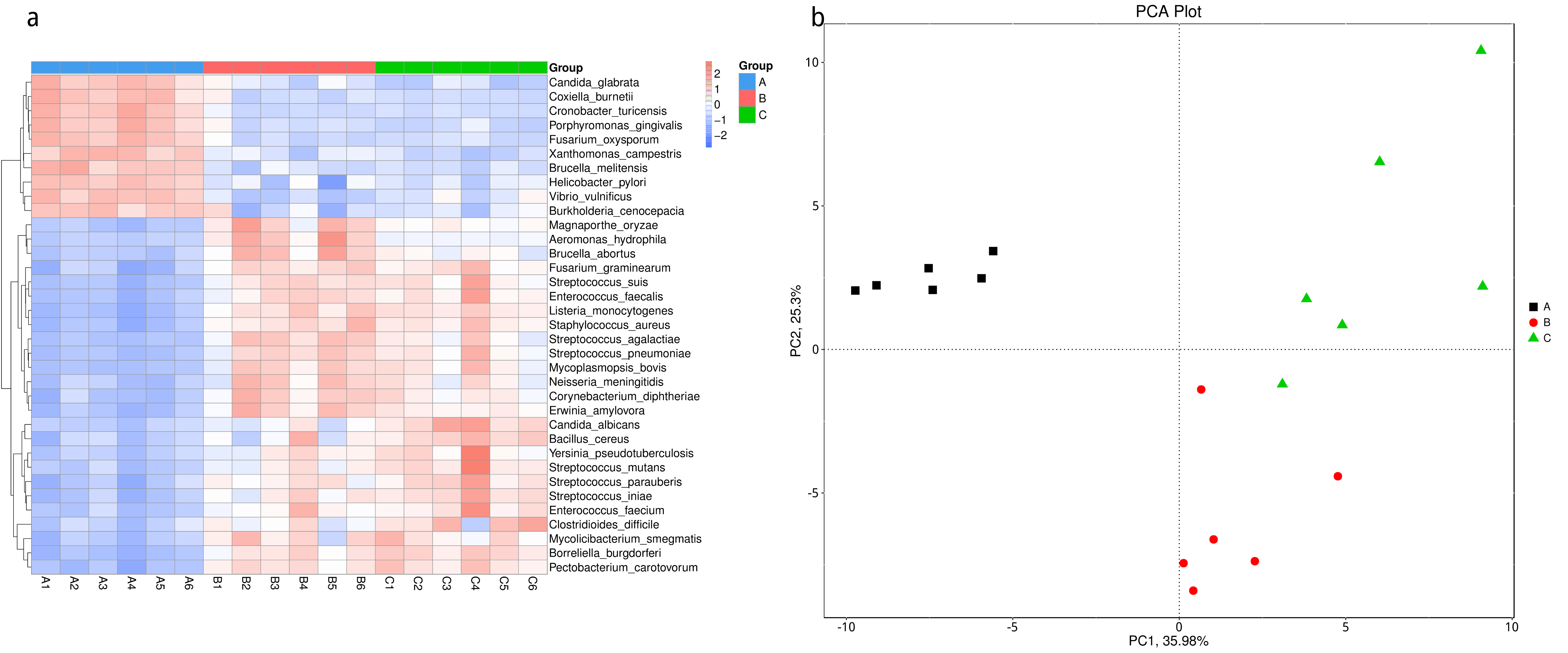
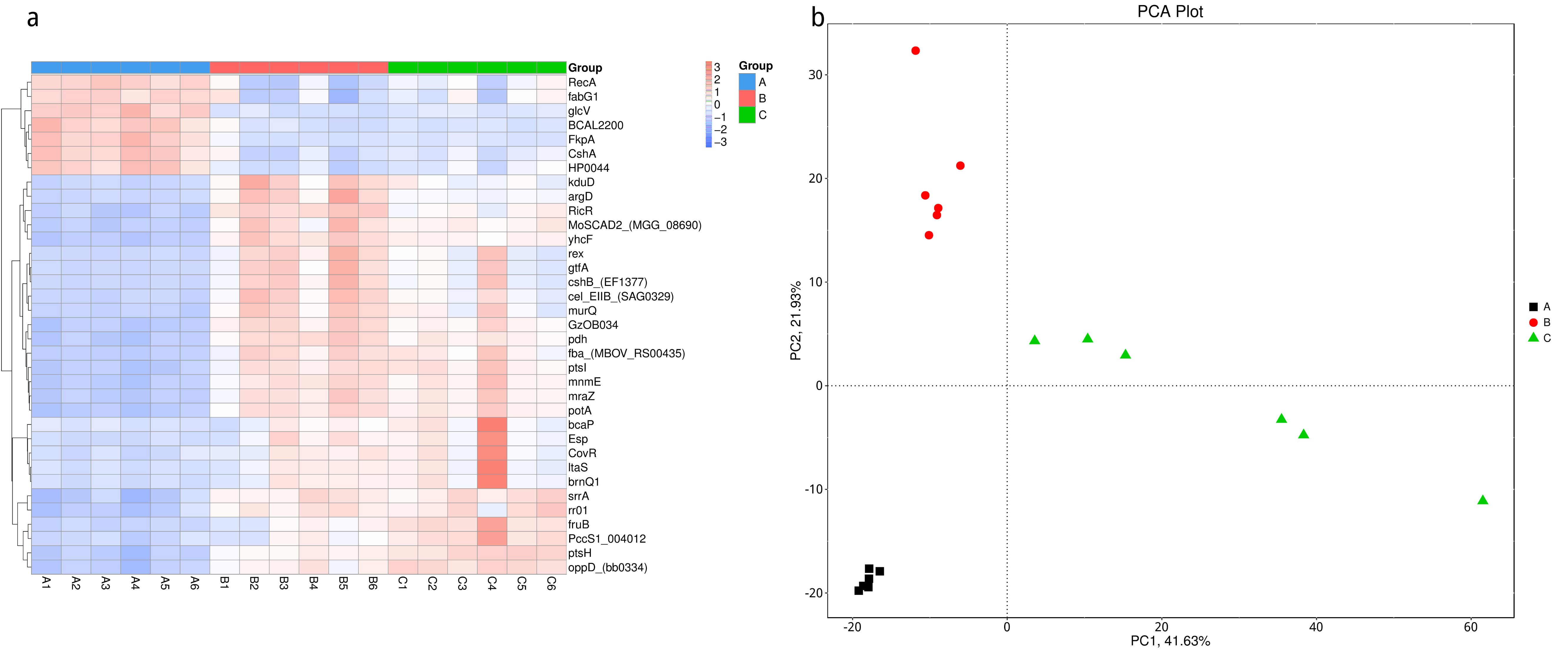
根据组间具有差异的物种进行主成分 PCA 分析和丰度聚类热图分析,展示结果如下::
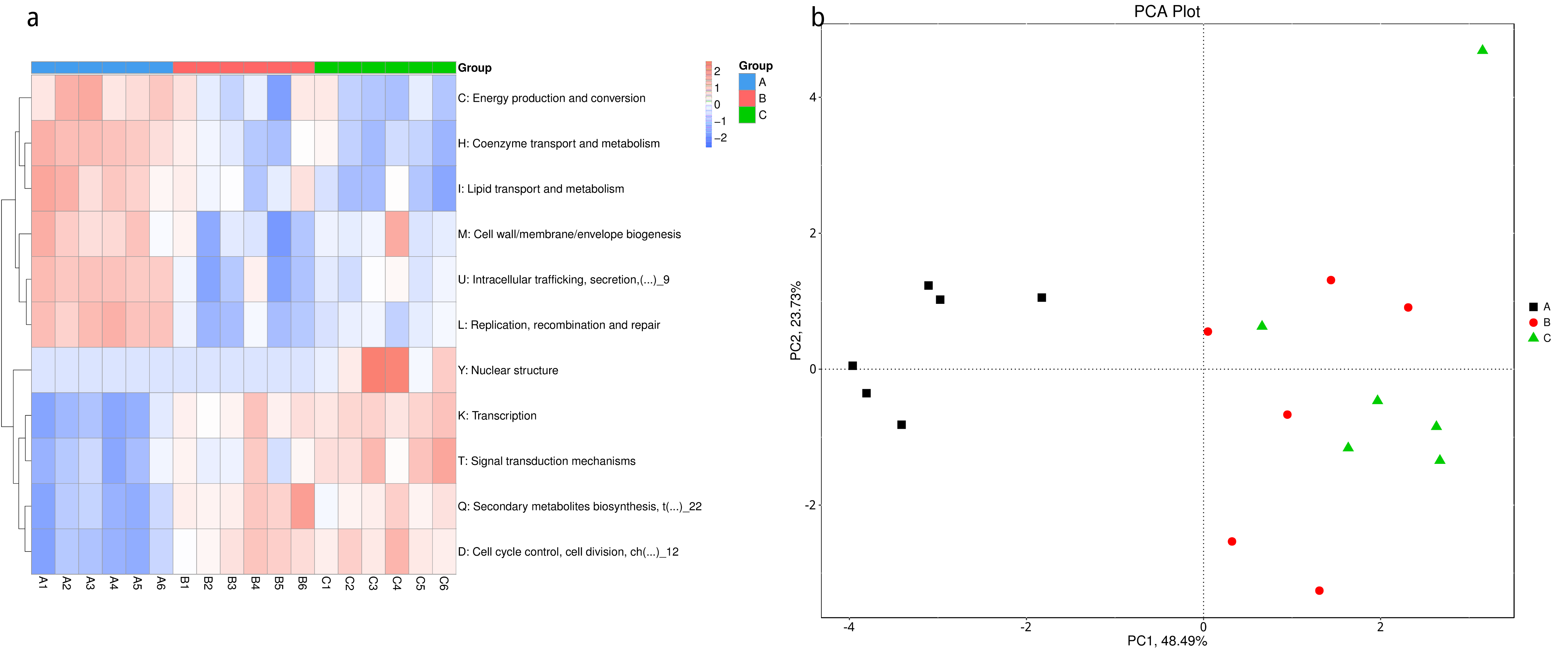
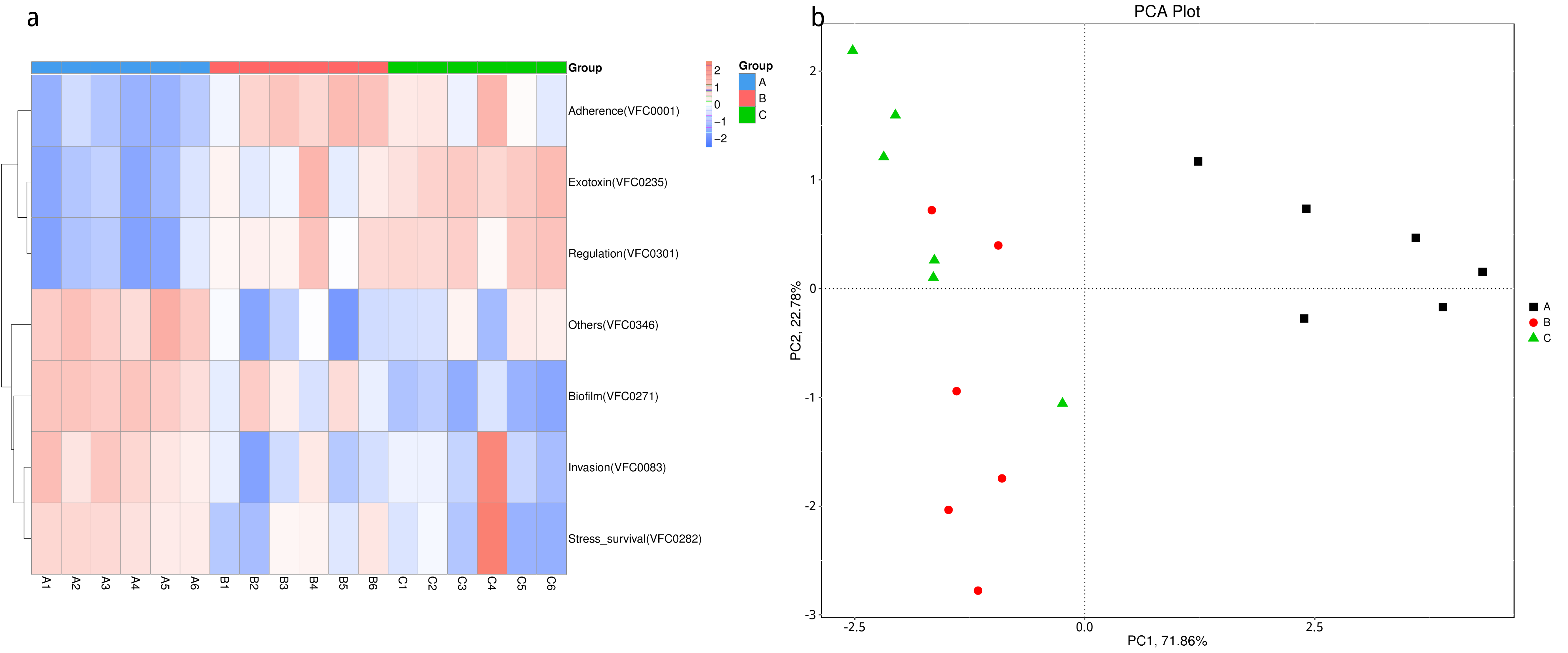
图4.53 基于显著性差异物种的丰度聚类热图和 PCA 分析
a) 为显著性差异物种的丰度聚类热图:横向为样品信息;纵向为物种注释信息;图中左侧的聚类树为物种聚类树;中间热图对应的值为每一行物种相对丰度经过标准化处理后得到的 Z 值;即一个样品在某个分类上的 Z 值为样品在该分类上的相对丰度和所有样品在该分类的平均相对丰度的差除以所有样品在该分类上的标准差所得到的值;b) 为显著性差异物种的 PCA 图:横坐标表示第一主成分,百分比则表示第一主成分对样品差异的贡献值;纵坐标表示第二主成分,百分比表示第二主成分对样品差异的贡献值;图中的每个点表示一个样品,同一个组的样品使用同一种颜色表示。
结果目录 :
各分类层级的 MetaGenomeSeq 分析结果见:result/06.StatisticalTest/MicroNR/MetaGenomeSeq_group1/*
以门水平为例,MetaGenomeSeq 分析结果见 : result/06.StatisticalTest/MicroNR/MetaGenomeSeq_group1/phylum/*.all.xls
从 MetaGenomeSeq 分析结果中,筛选出的 P value<=0.05的信息见 : result/06.StatisticalTest/MicroNR/MetaGenomeSeq_group1/phylum/*.psig.xls
从 MetaGenomeSeq 分析结果中,筛选出的 Q value<=0.05的信息见 : result/06.StatisticalTest/MicroNR/MetaGenomeSeq_group1/phylum/*.qsig.xls
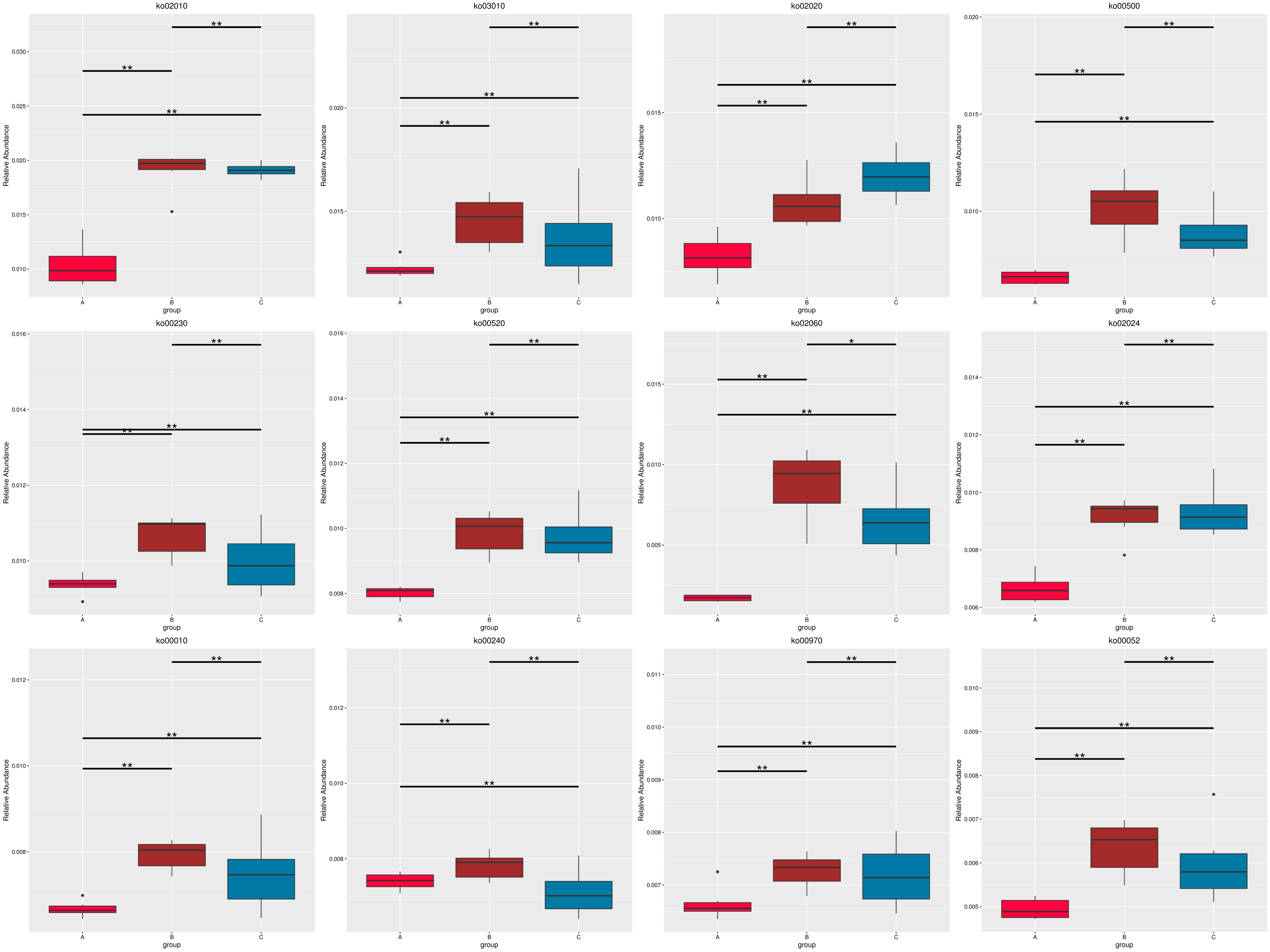
4.6.2.2 KEGG
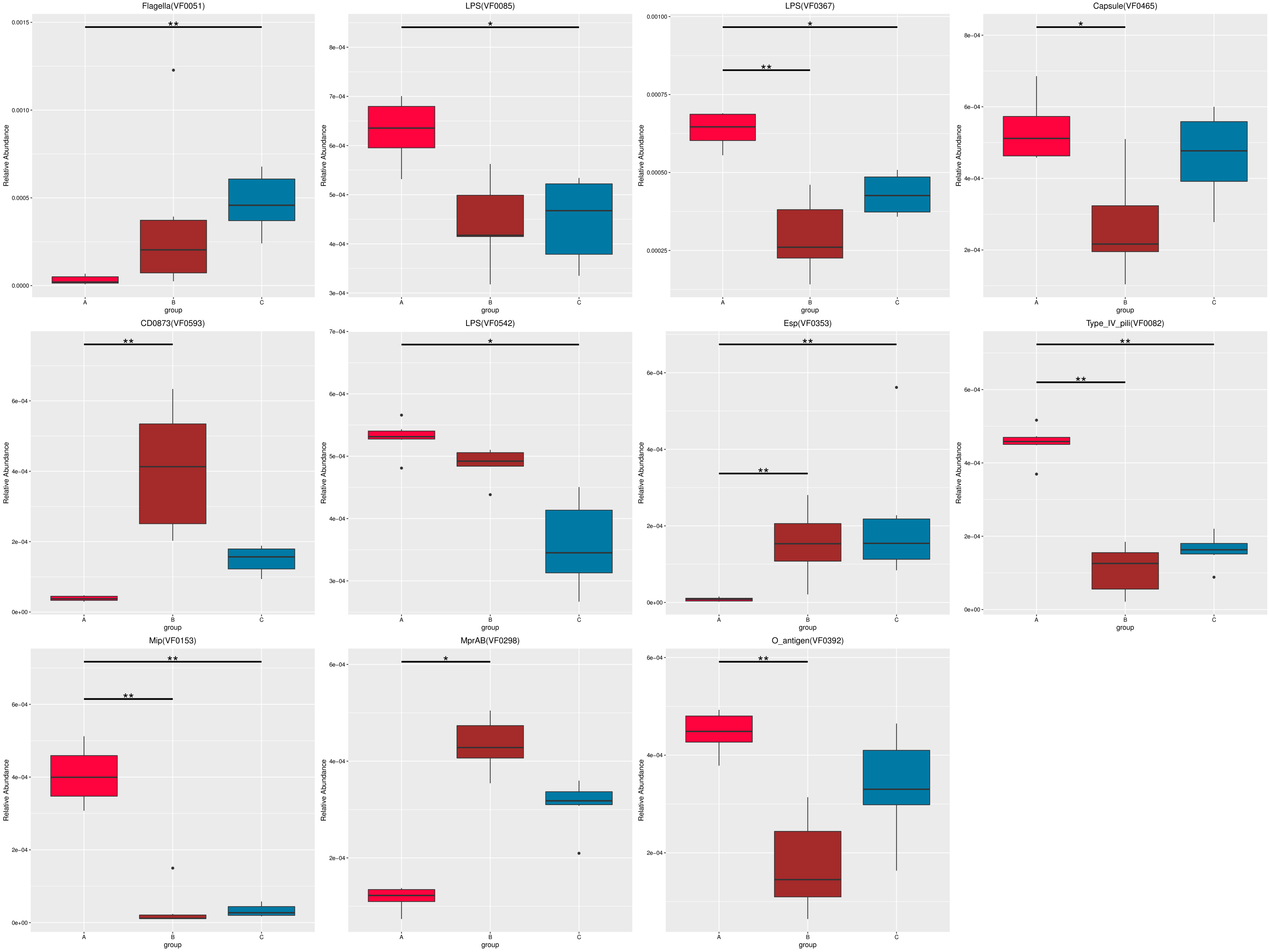
图4.54 基于 KEGG 水平的 MetaGenomeSeq 分析
图中,横向为样品分组;纵向为对应功能的相对丰度。横线代表具有显著性差异的两个分组,没有则表示此功能在两个分组间不存在差异。“*”表示两组间差异显著(q value < 0.05),“**”表示两组间差异极显著(q value < 0.01)。
根据组间具有差异的功能进行主成分 PCA 分析和丰度聚类热图分析,展示结果如下:
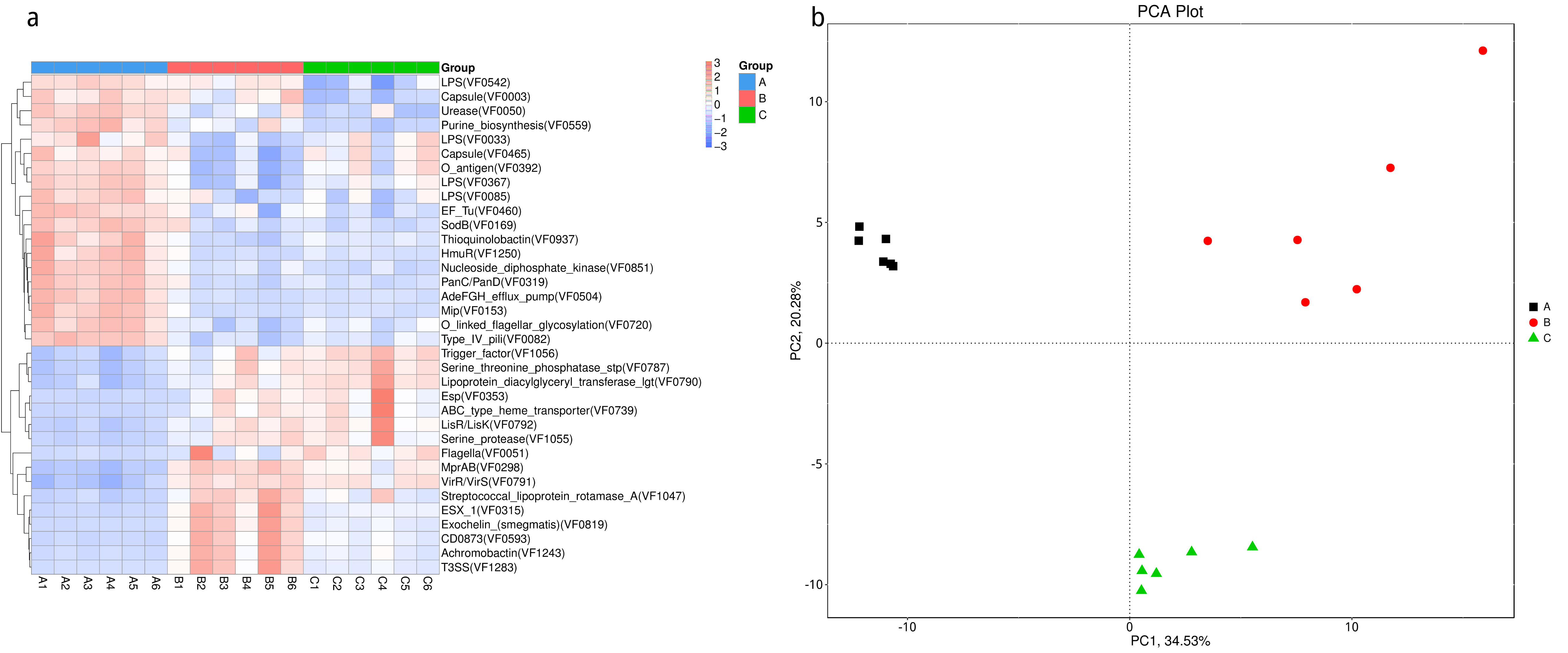
图4.55 基于显著性差异功能的丰度聚类热图和 PCA 分析
a) 为显著性差异功能的丰度聚类热图:横向为样品信息;纵向为功能注释信息;图中左侧的聚类树为功能聚类树;中间热图对应的值为每一行功能相对丰度经过标准化处理后得到的 Z 值;即一个样品在某个分类上的 Z 值为样品在该分类上的相对丰度和所有样品在该分类的平均相对丰度的差除以所有样品在该分类上的标准差所得到的值;b) 为显著性差异功能的 PCA 图:横坐标表示第一主成分,百分比则表示第一主成分对样品差异的贡献值;纵坐标表示第二主成分,百分比表示第二主成分对样品差异的贡献值;图中的每个点表示一个样品,同一个组的样品使用同一种颜色表示。
结果目录 :
各分类层级的 MetaGenomeSeq 分析结果见 : result/06.StatisticalTest/KEGG/MetaGenomeSeq_group1/*
以 ko 水平为例,MetaGenomeSeq 分析结果见 : result/06.StatisticalTest/KEGG/MetaGenomeSeq_group1/ko/*.all.xls
从 MetaGenomeSeq 分析结果中,筛选出的 P value<=0.05的信息见 : result/06.StatisticalTest/KEGG/MetaGenomeSeq_group1/ko/*.psig.xls
从 MetaGenomeSeq 分析结果中,筛选出的 Q value<=0.05的信息见 : result/06.StatisticalTest/KEGG/MetaGenomeSeq_group1/ko/*.qsig.xls
4.6.2.3 eggNOG
图4.56 基于 eggNOG 水平的 MetaGenomeSeq 分析
图中,横向为样品分组;纵向为对应功能的相对丰度。横线代表具有显著性差异的两个分组,没有则表示此功能在两个分组间不存在差异。“*”表示两组间差异显著(q value < 0.05),“**”表示两组间差异极显著(q value < 0.01)。
根据组间具有差异的功能进行主成分 PCA 分析和丰度聚类热图分析,展示结果如下:
图4.57 基于显著性差异功能的丰度聚类热图和 PCA 分析
a) 为显著性差异功能的丰度聚类热图:横向为样品信息;纵向为功能注释信息;图中左侧的聚类树为功能聚类树;中间热图对应的值为每一行功能相对丰度经过标准化处理后得到的 Z 值;即一个样品在某个分类上的 Z 值为样品在该分类上的相对丰度和所有样品在该分类的平均相对丰度的差除以所有样品在该分类上的标准差所得到的值;b) 为显著性差异功能的 PCA 图:横坐标表示第一主成分,百分比则表示第一主成分对样品差异的贡献值;纵坐标表示第二主成分,百分比表示第二主成分对样品差异的贡献值;图中的每个点表示一个样品,同一个组的样品使用同一种颜色表示。
结果目录 :
各分类层级的 MetaGenomeSeq 分析结果见 : result/06.StatisticalTest/eggNOG/MetaGenomeSeq_group1/*
以 level2 水平为例,MetaGenomeSeq 分析结果见 : result/06.StatisticalTest/eggNOG/MetaGenomeSeq_group1/level2/*.all.xls
从 MetaGenomeSeq 分析结果中,筛选出的 P value<=0.05的信息见 : result/06.StatisticalTest/eggNOG/MetaGenomeSeq_group1/level2/*.psig.xls
从 MetaGenomeSeq 分析结果中,筛选出的 Q value<=0.05的信息见 : result/06.StatisticalTest/eggNOG/MetaGenomeSeq_group1/level2/*.qsig.xls
4.6.2.4 CAZy
图4.58 基于 CAZy 水平的 MetaGenomeSeq 分析
图中,横向为样品分组;纵向为对应功能的相对丰度。横线代表具有显著性差异的两个分组,没有则表示此功能在两个分组间不存在差异。“*”表示两组间差异显著(q value < 0.05),“**”表示两组间差异极显著(q value < 0.01)。
根据组间具有差异的功能进行主成分 PCA 分析和丰度聚类热图分析,展示结果如下:
图4.59 基于显著性差异功能的丰度聚类热图和 PCA 分析
a) 为显著性差异功能的丰度聚类热图:横向为样品信息;纵向为功能注释信息;图中左侧的聚类树为功能聚类树;中间热图对应的值为每一行功能相对丰度经过标准化处理后得到的 Z 值;即一个样品在某个分类上的 Z 值为样品在该分类上的相对丰度和所有样品在该分类的平均相对丰度的差除以所有样品在该分类上的标准差所得到的值;b) 为显著性差异功能的 PCA 图:横坐标表示第一主成分,百分比则表示第一主成分对样品差异的贡献值;纵坐标表示第二主成分,百分比表示第二主成分对样品差异的贡献值;图中的每个点表示一个样品,同一个组的样品使用同一种颜色表示。
结果目录 :
各分类层级的 MetaGenomeSeq 分析结果见 : result/06.StatisticalTest/CAZy/MetaGenomeSeq_group1/*
以 level2 水平为例,MetaGenomeSeq 分析结果见 : result/06.StatisticalTest/CAZy/MetaGenomeSeq_group1/level2/*.all.xls
从 MetaGenomeSeq 分析结果中,筛选出的 P value<=0.05的信息见 : result/06.StatisticalTest/CAZy/MetaGenomeSeq_group1/level2/*.psig.xls
从 MetaGenomeSeq 分析结果中,筛选出的 Q value<=0.05的信息见 : result/06.StatisticalTest/CAZy/MetaGenomeSeq_group1/level2/*.qsig.xls
4.6.2.5 VFDB
图4.60 基于 VFDB 水平的 MetaGenomeSeq 分析
图中,横向为样品分组;纵向为对应功能的相对丰度。横线代表具有显著性差异的两个分组,没有则表示此功能在两个分组间不存在差异。“*”表示两组间差异显著(q value < 0.05),“**”表示两组间差异极显著(q value < 0.01)。
根据组间具有差异的功能进行主成分 PCA 分析和丰度聚类热图分析,展示结果如下:
图4.61 基于显著性差异功能的丰度聚类热图和 PCA 分析
a) 为显著性差异功能的丰度聚类热图:横向为样品信息;纵向为功能注释信息;图中左侧的聚类树为功能聚类树;中间热图对应的值为每一行功能相对丰度经过标准化处理后得到的 Z 值;即一个样品在某个分类上的 Z 值为样品在该分类上的相对丰度和所有样品在该分类的平均相对丰度的差除以所有样品在该分类上的标准差所得到的值;b) 为显著性差异功能的 PCA 图:横坐标表示第一主成分,百分比则表示第一主成分对样品差异的贡献值;纵坐标表示第二主成分,百分比表示第二主成分对样品差异的贡献值;图中的每个点表示一个样品,同一个组的样品使用同一种颜色表示。
结果目录 :
各分类层级的 MetaGenomeSeq 分析结果见 : result/06.StatisticalTest/VFDB/MetaGenomeSeq_group1/*
以 level2 水平为例,MetaGenomeSeq 分析结果见 : result/06.StatisticalTest/VFDB/MetaGenomeSeq_group1/level2/*.all.xls
从 MetaGenomeSeq 分析结果中,筛选出的 P value<=0.05的信息见 : result/06.StatisticalTest/VFDB/MetaGenomeSeq_group1/level2/*.psig.xls
从 MetaGenomeSeq 分析结果中,筛选出的 Q value<=0.05的信息见 : result/06.StatisticalTest/VFDB/MetaGenomeSeq_group1/level2/*.qsig.xls
4.6.2.6 PHI
图4.62 基于 PHI 水平的 MetaGenomeSeq 分析
图中,横向为样品分组;纵向为对应功能的相对丰度。横线代表具有显著性差异的两个分组,没有则表示此功能在两个分组间不存在差异。“*”表示两组间差异显著(q value < 0.05),“**”表示两组间差异极显著(q value < 0.01)。
根据组间具有差异的功能进行主成分 PCA 分析和丰度聚类热图分析,展示结果如下:
图4.63 基于显著性差异功能的丰度聚类热图和 PCA 分析
a) 为显著性差异功能的丰度聚类热图:横向为样品信息;纵向为功能注释信息;图中左侧的聚类树为功能聚类树;中间热图对应的值为每一行功能相对丰度经过标准化处理后得到的 Z 值;即一个样品在某个分类上的 Z 值为样品在该分类上的相对丰度和所有样品在该分类的平均相对丰度的差除以所有样品在该分类上的标准差所得到的值;b) 为显著性差异功能的 PCA 图:横坐标表示第一主成分,百分比则表示第一主成分对样品差异的贡献值;纵坐标表示第二主成分,百分比表示第二主成分对样品差异的贡献值;图中的每个点表示一个样品,同一个组的样品使用同一种颜色表示。
结果目录 :
各分类层级的 MetaGenomeSeq 分析结果见 : result/06.StatisticalTest/PHI/MetaGenomeSeq_group1/*
以 level1 水平为例,MetaGenomeSeq 分析结果见 : result/06.StatisticalTest/PHI/MetaGenomeSeq_group1/level1/*.all.xls
从 MetaGenomeSeq 分析结果中,筛选出的 P value<=0.05的信息见 : result/06.StatisticalTest/PHI/MetaGenomeSeq_group1/level1/*.psig.xls
从 MetaGenomeSeq 分析结果中,筛选出的 Q value<=0.05的信息见 : result/06.StatisticalTest/PHI/MetaGenomeSeq_group1/level1/*.qsig.xls
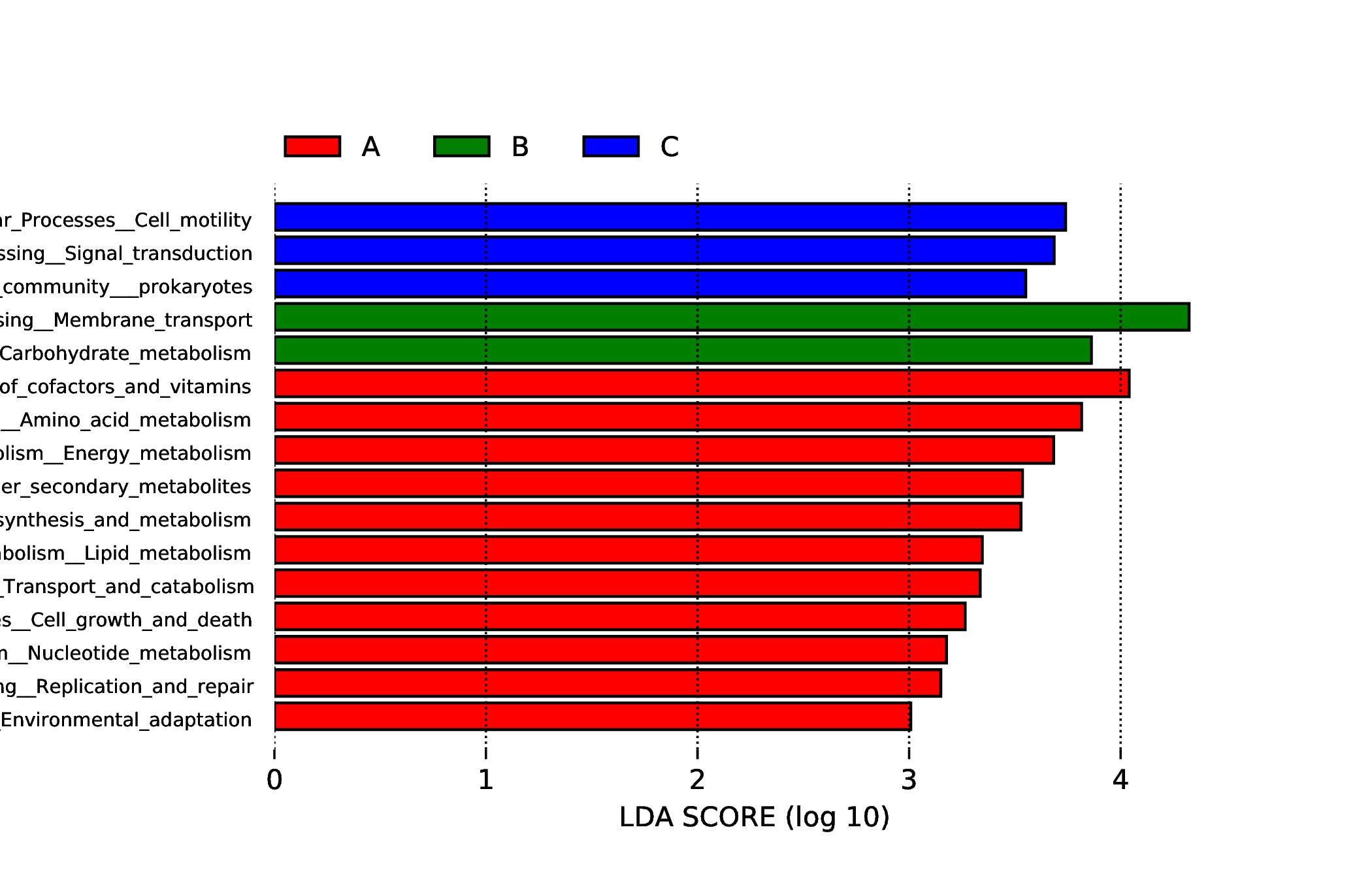
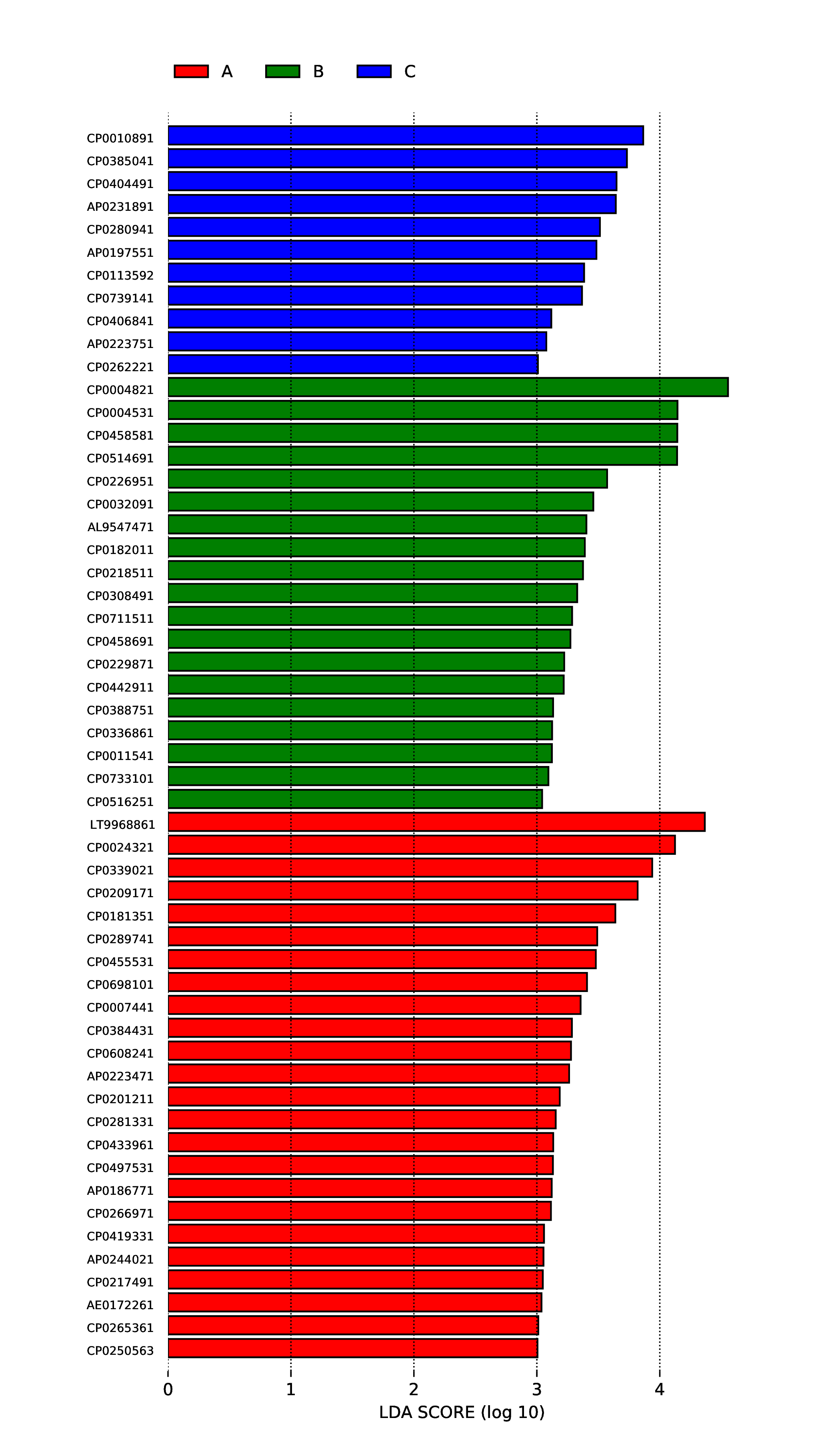
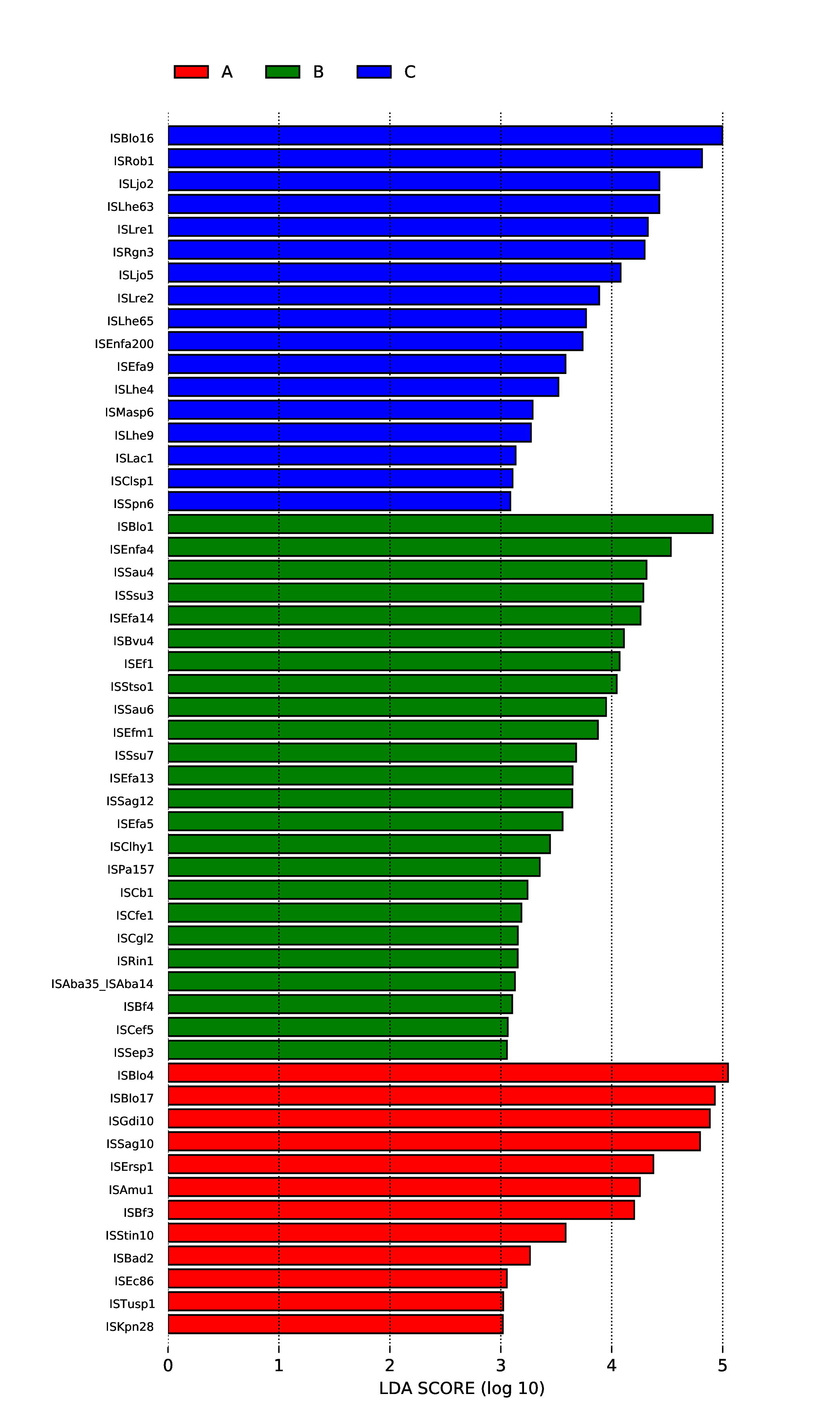
4.6.3 LEfSe 分析
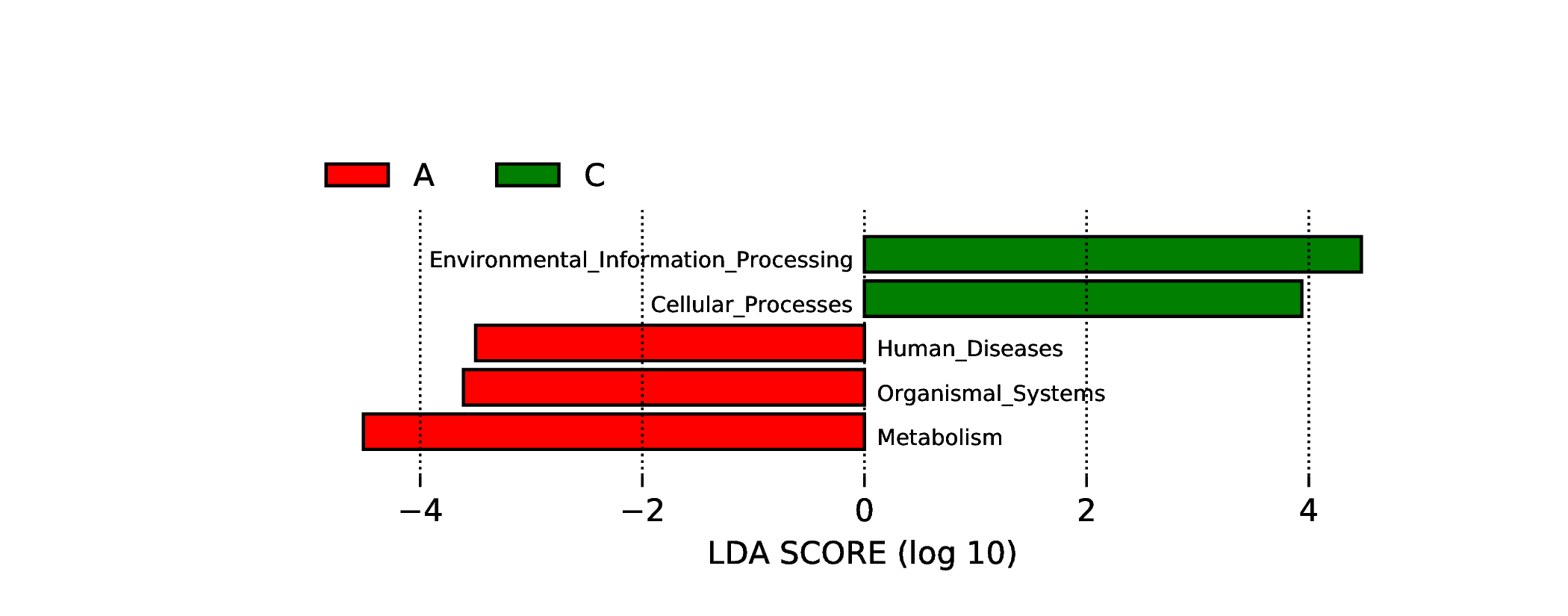
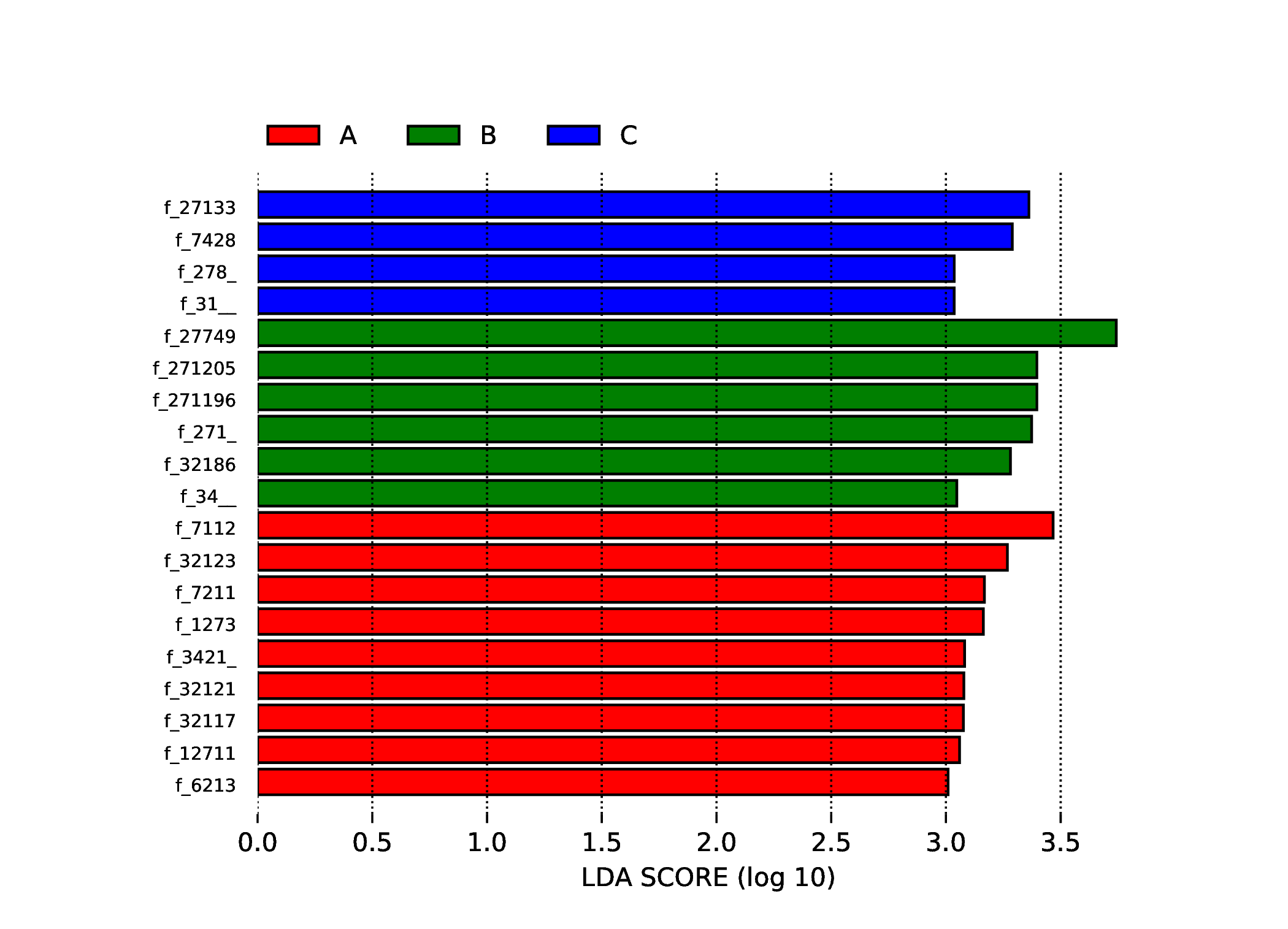
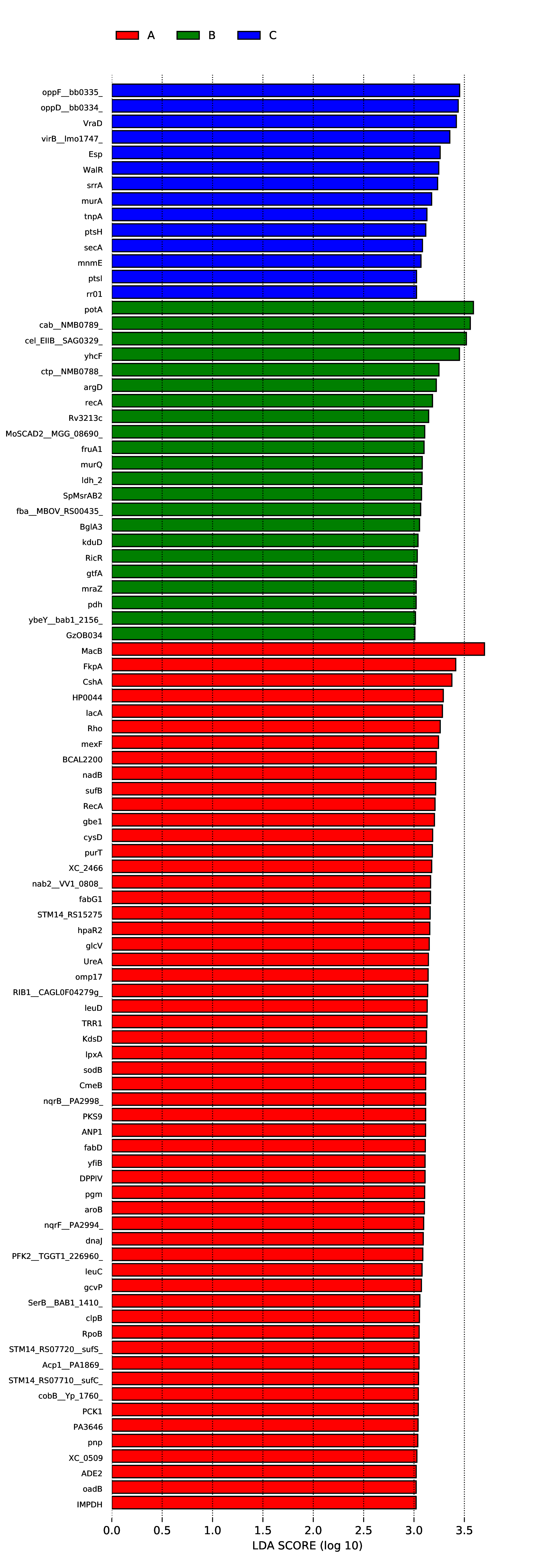
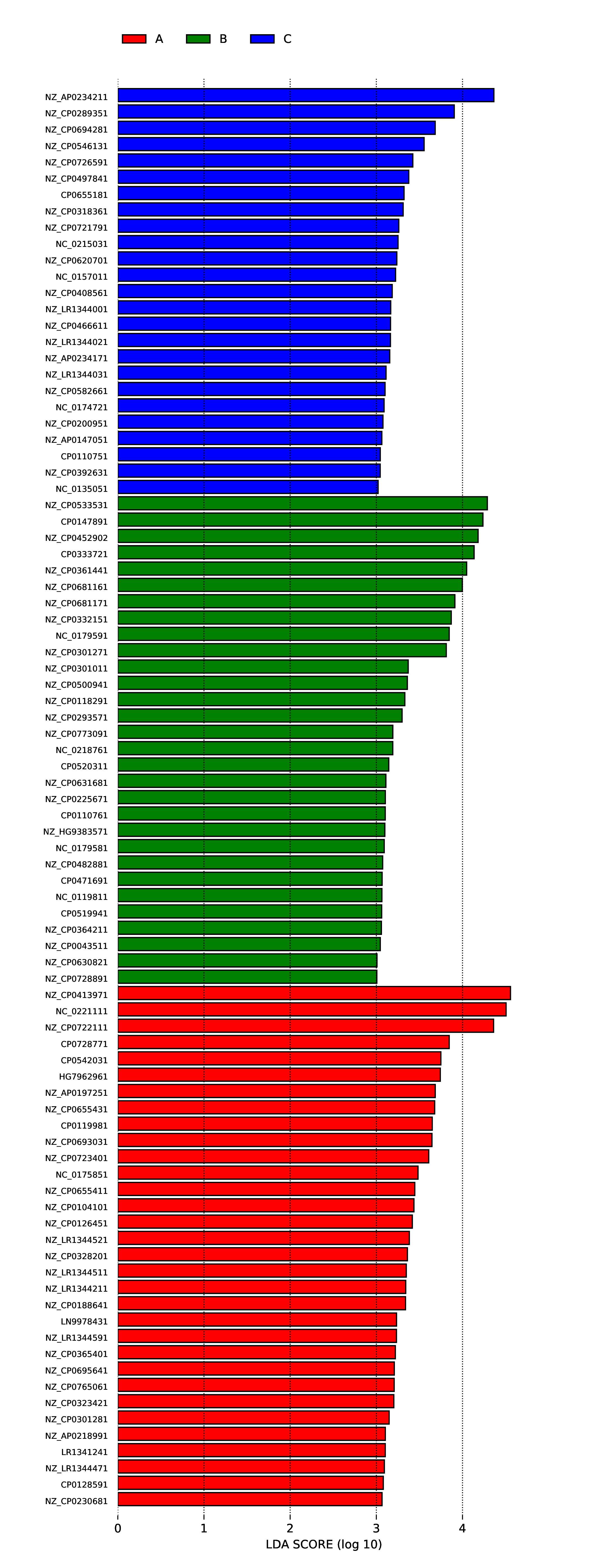
为了筛选组间具有显著差异的Biomarker,首先通过秩和检验的方法检测不同分组间的差异物种或功能并通过LDA(线性判别分析)实现降维并评估差异物种或功能的影响大小,即得到LDA score ;组间差异的LEfSe分析结果包括三部分,分别是LDA值分布柱状图,进化分支图(物种独有的系统发育分布)和组间具有统计学差异的Biomarker在不同组中丰度比较图。差异物种或功能的LDA值分布图如下:
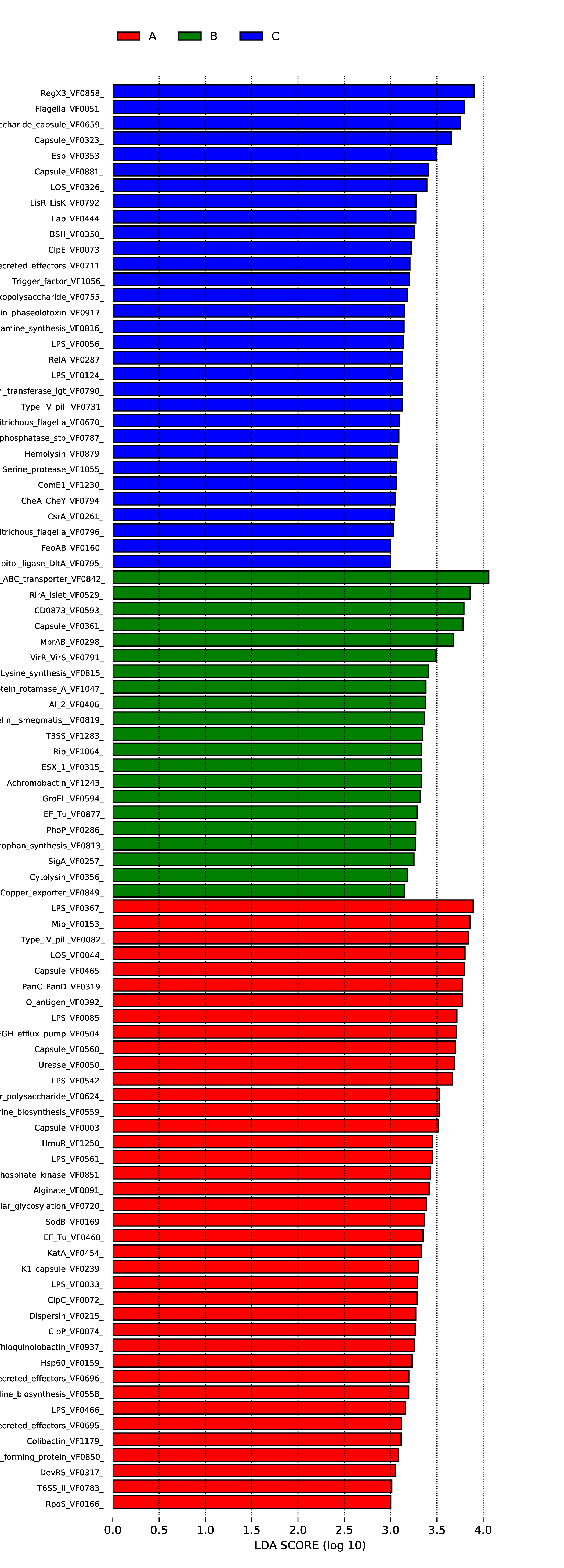
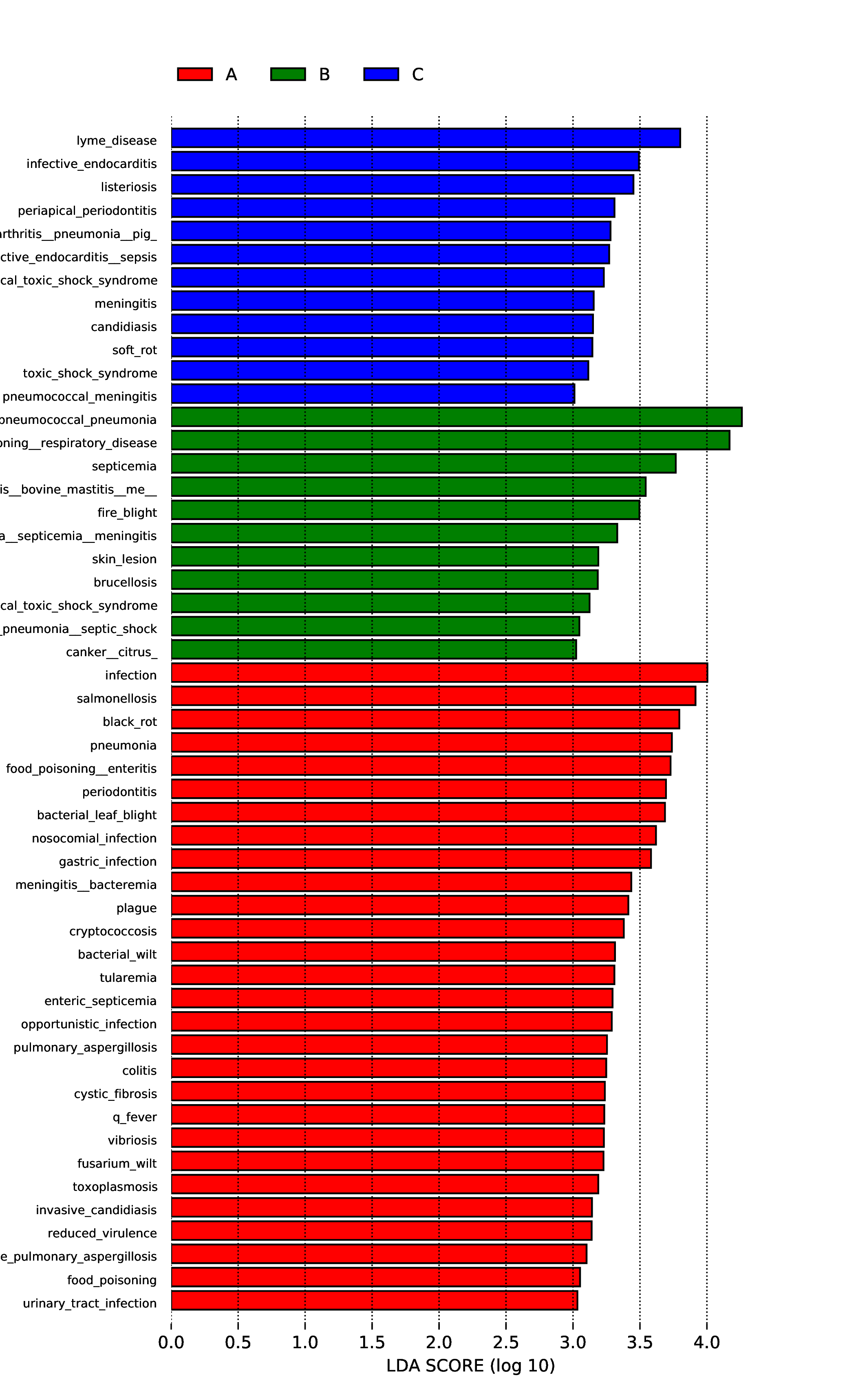
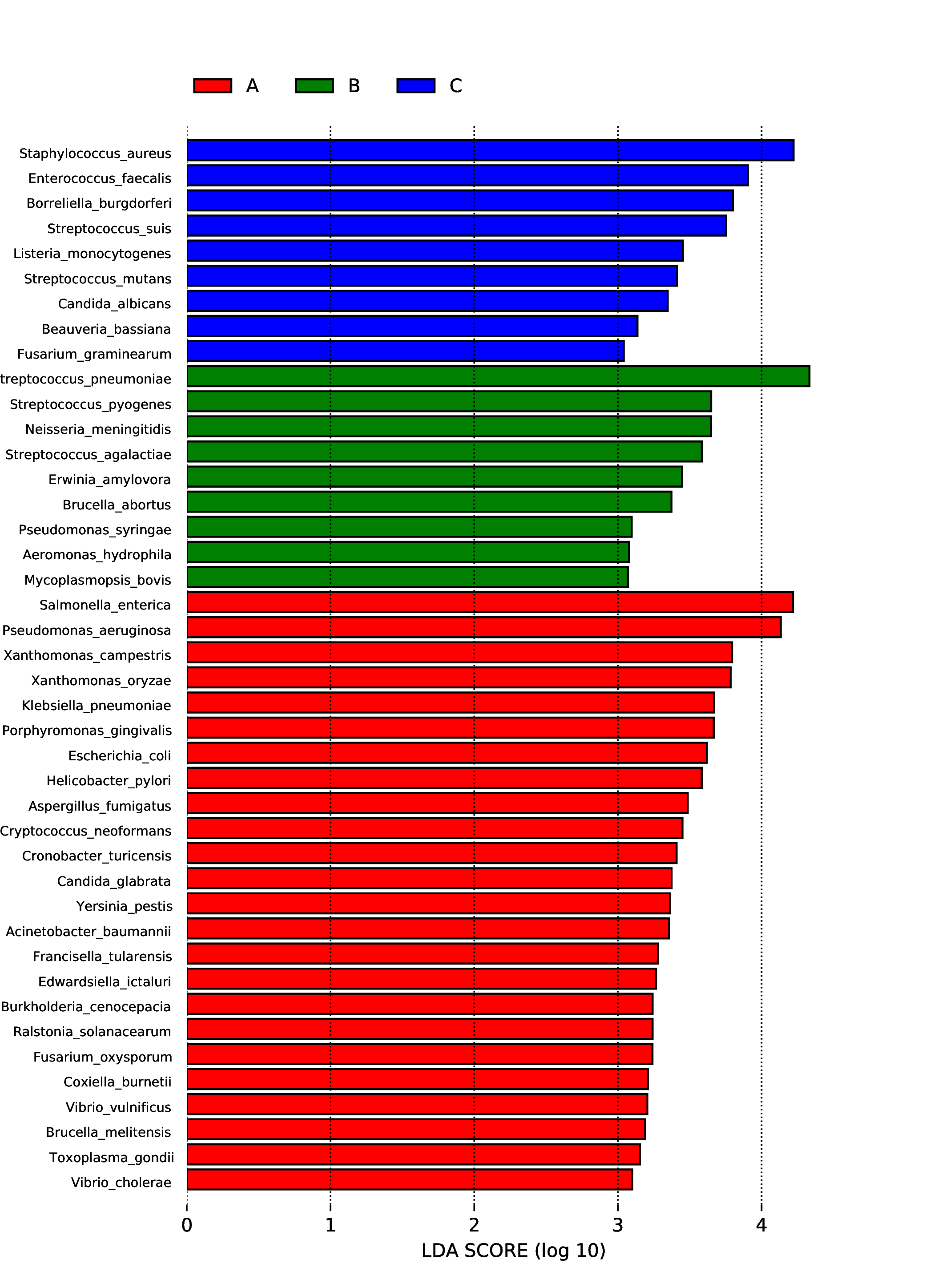
4.6.3.1 Micro_NR

图4.64 差异物种的LDA值分布图
说明 :左图为差异物种的LDA值分布图,LDA值分布柱状图中展示了LDA Score大于设定值(默认设置为4)的物种,即组间具有统计学差异的Biomarker,柱状图的长度代表差异物种的影响大小(即为 LDA Score)。右图为差异物种的进化分支图,由内至外辐射的圆圈代表了由门至属(或种)的分类级别。在不同分类级别上的每一个小圆圈代表该水平下的一个分类,小圆圈直径大小与相对丰度大小呈正比。着色原则:无显著差异的物种统一着色为黄色,差异物种Biomarker跟随组进行着色,红色节点表示在红色组别中起到重要作用的微生物类群,绿色节点表示在绿色组别中起到重要作用的微生物类群。图中英文字母表示的物种名称在右侧图例中进行展示。
结果目录 :
LEfSe分析结果见 : result/06.StatisticalTest/MicroNR/LEfSe_group1/*/*.{pdf,png}
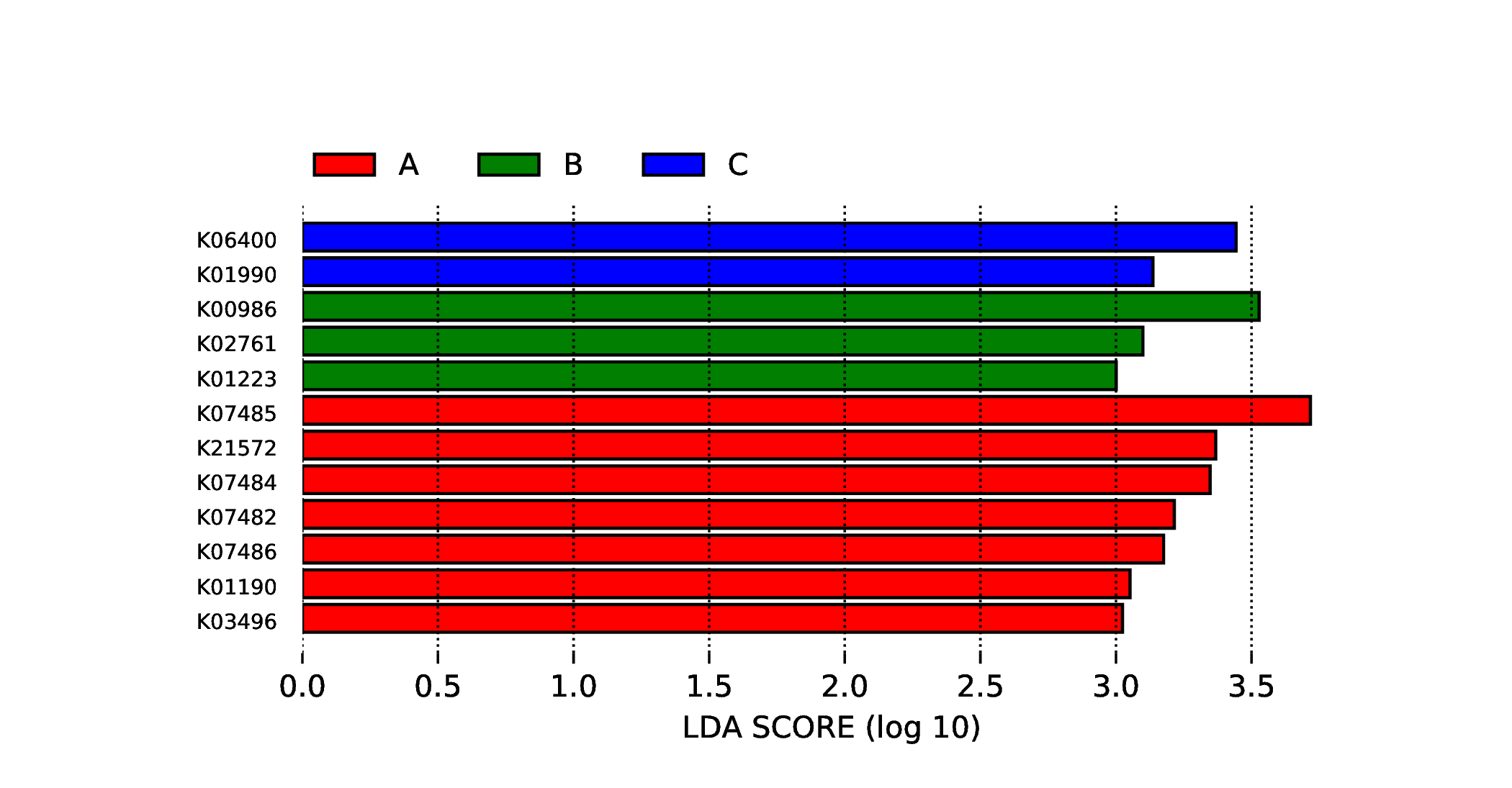
4.6.3.2 KEGG
图4.65 差异功能的LDA值分布图
说明 :差异功能的LDA值分布图,LDA值分布柱状图中展示了LDA Score大于设定值(默认设置为3)的功能,即组间具有统计学差异的Biomarker,柱状图的长度代表差异功能的影响大小(即为 LDA Score)。
结果目录 :
LEfSe分析结果见 : result/06.StatisticalTest/KEGG/LEfSe_group1/*/*.{pdf,png}
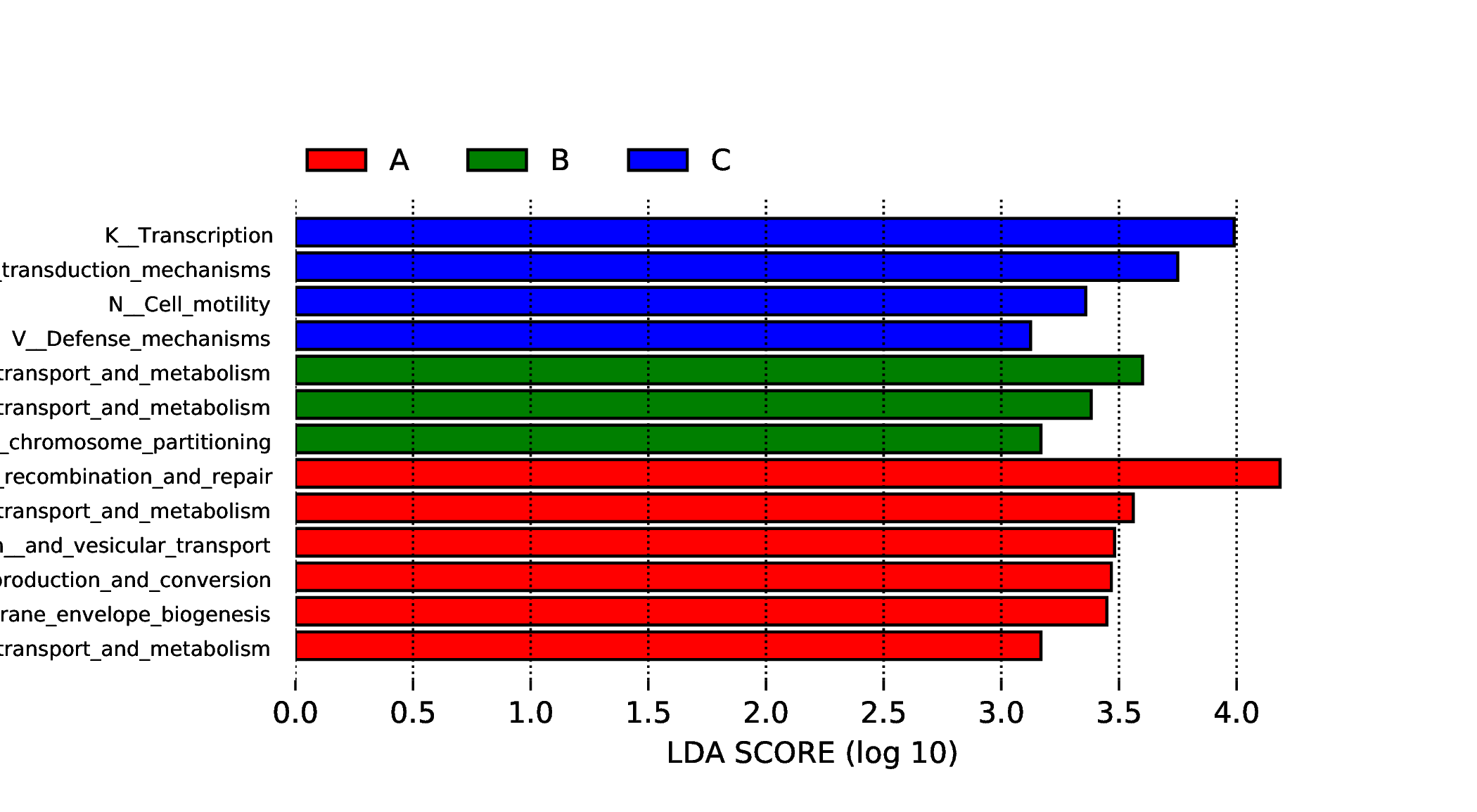
4.6.3.3 eggNOG
图4.66 差异功能的LDA值分布图
说明 :差异功能的LDA值分布图,LDA值分布柱状图中展示了LDA Score大于设定值(默认设置为3)的功能,即组间具有统计学差异的Biomarker,柱状图的长度代表差异功能的影响大小(即为 LDA Score)。
结果目录 :
LEfSe分析结果见 : result/06.StatisticalTest/eggNOG/LEfSe_group1/*/*.{pdf,png}
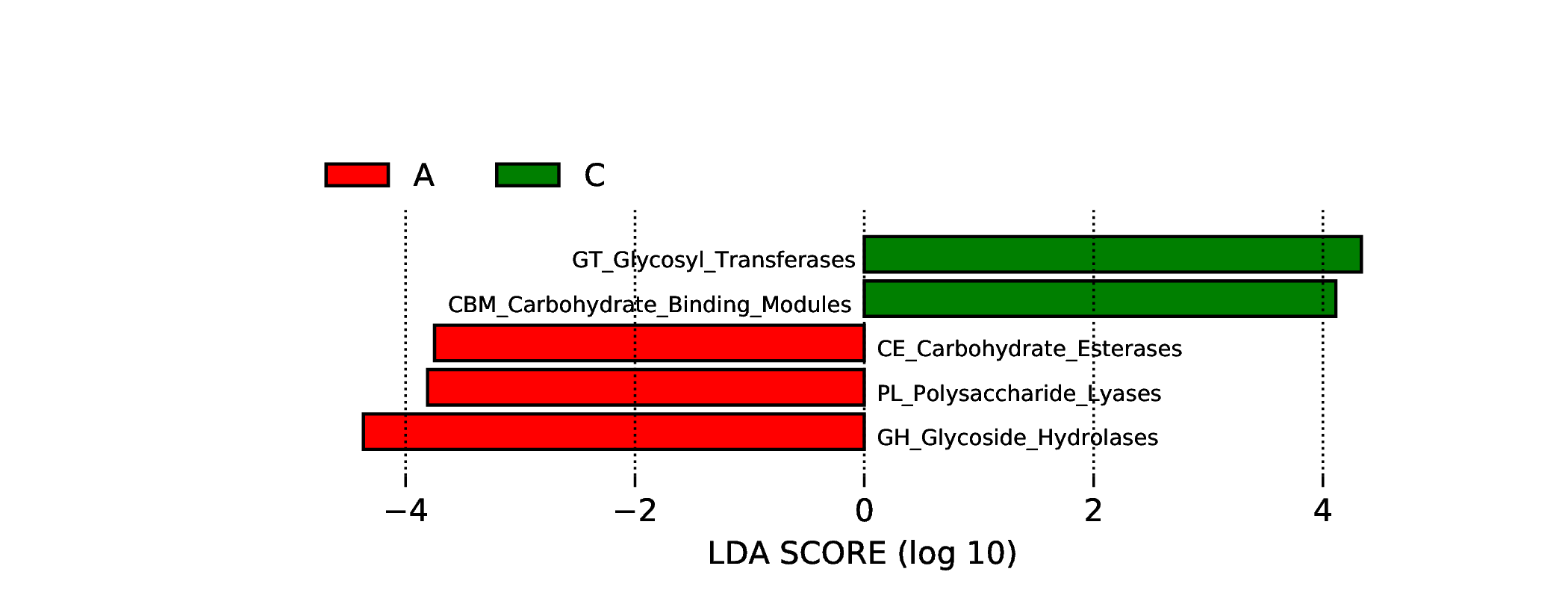
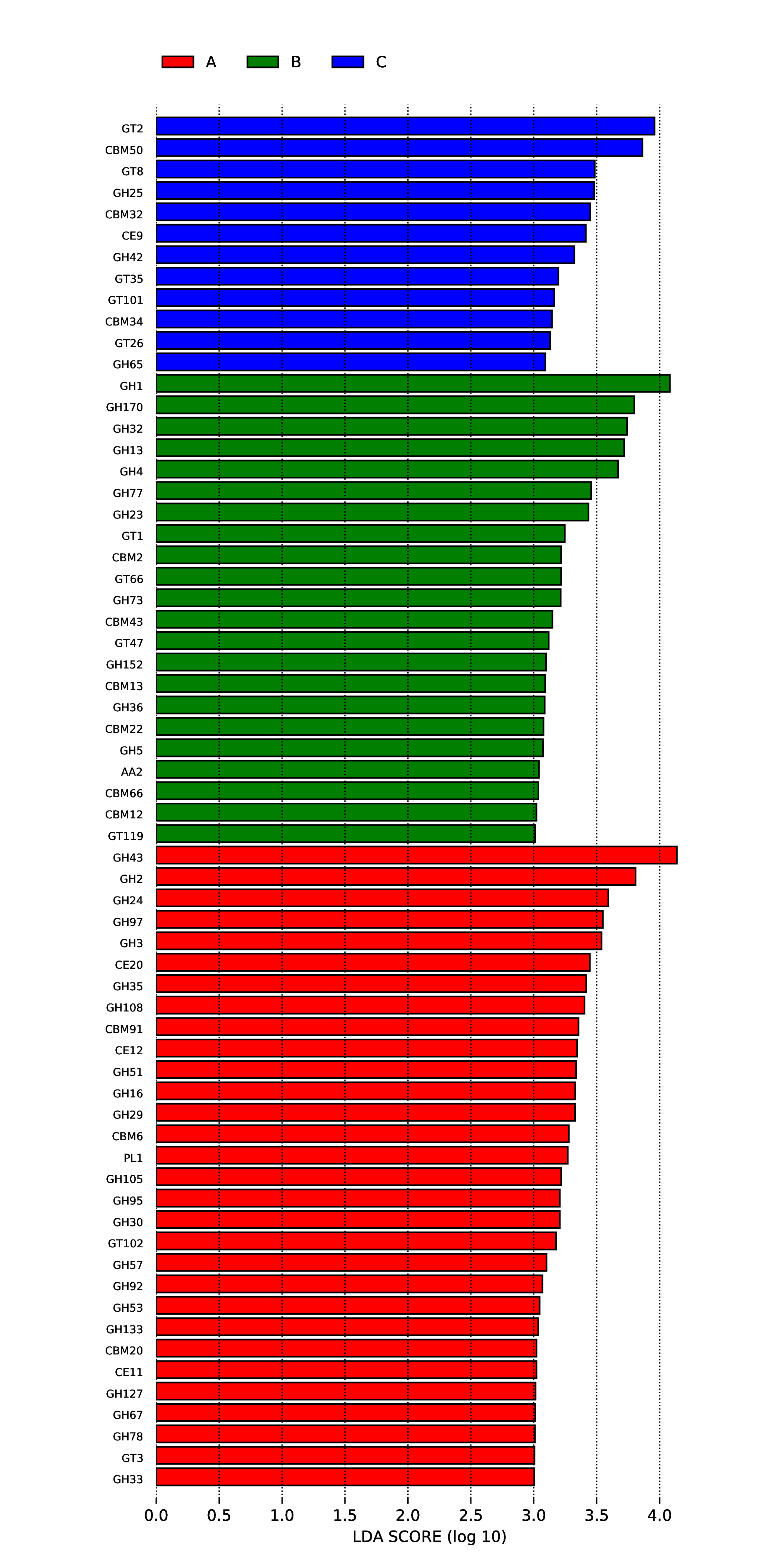
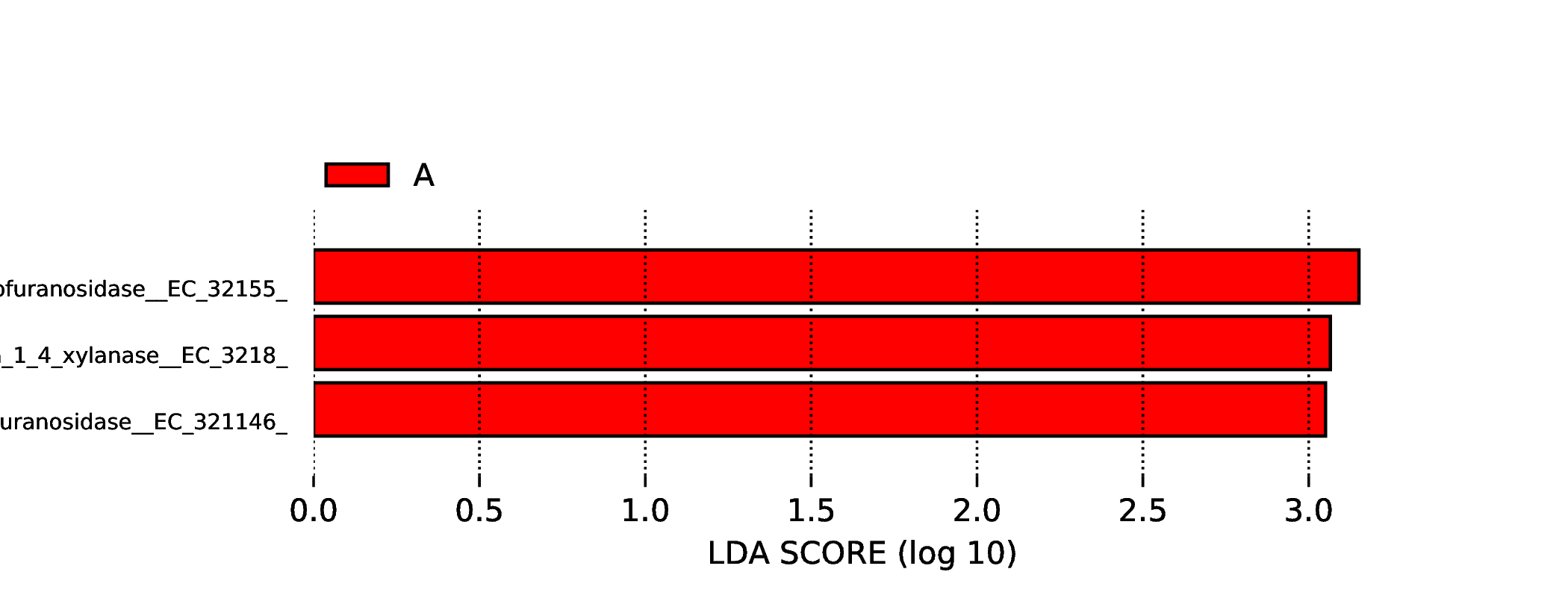
4.6.3.4 CAZy
图4.67 差异功能的LDA值分布图
说明 :差异功能的LDA值分布图,LDA值分布柱状图中展示了LDA Score大于设定值(默认设置为3)的功能,即组间具有统计学差异的Biomarker,柱状图的长度代表差异功能的影响大小(即为 LDA Score)。
结果目录 :
LEfSe分析结果见 : result/06.StatisticalTest/CAZy/LEfSe_group1/*/*.{pdf,png}
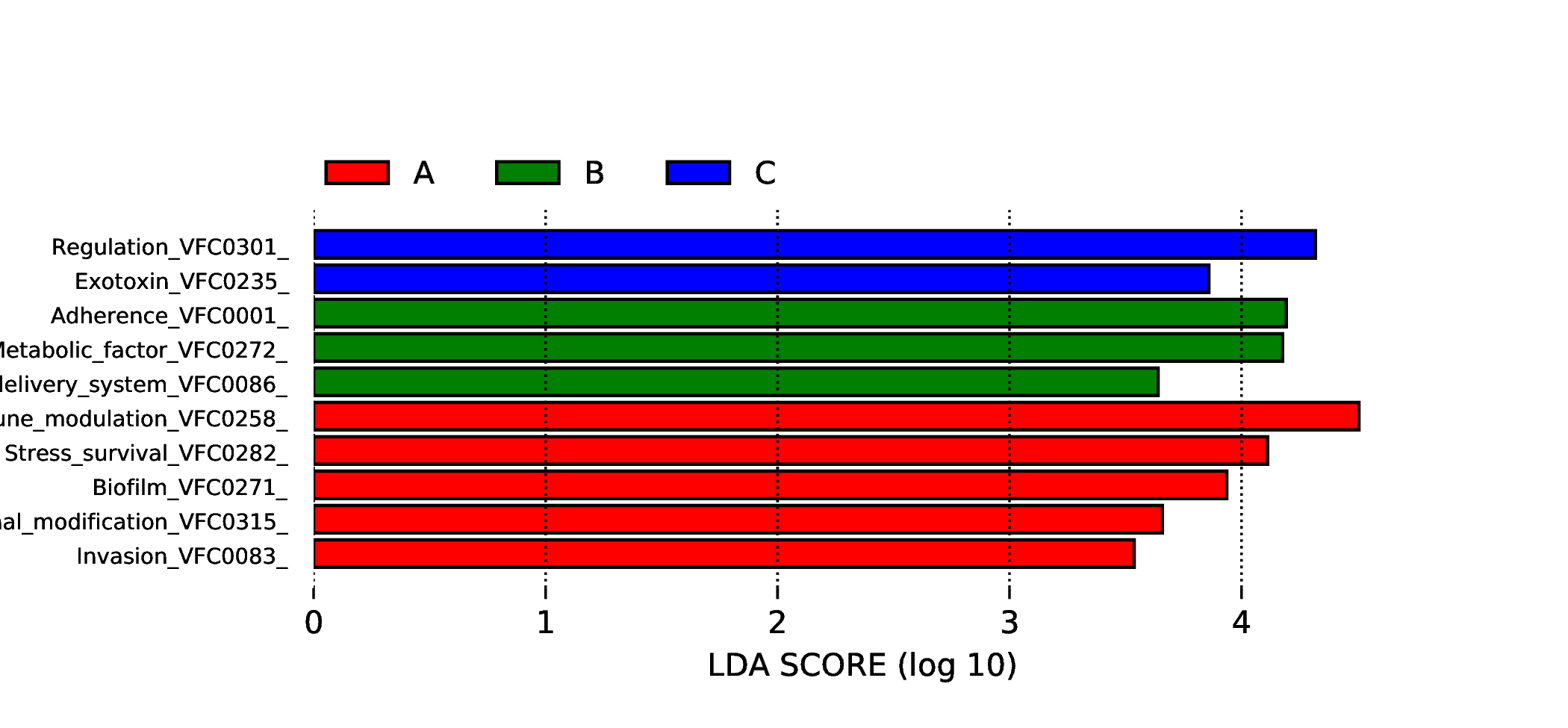
4.6.3.5 VFDB
图4.68 差异功能的LDA值分布图
说明 :差异功能的LDA值分布图,LDA值分布柱状图中展示了LDA Score大于设定值(默认设置为3)的功能,即组间具有统计学差异的Biomarker,柱状图的长度代表差异功能的影响大小(即为 LDA Score)。
结果目录 :
LEfSe分析结果见 : result/06.StatisticalTest/VFDB/LEfSe_group1/*/*.{pdf,png}
4.6.3.6 PHI
图4.69 差异功能的LDA值分布图
说明 :差异功能的LDA值分布图,LDA值分布柱状图中展示了LDA Score大于设定值(默认设置为3)的功能,即组间具有统计学差异的Biomarker,柱状图的长度代表差异功能的影响大小(即为 LDA Score)。
结果目录 :
LEfSe分析结果见 : result/06.StatisticalTest/PHI/LEfSe_group1/*/*.{pdf,png}
4.6.4 iPATH 分析
为了研究不同分组(不同样品)在代谢通路图中的差异,绘制了代谢通路网页版结果展示,整体网页版报告分为两部分:
第一部分为 KEGG 9 大 pathway overview 图: 图中,展示了两个分组(或两个样品)共有及特有的代谢通路信息,在代谢通路图中,节点代表各种化合物,边代表一系列的酶类反应,例如AvsB,红色代表两个分组(或两个样品)共有的酶类反应,蓝色代表分组 A(或样品 A)独有的酶类反应,绿色代表分组 B(或样品 B)独有的酶类反应;
第二部分为注释到的 pathway 代谢通路图: 在代谢通路图中,节点代表各种化合物, 方框代表酶类信息(默认边框为黑色,背景为白色),不同颜色的方框代表注释为该酶类的不同 Unigenes 数目。
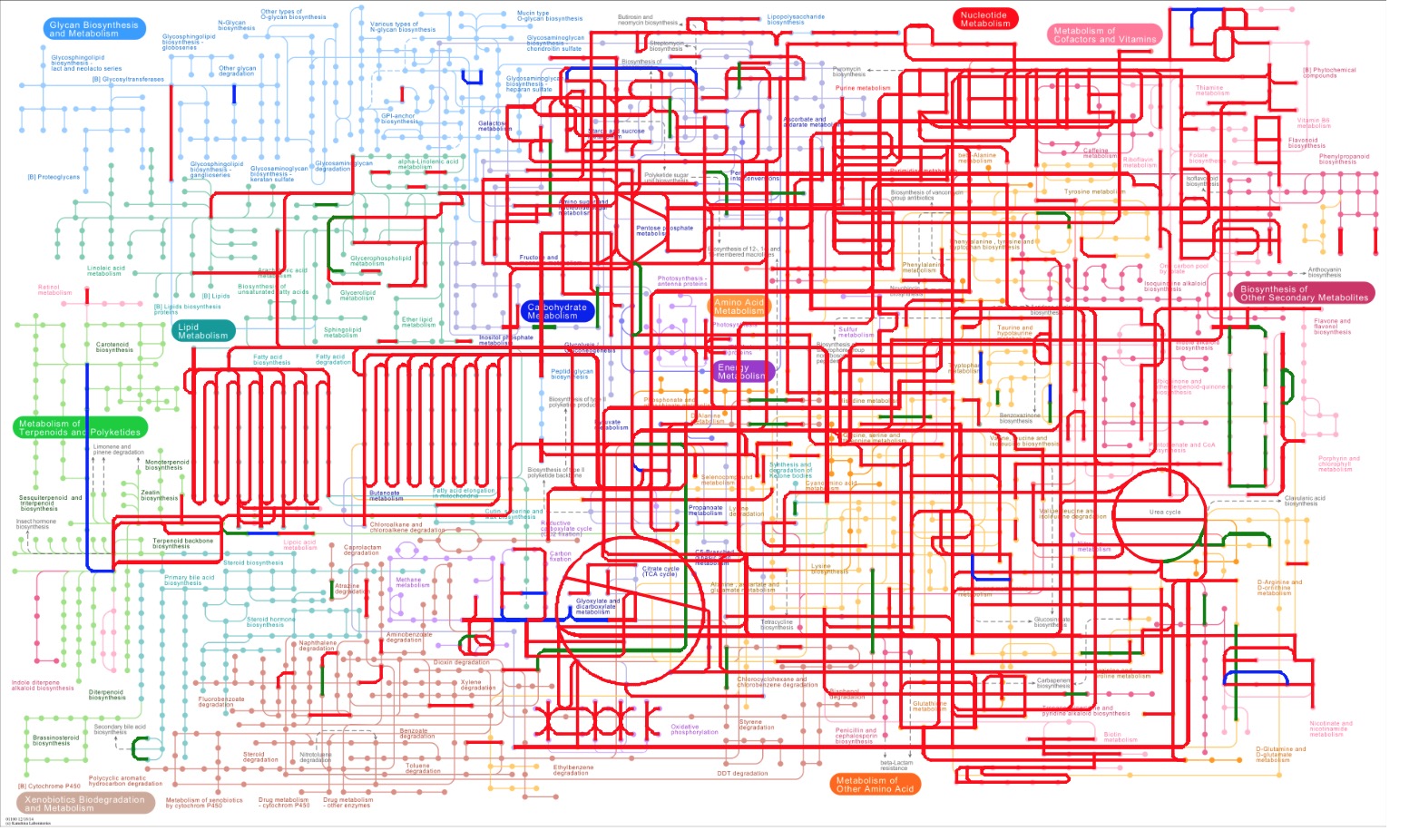
图4.70 多样品代谢通路比较分析示例图
结果目录 :
多样品代谢通路比较分析结果见 : result/06.StatisticalTest/KEGG/iPATH/Pathway_group1
4.7 抗性基因注释与分析
不管是人肠道微生物还是其他环境微生物中,抗性基因是普遍存在的。抗生素的滥用导致人体和环境中微生物群落发生不可逆的变化,对人体健康和生态环境造成风险,因此抗性基因的相关研究受到了研究者的广泛关注(Martínez et al., 2014)。
4.7.1 功能注释
4.7.1.1 CARD抗性基因注释
The Comprehensive Antibiotic Resistance Database是近年来新出现的抗性基因数据库,它具有信息全面,对用户友好,更新维护及时等优势。该数据库的核心构成是Antibiotic Resistance Ontology(ARO),它整合了序列、抗生素抗性、作用机制、ARO之间的关联等信息,并在线提供ARO与PDB、NCBI等数据库的接口(Jia et al., 2017)。
4.7.1.1.1 注释基本步骤
抗性基因的注释,使用CARD数据库(v2.0.1)提供的Resistance Gene Identifier (RGI)软件将 Unigenes 与CARD数据库进行比对(RGI内置blastp,采用bitscore值对比对结果进行评分)(Mcarthur et al., 2013);根据RGI的比对结果,结合Unigenes的丰度信息,统计出各ARO的相对丰度。从ARO的丰度出发,进行丰度柱形图展示,丰度聚类热图展示,丰度分布圈图展示,组间ARO差异分析,抗性基因(注释到ARO的unigenes)物种归属分析等(对部分名称较长的ARO,用其前三个单词与下划线缩写的形式展示)。
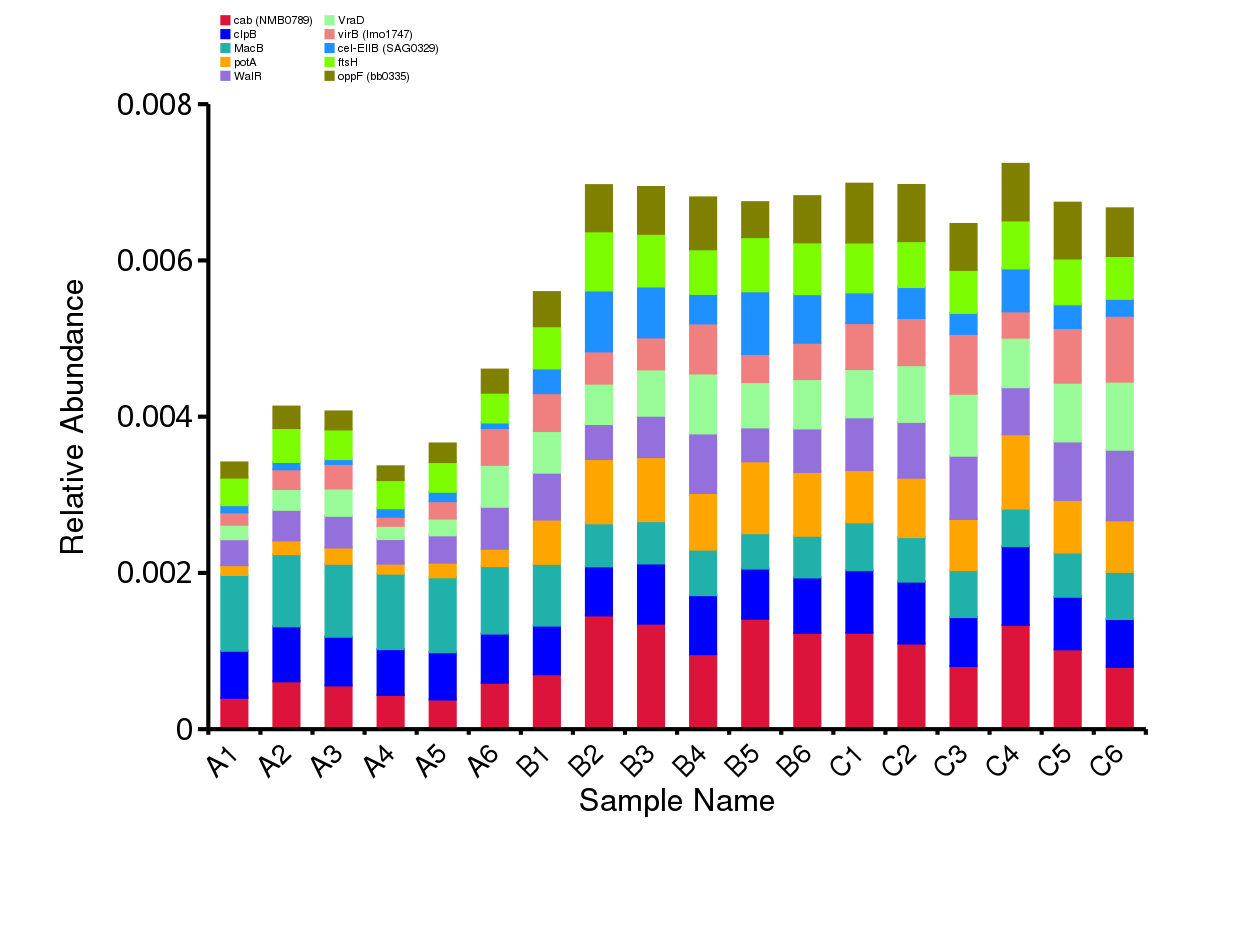
4.7.1.1.2 功能相对丰度概况
从抗性基因的相对丰度表出发,计算各个样品中ARO的含量,筛选出最大丰度排名前20的ARO结果展示如下:
图4.71 不同ARO在各样本中的丰度柱形图
说明:ARO在各个样品中所有基因的相对丰度。ppm,表示单位为ppm,即将原始相对丰度数据放大10e6倍的结果;relative,表示top20 ARO在所有 ARO中的相对丰度,others为非top 20 ARO相对丰度总和。
结果目录:
top20 的 ARO 在各样品的相对丰度见:result/07.ResistantGene/CARD/Anno/Bar/ARO/Unigenes.ARO.ppm.{png,svg}
top20 的 ARO 在各样品的相对百分含量见:result/07.ResistantGene/CARD/Anno/Bar/ARO/Unigenes.ARO.RelativePercent.{png,svg}
4.7.1.2 MGEs转移元件注释
可移动遗传元件mobile genetic elements (MGEs),是指能在基因组内转移的一类DNA,包含整合子、插入序列、质粒等。其中,整合子(integrons)能够捕获和整合外源性基因,使之转变为功能性基因的表达单位,增强细菌生存的适应性,并导致多重耐药基因在细菌中的水平传播;插入序列(Insertion Sequence,IS)是最简单的转座元件,可以通过转座作用在不同位置插入到基因组中,从而可能引起基因突变、基因表达改变等,对生物体的遗传特性和表型产生影响;质粒(plasmid)能够自主复制,大部分质粒是环状构型,存在于许多细菌以及酵母菌等生物中,乃至于植物的线粒体中。
4.7.1.2.1 注释基本步骤
使用blastn软件将Unigenes与插入序列(isfinder,https://isfinder.biotoul.fr/)、整合子(integrall,http://integrall.bio.ua.pt/)和质粒(PLSDB,https://ccb-microbe.cs.uni-saarland.de/plsdb/)数据库进行比对,对于每一条序列的比对结果,选取score最高的比对结果(one HSP > 60 bits)。
4.7.1.2.2 功能相对丰度概况
从抗性基因的相对丰度表出发,计算各个样品中MGEs的含量和百分比,筛选出最大丰度排名前20的MGEs结果展示如下:
图4.72 不同MGEs在各样本中的丰度柱形图
说明:表示top20 MGEs在所有MGEs中的相对丰度,Others为非top 20 MGEs相对丰度总和(NonARO除外)。
结果目录:
top20 的 MGEs 在各样品的相对丰度见:result/07.ResistantGene/MGES/*/Anno/Bar/*/Unigenes.*.ppm.{png,svg}
top20 的 MGEs 在各样品的相对百分含量见:result/07.ResistantGene/MGES/*/Anno/Bar/*/Unigenes.*.RelativePercent.{png,svg}
4.7.2 功能多样性分析
4.7.2.1 Alpha多样性分析
4.7.2.1.1 CARD
从抗性基因注释 Absolute 表出发,计算得到每个样本内物种的丰富度以及多样性。该表展示 CARD 结果。
表4.10 Alpha多样性分析统计结果
| Sample | ACE | chao1 | shannon | simpson | observed_species | goods_coverage |
|---|---|---|---|---|---|---|
| A1 | 43.4293228139382 | 42.25 | 0.0108799927823185 | 0.00213200249121237 | 42 | 1 |
| A2 | 42 | 42 | 0.0108125588490909 | 0.00205967057114076 | 42 | 1 |
| A3 | 47.6708382526564 | 49 | 0.0102213839331502 | 0.00194342009360282 | 46 | 1 |
| A4 | 36.3895184341492 | 36 | 0.0102539067824851 | 0.00201037859773734 | 36 | 1 |
| A5 | 48.2867741935484 | 50 | 0.0114341090936032 | 0.0022065765958339 | 47 | 1 |
| A6 | 48.5463059313215 | 47.3333333333333 | 0.010932664138539 | 0.00205451641063437 | 47 | 1 |
| B1 | 63.3033204569738 | 63 | 0.0150913616056208 | 0.00284167796574919 | 63 | 1 |
| B2 | 59 | 59 | 0.0152080666931286 | 0.00301811527226215 | 59 | 1 |
| B3 | 59.3798638730575 | 59 | 0.0143670337710835 | 0.00281104427513068 | 59 | 1 |
| B4 | 62.471360039362 | 62 | 0.0148030807153351 | 0.00279805836584013 | 62 | 1 |
| B5 | 57.1100242199074 | 56 | 0.0160782550731268 | 0.0032536383384687 | 54 | 1 |
| B6 | 63.256372325899 | 63 | 0.0159735301416334 | 0.00307908127553369 | 63 | 1 |
| C1 | 51.037037037037 | 50 | 0.0123106958597068 | 0.00234614892723195 | 49 | 1 |
| C2 | 52 | 52 | 0.0131942103245942 | 0.0025078619319544 | 52 | 1 |
| C3 | 52 | 52 | 0.0143076301804716 | 0.00273236245309016 | 52 | 1 |
| C4 | 49.4855586046236 | 49 | 0.0111012746113426 | 0.00212270389053248 | 49 | 1 |
| C5 | 54.3632 | 54 | 0.0141608344719529 | 0.00270314480666356 | 54 | 1 |
| C6 | 53.0677551020408 | 53 | 0.014489028117648 | 0.00278805913838731 | 52 | 1 |
显示注释
(1)ACE:估计群落中功能数目。
(2)chao1:估计群落样品中包含的功能总数,群落中低丰度功能越多,chao1指数越大。
(3)shannon:样品中的分类总数及其占比。群落多样性越高,功能分布越均匀,shannon指数越大。
(4)simpson:表征群落内功能分布的多样性和均匀度,功能均匀度越好,Simpson指数越大。
(5)observed_species:直观观测到的功能数目,指数越大观测到的功能越多。
(6)goods_coverage:覆盖度,测序覆盖度越高,指数越大。
结果目录:
CARD Alpha多样性指数汇总表见:result/07.ResistantGene/CARD/Diversity/AlphaDiversity/Alpha_index_table/*.xls
4.7.2.1.2 MGEs
从抗性基因注释 Absolute 表出发,计算得到每个样本内物种的丰富度以及多样性。该表展示 Integrall 结果。
表4.11 Integrall 多样性分析统计结果
| Sample | ACE | chao1 | shannon | simpson | observed_species | goods_coverage |
|---|---|---|---|---|---|---|
| A1 | 253.709411148522 | 254.5 | 3.94261174483602 | 0.961561709975321 | 251 | 1 |
| A2 | 258.944668303247 | 258.5 | 3.81769277128211 | 0.95262997851552 | 258 | 1 |
| A3 | 273.667203072505 | 277 | 3.92077733899562 | 0.958484400852559 | 266 | 1 |
| A4 | 240.606307234303 | 241.2 | 3.67639178067444 | 0.944648198754464 | 237 | 1 |
| A5 | 260.284893617021 | 261.142857142857 | 4.07272978948873 | 0.967730262426443 | 256 | 1 |
| A6 | 266.599948373989 | 270 | 3.70138572558111 | 0.941327752171184 | 263 | 1 |
| B1 | 312.140416396996 | 313 | 4.04029826613588 | 0.965865120097148 | 310 | 1 |
| B2 | 282.923252978464 | 283.5 | 3.64546810254977 | 0.949093532827869 | 279 | 1 |
| B3 | 290.614629644684 | 290.076923076923 | 3.70616253015464 | 0.952434746680321 | 285 | 1 |
| B4 | 304.838701702928 | 304.428571428571 | 4.05330763499738 | 0.967275816998184 | 304 | 1 |
| B5 | 273.155567986051 | 277.272727272727 | 3.47350642962142 | 0.93978629078754 | 260 | 1 |
| B6 | 295.200402138209 | 294.75 | 3.85156085530933 | 0.957985152596157 | 294 | 1 |
| C1 | 273.172961838221 | 274.142857142857 | 3.95012014275983 | 0.964234266177722 | 269 | 1 |
| C2 | 275.245611907886 | 275 | 3.9431808248913 | 0.963283717044341 | 272 | 1 |
| C3 | 285.001328107919 | 283.625 | 4.08839536849014 | 0.969327640017278 | 281 | 1 |
| C4 | 271.535993981054 | 269.111111111111 | 3.83644337281334 | 0.960256883389439 | 263 | 1 |
| C5 | 273.129500818331 | 274.2 | 4.02848344354065 | 0.967376132376368 | 270 | 1 |
| C6 | 279.926779836336 | 289 | 4.09889366718822 | 0.969974783074737 | 274 | 1 |
显示注释
(1)ACE:估计群落中功能数目。
(2)chao1:估计群落样品中包含的功能总数,群落中低丰度功能越多,chao1指数越大。
(3)shannon:样品中的分类总数及其占比。群落多样性越高,功能分布越均匀,shannon指数越大。
(4)simpson:表征群落内功能分布的多样性和均匀度,功能均匀度越好,Simpson指数越大。
(5)observed_species:直观观测到的功能数目,指数越大观测到的功能越多。
(6)goods_coverage:覆盖度,测序覆盖度越高,指数越大。
结果目录:
MGEs-integrall Alpha多样性指数汇总表见:result/07.ResistantGene/MGES/integrall/Diversity/AlphaDiversity/Alpha_index_table/*.xls
从抗性基因注释 Absolute 表出发,计算得到每个样本内物种的丰富度以及多样性。该表展示 Isfinder 结果。
表4.12 Isfinder 多样性分析统计结果
| Sample | ACE | chao1 | shannon | simpson | observed_species | goods_coverage |
|---|---|---|---|---|---|---|
| A1 | 41.1948951325683 | 46 | 2.21809121135371 | 0.847318863773997 | 36 | 1 |
| A2 | 45.2024979303891 | 45 | 2.27945145940268 | 0.843845173973907 | 45 | 1 |
| A3 | 46.2274436090226 | 45.25 | 2.59160492321184 | 0.890220685818359 | 45 | 1 |
| A4 | 42.4237034663248 | 42.5 | 1.96332334762071 | 0.793209662826052 | 41 | 1 |
| A5 | 49.3565750882759 | 49 | 2.46233490700998 | 0.857841391591495 | 49 | 1 |
| A6 | 44.5208124065953 | 44 | 2.32037044329062 | 0.831581542475161 | 43 | 1 |
| B1 | 89.2641101809344 | 92 | 3.32244133235588 | 0.944073134737333 | 85 | 1 |
| B2 | 80.1805989728704 | 79.5 | 3.13138754341404 | 0.917693731796936 | 79 | 1 |
| B3 | 74.2762390670554 | 74 | 3.02243566918527 | 0.919285662842829 | 74 | 1 |
| B4 | 85.252254798325 | 85 | 3.45114302584521 | 0.953661552781257 | 84 | 1 |
| B5 | 70.3033307058211 | 69.4285714285714 | 2.88728534841578 | 0.901008106318343 | 69 | 1 |
| B6 | 80.4743500958875 | 79.75 | 3.32171669965773 | 0.940187766581334 | 79 | 1 |
| C1 | 65.7386377808263 | 63.5 | 2.72534161535579 | 0.883339848244223 | 61 | 1 |
| C2 | 71.3790555555556 | 71 | 2.8276204616678 | 0.893843497317979 | 71 | 1 |
| C3 | 64.3314675351109 | 64 | 2.86092409916657 | 0.89072669149166 | 64 | 1 |
| C4 | 61.6484375 | 61 | 2.79289058563099 | 0.906213802803415 | 61 | 1 |
| C5 | 64 | 64 | 2.79532166041778 | 0.888639077149691 | 64 | 1 |
| C6 | 64.354071963272 | 64 | 2.74192039440485 | 0.878250820258648 | 64 | 1 |
显示注释
(1)ACE:估计群落中功能数目。
(2)chao1:估计群落样品中包含的功能总数,群落中低丰度功能越多,chao1指数越大。
(3)shannon:样品中的分类总数及其占比。群落多样性越高,功能分布越均匀,shannon指数越大。
(4)simpson:表征群落内功能分布的多样性和均匀度,功能均匀度越好,Simpson指数越大。
(5)observed_species:直观观测到的功能数目,指数越大观测到的功能越多。
(6)goods_coverage:覆盖度,测序覆盖度越高,指数越大。
结果目录:
MGEs-isfinder Alpha多样性指数汇总表见:result/07.ResistantGene/MGES/isfinder/Diversity/AlphaDiversity/Alpha_index_table/*.xls
从抗性基因注释 Absolute 表出发,计算得到每个样本内物种的丰富度以及多样性。该表展示 Plasmid 结果。
表4.13 Plasmid 多样性分析统计结果
| Sample | ACE | chao1 | shannon | simpson | observed_species | goods_coverage |
|---|---|---|---|---|---|---|
| A1 | 1052.91759097362 | 1083.56 | 4.58480391144521 | 0.967182793041138 | 998 | 1 |
| A2 | 1144.50221130055 | 1161.62222222222 | 4.85613872474593 | 0.981143680101542 | 1111 | 1 |
| A3 | 1182.06990673271 | 1201.42 | 4.80865645311165 | 0.977773150563363 | 1135 | 1 |
| A4 | 948.479933794371 | 954.448275862069 | 4.54251592898836 | 0.97264929558409 | 904 | 1 |
| A5 | 1134.56531063942 | 1143.59649122807 | 4.66961284670357 | 0.973095638978488 | 1100 | 1 |
| A6 | 1209.90212956472 | 1221.875 | 4.74157743395307 | 0.977725113715331 | 1185 | 1 |
| B1 | 1617.30104555384 | 1645.96226415094 | 4.84812107017735 | 0.969697979730342 | 1577 | 1 |
| B2 | 1451.51252779942 | 1469.11428571429 | 4.63036547618701 | 0.975371616879388 | 1408 | 1 |
| B3 | 1419.81688067635 | 1441.33823529412 | 4.85926271522677 | 0.98097149464482 | 1370 | 1 |
| B4 | 1590.55312254093 | 1596.51666666667 | 4.78222670369474 | 0.966610320883688 | 1565 | 1 |
| B5 | 1277.35783322903 | 1293.84375 | 4.60412223606863 | 0.975943227752389 | 1227 | 1 |
| B6 | 1513.61193568103 | 1524.6 | 4.85114709003732 | 0.974663215617233 | 1488 | 1 |
| C1 | 1310.81171788802 | 1335.05172413793 | 4.86447804451848 | 0.974340015257395 | 1266 | 1 |
| C2 | 1371.60921797556 | 1394.46808510638 | 4.83697773114787 | 0.973513742561464 | 1337 | 1 |
| C3 | 1393.97589641123 | 1441.375 | 4.70304205564494 | 0.959330617751046 | 1350 | 1 |
| C4 | 1337.28261734878 | 1377.10909090909 | 4.89927459134063 | 0.981487928371699 | 1274 | 1 |
| C5 | 1324.31976123196 | 1341.875 | 4.67588232789292 | 0.958257490544209 | 1293 | 1 |
| C6 | 1372.59878128302 | 1404.26315789474 | 4.5824911567591 | 0.946232923724435 | 1337 | 1 |
显示注释
(1)ACE:估计群落中功能数目。
(2)chao1:估计群落样品中包含的功能总数,群落中低丰度功能越多,chao1指数越大。
(3)shannon:样品中的分类总数及其占比。群落多样性越高,功能分布越均匀,shannon指数越大。
(4)simpson:表征群落内功能分布的多样性和均匀度,功能均匀度越好,Simpson指数越大。
(5)observed_species:直观观测到的功能数目,指数越大观测到的功能越多。
(6)goods_coverage:覆盖度,测序覆盖度越高,指数越大。
结果目录:
MGEs-plasmid Alpha多样性指数汇总表见:result/07.ResistantGene/MGES/plasmid/Diversity/AlphaDiversity/Alpha_index_table/*.xls
4.7.2.2 基于相对丰度的样品聚类分析
4.7.2.2.1 CARD
根据所有样品在CARD数据库中的功能注释及丰度信息,选取丰度排名前 35 的功能及它们在每个样品中的丰度信息绘制热图,并从功能差异层面进行聚类。
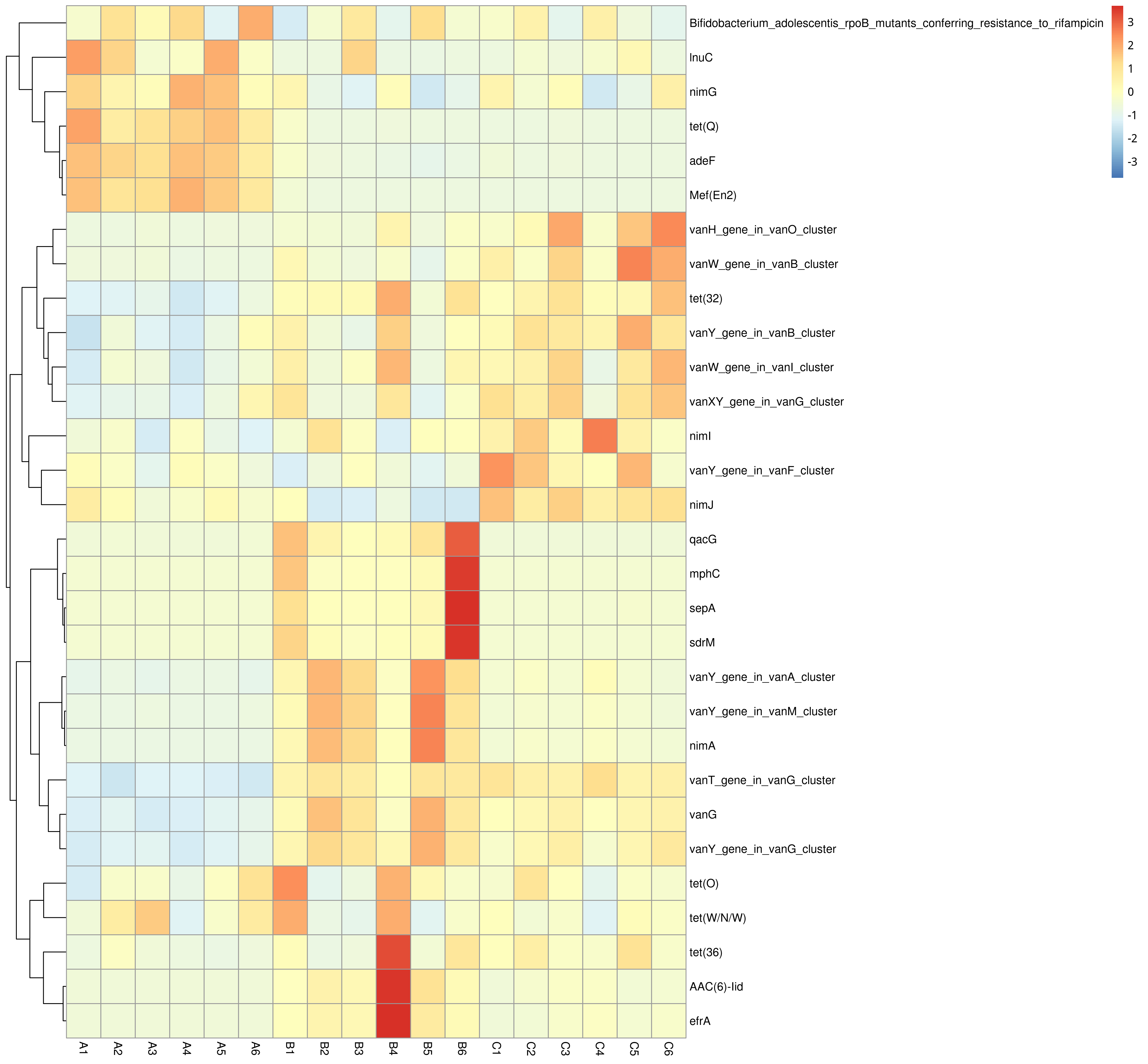
图4.73 CARD 功能丰度聚类热图
说明:横向为样品信息;纵向为CARD 注释信息;图中左侧的聚类树为功能聚类树;中间热图对应的值为每一行功能相对丰度经过标准化处理后得到的 Z 值;即一个样品在某个分类上的 Z 值为样品在该分类上的相对丰度和所有样品在该分类的平均相对丰度的差除以所有样品在该分类上的标准差所得到的值
结果目录:
CARD丰度聚类热图见 : result/07.ResistantGene/CARD/Anno/Heatmap_sample/ARO/ARO_heat/heat.{pdf,png}
4.7.2.2.2 MGEs
根据所有样品在MGEs 数据库中的功能注释及丰度信息,选取丰度排名前 35 的功能及它们在每个样品中的丰度信息绘制热图,并从功能差异层面进行聚类。
图4.74 MGEs 功能丰度聚类热图
说明:横向为样品信息;纵向为MGEs 注释信息;图中左侧的聚类树为功能聚类树;中间热图对应的值为每一行功能相对丰度经过标准化处理后得到的 Z 值;即一个样品在某个分类上的 Z 值为样品在该分类上的相对丰度和所有样品在该分类的平均相对丰度的差除以所有样品在该分类上的标准差所得到的值
结果目录:
MGEs 丰度聚类热图见 : result/07.ResistantGene/MGES/*/Anno/Heatmap_sample/*/*_heat/heat.{pdf,png}
4.7.2.3 主成分(PCA)分析
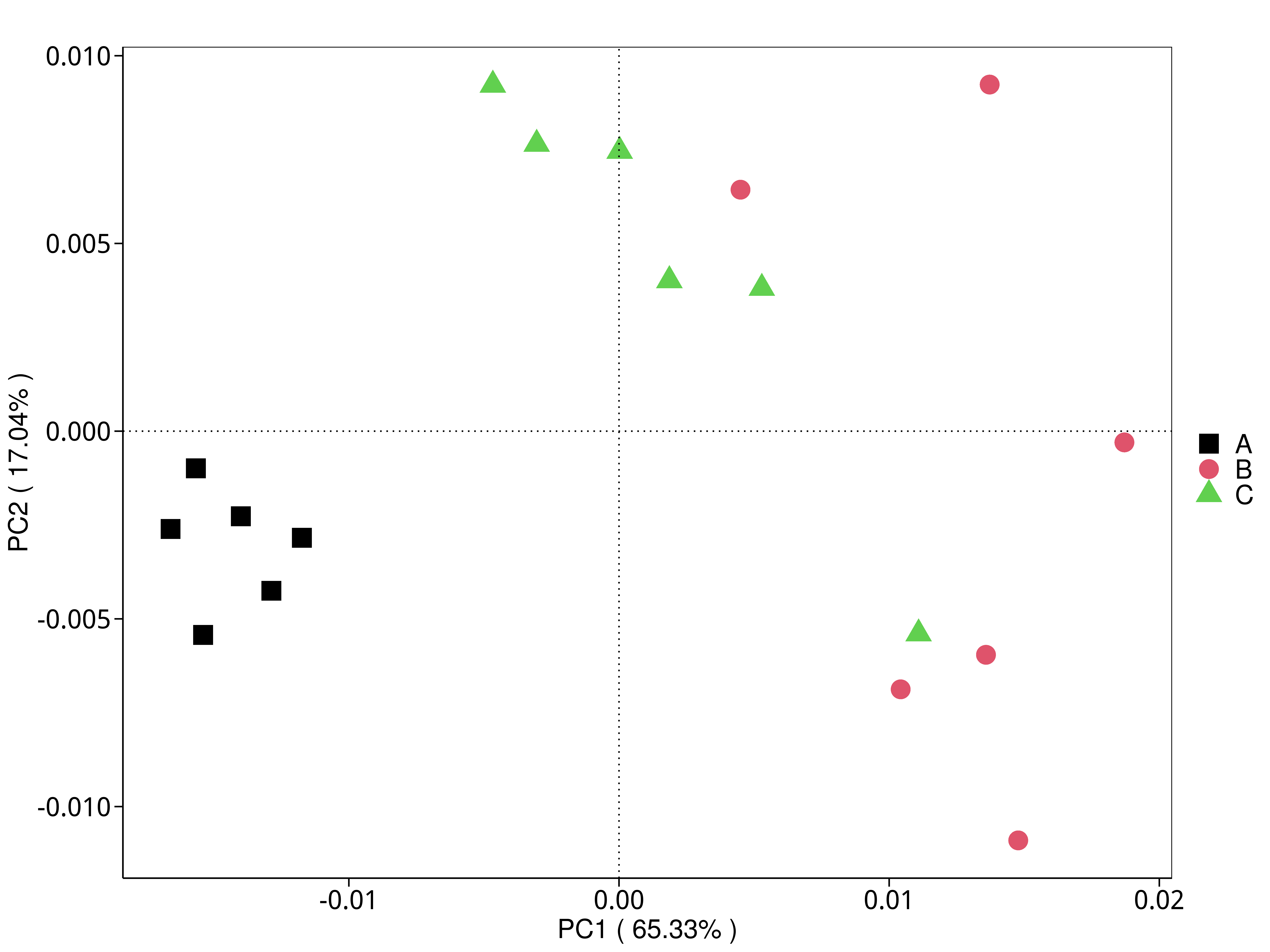
主成分分析 (PCA,Principal Component Analysis)是基于线型模型的一种降维分析,它应用方差分解的方法对多维数据进行降维,从而提取出数据中最主要的元素和结构(Rao et al., 2002);PCA 能够提取出最大程度反映样品间差异的两个坐标轴,从而将多维数据的差异反映在二维坐标图上,进而揭示复杂数据背景下的简单规律。
4.7.2.3.1 CARD
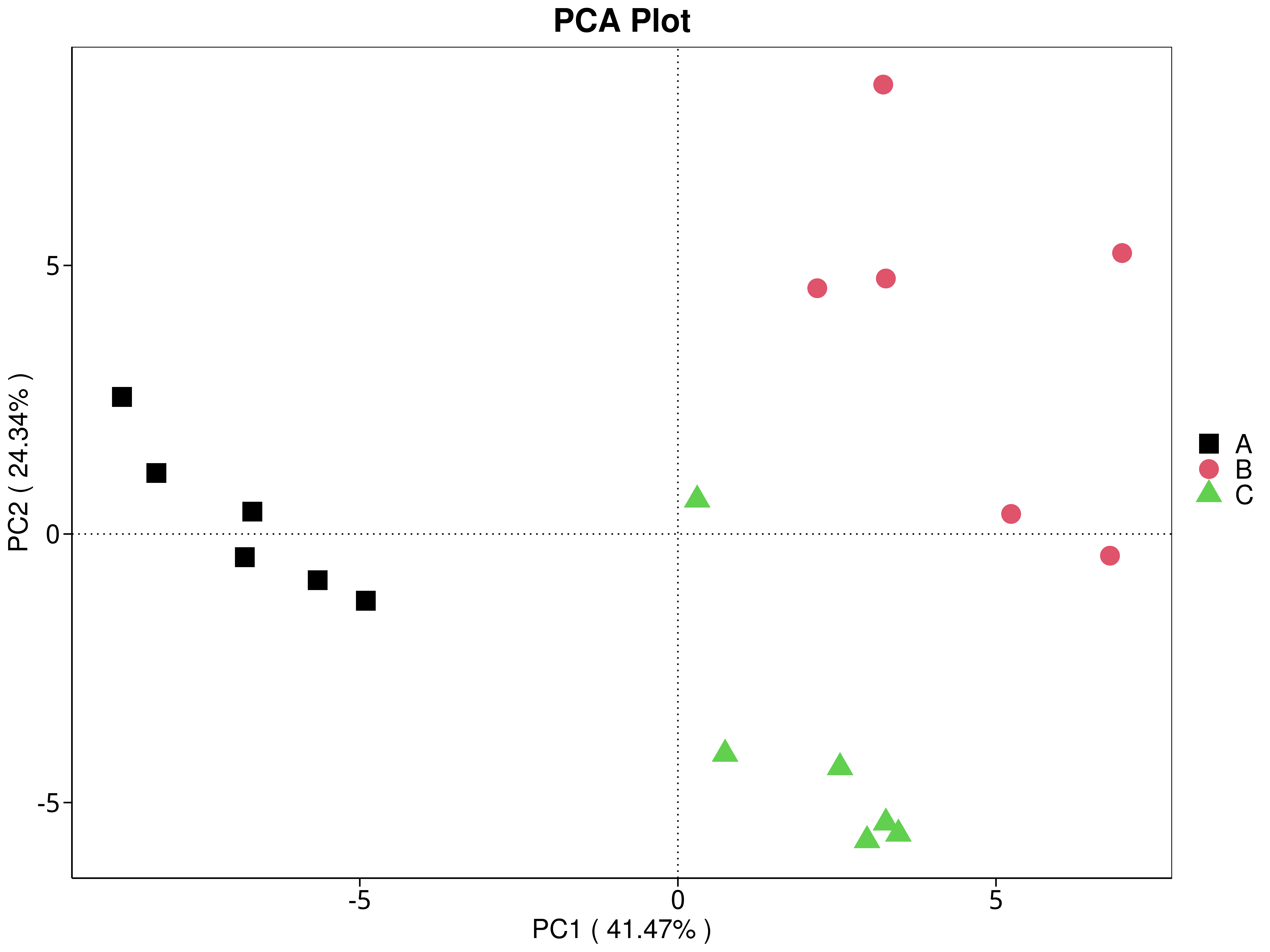
图4.75 基于 CARD 水平的 PCA 结果展示
说明:PCA分析,横坐标表示第一主成分,百分比则表示第一主成分对样品差异的贡献值;纵坐标表示第二主成分,百分比表示第二主成分对样品差异的贡献值;图中的每个点表示一个样品,同一个组的样品使用同一种颜色表示,没有分组默认按照样本出图
结果目录:
标注样品名的PCA图见 : result/07.ResistantGene/CARD/Diversity/PCA_group1/ARO/PCA_lable.{png,pdf}
未标注样品名的PCA图见 : result/07.ResistantGene/CARD/Diversity/PCA_group1/ARO/PCA.{png,pdf}
未标示样品名称的带聚类圈的PCA图见 : result/07.ResistantGene/CARD/Diversity/PCA_group1/ARO/PCA_circle.{png,pdf}
标示样品名称的带聚类圈的PCA图见 : result/07.ResistantGene/CARD/Diversity/PCA_group1/ARO/PCA_circleLable.{png,pdf}
4.7.2.3.2 MGEs
图4.76 基于 MGEs 水平的 PCA 结果展示
说明:PCA分析,横坐标表示第一主成分,百分比则表示第一主成分对样品差异的贡献值;纵坐标表示第二主成分,百分比表示第二主成分对样品差异的贡献值;图中的每个点表示一个样品,同一个组的样品使用同一种颜色表示,没有分组默认按照样本出图
结果目录:
标注样品名的PCA图见 : result/07.ResistantGene/MGES/*/Diversity/PCA_group1/*/PCA_lable.{png,pdf}
未标注样品名的PCA图见 : result/07.ResistantGene/MGES/*/Diversity/PCA_group1/*/PCA.{png,pdf}
未标示样品名称的带聚类圈的PCA图见 : result/07.ResistantGene/MGES/*/Diversity/PCA_group1/*/PCA_circle.{png,pdf}
标示样品名称的带聚类圈的PCA图见 : result/07.ResistantGene/MGES/*/Diversity/PCA_group1/*/PCA_circleLable.{png,pdf}
4.7.2.4 主坐标(PCoA)分析
主坐标分析(PCoA,Principal Co-ordinates Analysis),是通过一系列的特征值和特征向量排序从多维数据中提取出最主要的元素和结构。我们基于Bray-Curtis 距离来进行PCoA分析,并选取贡献率最大的主坐标组合进行作图展示。如果样品距离越接近,表示功能组成结构越相似,因此群落结构相似度高的样品倾向于聚集在一起,群落差异很大的样品则会远远分开。 基于不同分类层级的功能丰度表得到Bray-Curtis 距离矩阵,我们进行了 PCoA分析。
4.7.2.4.1 CARD
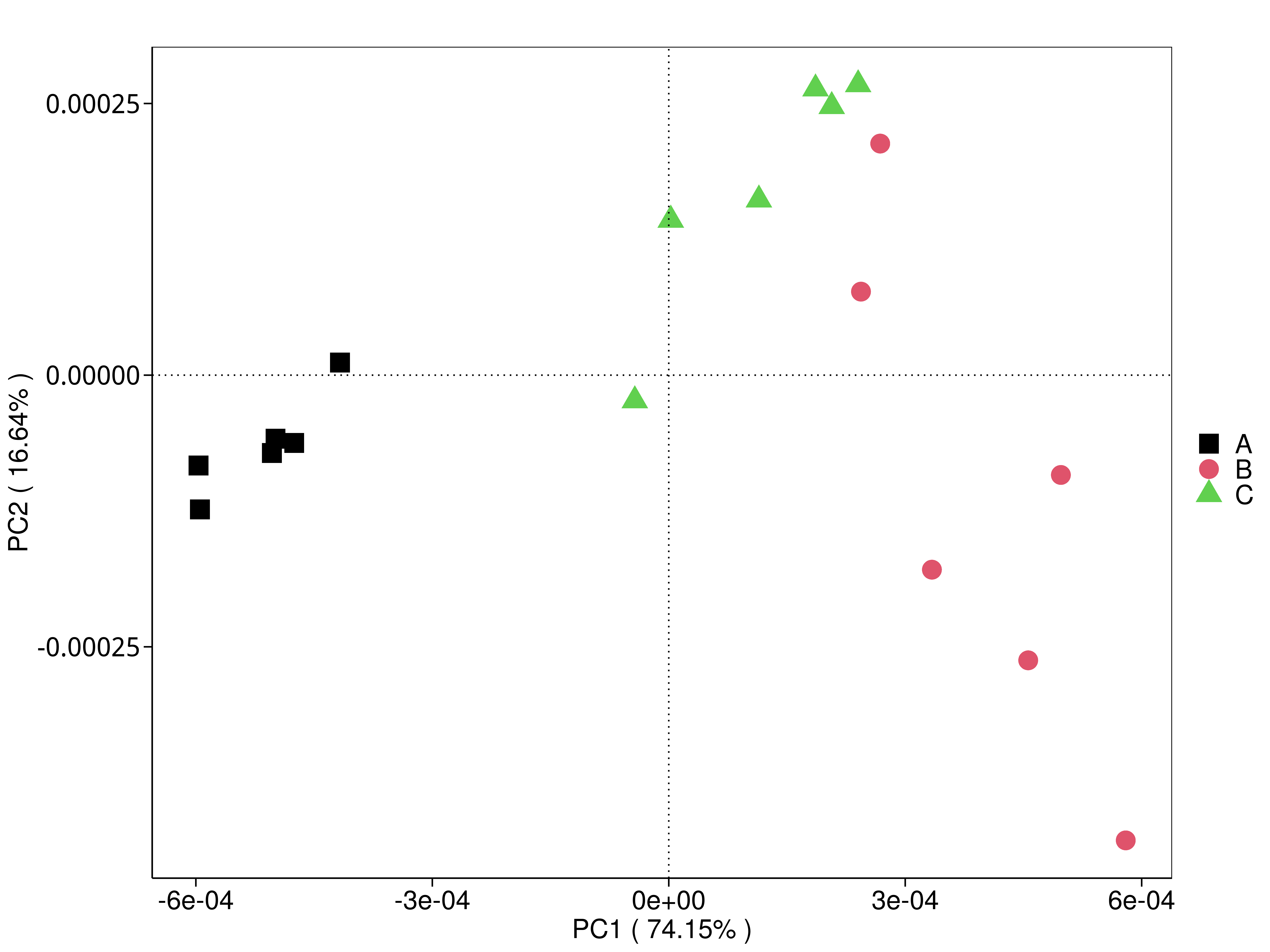
图4.77 基于 CARD 水平的 PCoA 结果展示
说明:PCoA分析,横坐标表示一个主坐标,纵坐标表示另一个主坐标,百分比表示主坐标对样本差异的贡献值;图中的每个点表示一个样本,同一个组的样本使用同一种颜色表示
结果目录:
标注样品名的PCoA图见 : result/07.ResistantGene/CARD/Diversity/PCoA_group1/ARO/PCoA_lable.{png,pdf}
未标注样品名的PCoA图见 : result/07.ResistantGene/CARD/Diversity/PCoA_group1/ARO/PCoA.{png,pdf}
未标示样品名称的带聚类圈的PCoA图见 : result/07.ResistantGene/CARD/Diversity/PCoA_group1/ARO/PCoA_circle.{png,pdf}
标示样品名称的带聚类圈的PCoA图见 : result/07.ResistantGene/CARD/Diversity/PCoA_group1/ARO/PCoA_circleLable.{png,pdf}
4.7.2.4.2 MGEs
图4.78 基于 MGEs 水平的 PCoA 结果展示
说明:PCoA分析,横坐标表示一个主坐标,纵坐标表示另一个主坐标,百分比表示主坐标对样本差异的贡献值;图中的每个点表示一个样本,同一个组的样本使用同一种颜色表示
结果目录:
标注样品名的PCoA图见 : result/07.ResistantGene/MGES/*/Diversity/PCoA_group1/*/PCoA_lable.{png,pdf}
未标注样品名的PCoA图见 : result/07.ResistantGene/MGES/*/Diversity/PCoA_group1/*/PCoA.{png,pdf}
未标示样品名称的带聚类圈的PCoA图见 : result/07.ResistantGene/MGES/*/Diversity/PCoA_group1/*/PCoA_circle.{png,pdf}
标示样品名称的带聚类圈的PCoA图见 : result/07.ResistantGene/MGES/*/Diversity/PCoA_group1/*/PCoA_circleLable.{png,pdf}
4.7.2.5 非度量多维尺度(NMDS)分析
NMDS是非线性模型,其目的是为了克服线性模型的缺点,更好地反映生态学数据的非线性结构(Legendre, 1998),应用NMDS分析,根据样本中包含的功能信息,以点的形式反映在多维空间上,而不同样本间的差异程度则是通过点与点间的距离体现,能够反映样本的组间或组内差异。基于不同分类层级的功能丰度表得到Bray-Curtis 距离矩阵,我们进行了 NMDS分析。
4.7.2.5.1 CARD
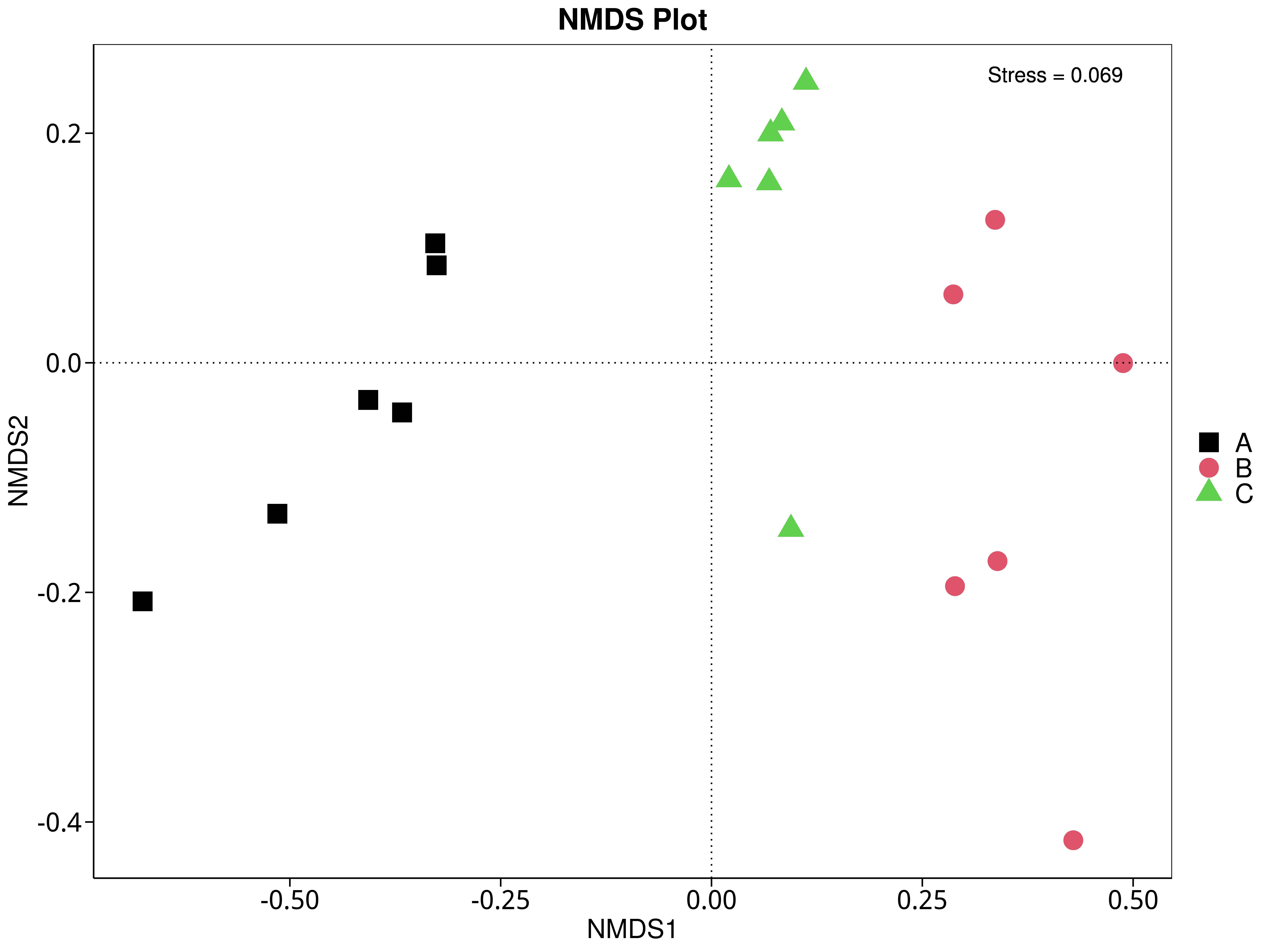
图4.79 基于 CARD 水平的 NMDS 结果展示
说明:图中的每个点表示一个样本,点与点之间的距离表示差异程度,同一个组的样本使用同一种颜色表示。Stress小于0.2时,说明NMDS可以准确反映样本间的差异程度
结果目录:
标注样品名的NMDS图见 : result/07.ResistantGene/CARD/Diversity/NMDS_group1/ARO/NMDS_lable.{png,pdf}
未标注样品名的NMDS图见 : result/07.ResistantGene/CARD/Diversity/NMDSgroup1/ARO/NMDS.{png,pdf}
未标示样品名称的带聚类圈的NMDS图见 : result/07.ResistantGene/CARD/Diversity/NMDS_group1/ARO/NMDS_circle.{png,pdf}
标示样品名称的带聚类圈的NMDS图见 : result/07.ResistantGene/CARD/Diversity/NMDS_group1/ARO/NMDS_circleLable.{png,pdf}
4.7.2.5.2 MGEs
图4.80 基于 MGEs 水平的 NMDS 结果展示
说明:图中的每个点表示一个样本,点与点之间的距离表示差异程度,同一个组的样本使用同一种颜色表示。Stress小于0.2时,说明NMDS可以准确反映样本间的差异程度
结果目录:
标注样品名的NMDS图见 : result/07.ResistantGene/MGEs/*/Diversity/PCA_group1/*/NMDS_lable.{png,pdf}
未标注样品名的NMDS图见 : result/07.ResistantGene/MGEs/*/Diversity/PCA_group1/*/NMDS.{png,pdf}
未标示样品名称的带聚类圈的NMDS图见 : result/07.ResistantGene/MGES/*/Diversity/PCA_group1/*/NMDS_circle.{png,pdf}
标示样品名称的带聚类圈的NMDS图见 : result/07.ResistantGene/MGES/*/Diversity/PCA_group1/*/NMDS_circleLable.{png,pdf}
4.7.3 抗性基因组间差异统计检验分析
4.7.3.1 Anosim 分析
Anosim分析是一种非参数检验,用来检验组间的差异是否显著大于组内差异,从而判断分组是否有意义,详细计算过程可查看Anosim(https://www.rdocumentation.org/packages/vegan/versions/2.3-5/topics/anosim)。
4.7.3.1.1 CARD
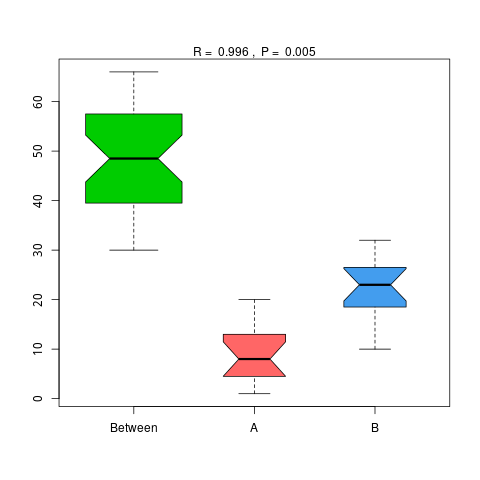
图4.81 基于CARD 水平的 Anosim 分析
说明:横向为分组信息,纵向为距离信息。Between为两组合并信息,between中位线高于另外两组中位线为分组信息较好。R-value介于(-1,1)之间,R-value大于0,说明组间差异大于组内差异。R-value小于0,说明组内差异大于组间差异,统计分析的可信度用 P-value 表示,P< 0.05 表示统计具有显著性
结果目录:
Anosim分析结果见 : result/07.ResistantGene/CARD/StatisticalTest/Anosim_group1
4.7.3.1.2 MGEs
图4.82 基于 MGEs 水平的 Anosim 分析
说明:横向为分组信息,纵向为距离信息。Between为两组合并信息,between中位线高于另外两组中位线为分组信息较好。R-value介于(-1,1)之间,R-value大于0,说明组间差异大于组内差异。R-value小于0,说明组内差异大于组间差异,统计分析的可信度用 P-value 表示,P< 0.05 表示统计具有显著性
结果目录:
Anosim分析结果见 : result/07.ResistantGene/MGES/*/StatisticalTest/Anosim_group1
4.7.3.2 MetaGenomeSeq 分析
为了研究组间具有显著性差异的功能,从不同层级的功能丰度表出发,利用 MetaGenomeSeq 方法对组间的功能丰度数据进行假设检验得到 p 值,通过对 p 值的校正,得到 q 值;最后根据 q 值筛选具有显著性差异的功能,并绘制差异功能在组间的丰度分布箱图。
4.7.3.2.1 CARD
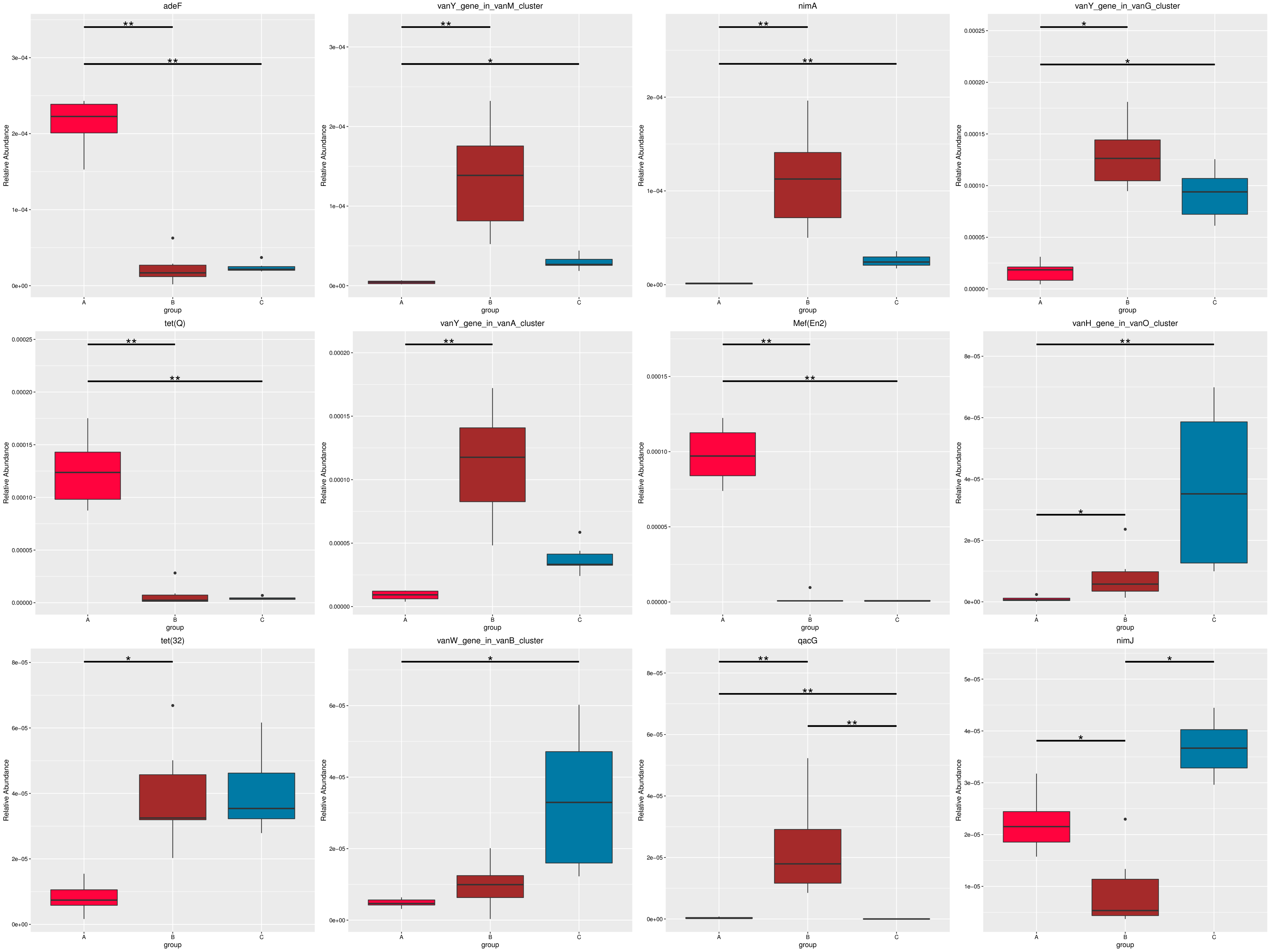
图4.83 基于CARD 水平的 MetaGenomeSeq 分析
图中,横向为样品分组;纵向为对应功能的相对丰度。横线代表具有显著性差异的两个分组,没有则表示此功能在两个分组间不存在差异。“*”表示两组间差异显著(q value < 0.05),“**”表示两组间差异极显著(q value < 0.01)。
根据组间具有差异的功能进行主成分 PCA 分析和丰度聚类热图分析,展示结果如下:
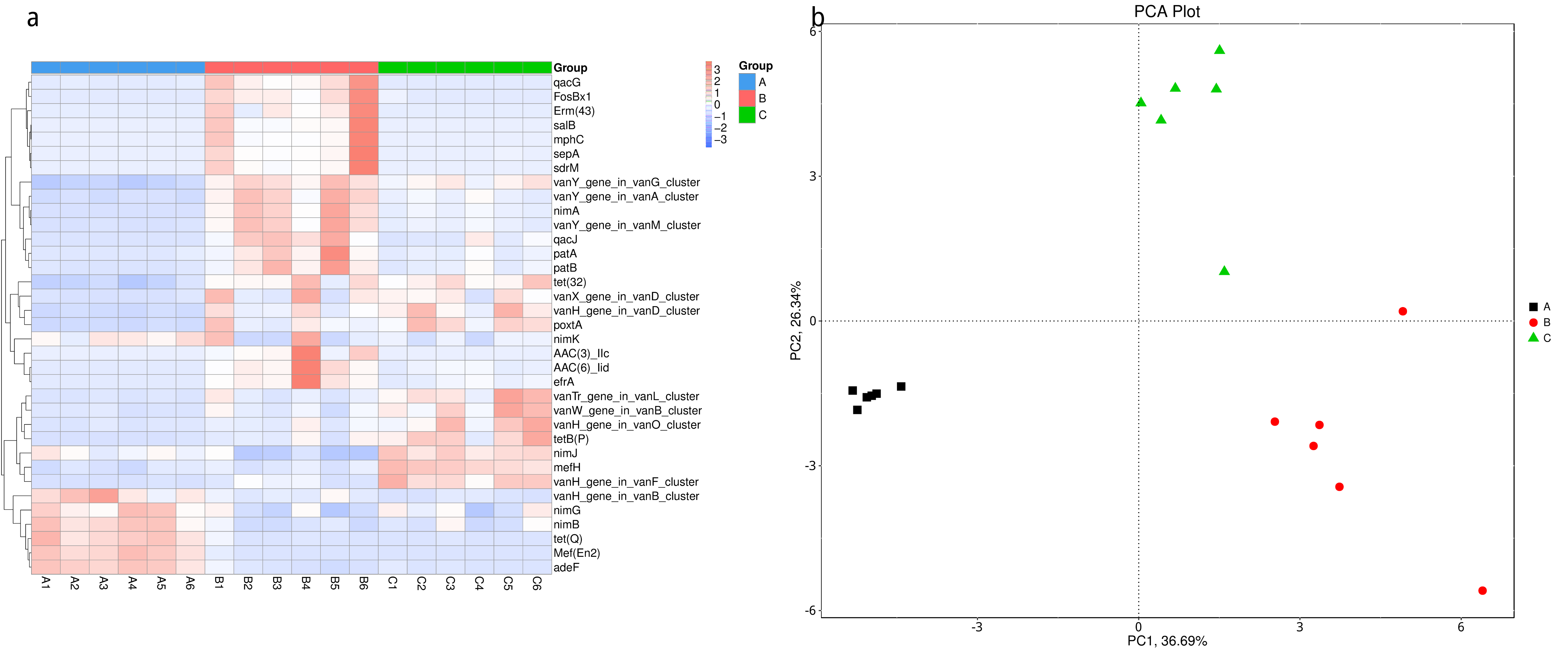
图4.84 基于显著性差异功能的丰度聚类热图和 PCA 分析
a) 为显著性差异功能的丰度聚类热图:横向为样品信息;纵向为功能注释信息;图中左侧的聚类树为功能聚类树;中间热图对应的值为每一行功能相对丰度经过标准化处理后得到的 Z 值;即一个样品在某个分类上的 Z 值为样品在该分类上的相对丰度和所有样品在该分类的平均相对丰度的差除以所有样品在该分类上的标准差所得到的值;b) 为显著性差异功能的 PCA 图:横坐标表示第一主成分,百分比则表示第一主成分对样品差异的贡献值;纵坐标表示第二主成分,百分比表示第二主成分对样品差异的贡献值;图中的每个点表示一个样品,同一个组的样品使用同一种颜色表示。
结果目录 :
MetaGenomeSeq 分析结果见 : result/07.ResistantGene/CARD/StatisticalTest/MetaGenomeSeq_group1/ARO
从 MetaGenomeSeq 分析结果中,筛选出的 P value<=0.05的信息见 : result/07.ResistantGene/CARD/StatisticalTest/MetaGenomeSeq_group1/ARO/*.psig.xls
从 MetaGenomeSeq 分析结果中,筛选出的 Q value<=0.05的信息见 : result/07.ResistantGene/CARD/StatisticalTest/MetaGenomeSeq_group1/ARO/*.qsig.xls
4.7.3.2.2 MGEs
图4.85 基于MGEs 水平的 MetaGenomeSeq 分析
图中,横向为样品分组;纵向为对应功能的相对丰度。横线代表具有显著性差异的两个分组,没有则表示此功能在两个分组间不存在差异。“*”表示两组间差异显著(q value < 0.05),“**”表示两组间差异极显著(q value < 0.01)。
根据组间具有差异的功能进行主成分 PCA 分析和丰度聚类热图分析,展示结果如下:
图4.86 基于显著性差异功能的丰度聚类热图和 PCA 分析
a) 为显著性差异功能的丰度聚类热图:横向为样品信息;纵向为功能注释信息;图中左侧的聚类树为功能聚类树;中间热图对应的值为每一行功能相对丰度经过标准化处理后得到的 Z 值;即一个样品在某个分类上的 Z 值为样品在该分类上的相对丰度和所有样品在该分类的平均相对丰度的差除以所有样品在该分类上的标准差所得到的值;b) 为显著性差异功能的 PCA 图:横坐标表示第一主成分,百分比则表示第一主成分对样品差异的贡献值;纵坐标表示第二主成分,百分比表示第二主成分对样品差异的贡献值;图中的每个点表示一个样品,同一个组的样品使用同一种颜色表示。
结果目录 :
MetaGenomeSeq 分析结果见 : result/07.ResistantGene/MGES/*/StatisticalTest/MetaGenomeSeq_group1/*
从 MetaGenomeSeq 分析结果中,筛选出的 P value<=0.05的信息见 : result/07.ResistantGene/MGES/*/StatisticalTest/MetaGenomeSeq_group1/*/*.psig.xls
从 MetaGenomeSeq 分析结果中,筛选出的 Q value<=0.05的信息见 : result/07.ResistantGene/MGES/*/StatisticalTest/MetaGenomeSeq_group1/*/*.qsig.xls
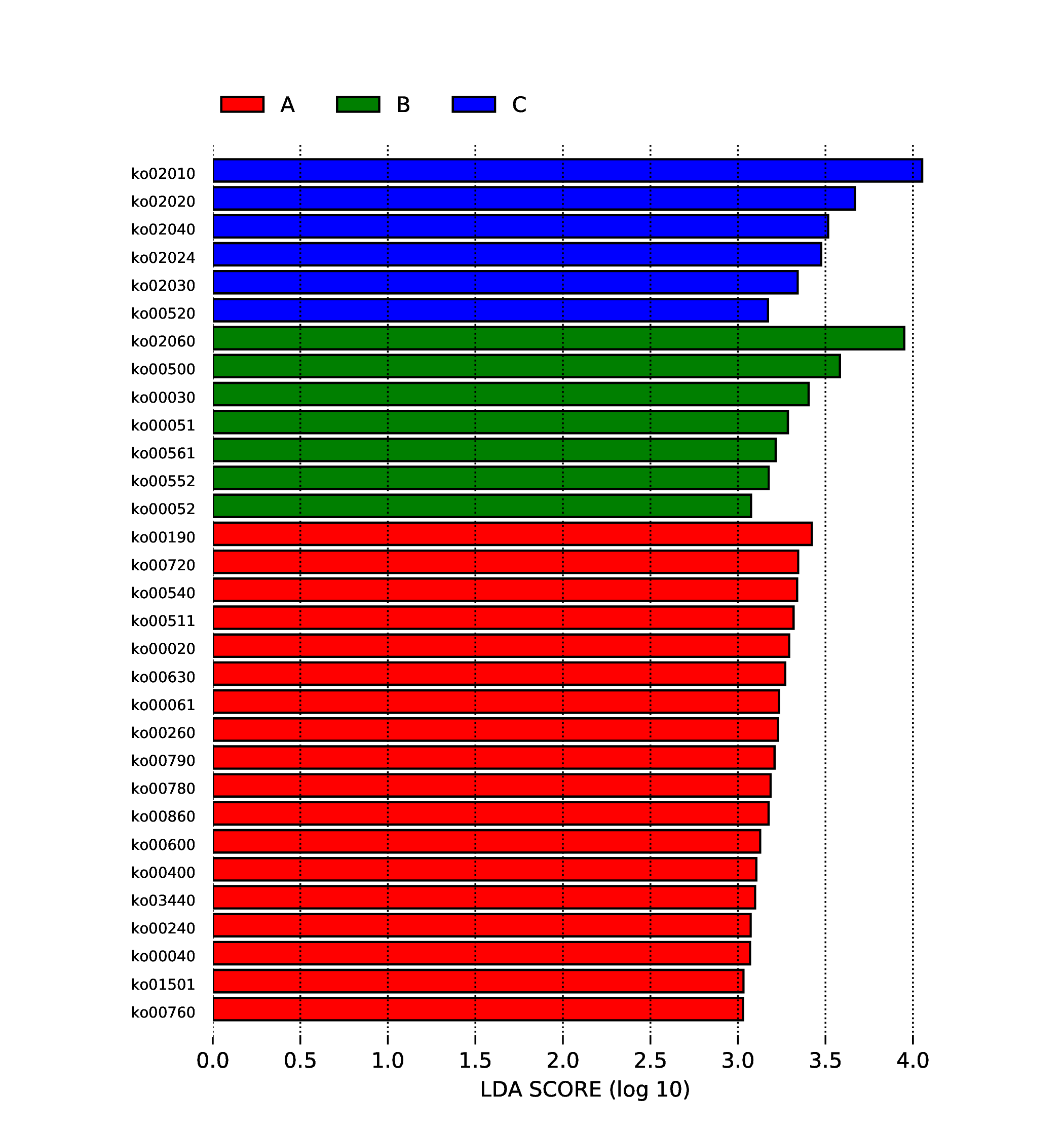
4.7.3.3 LEfSe分析
为了筛选组间具有显著差异的Biomarker,首先通过秩和检验的方法检测不同分组间的差异功能并通过LDA(线性判别分析)实现降维并评估差异功能的影响大小,即得到LDA score ;组间差异功能的LEfSe分析LDA值分布柱状图如下:
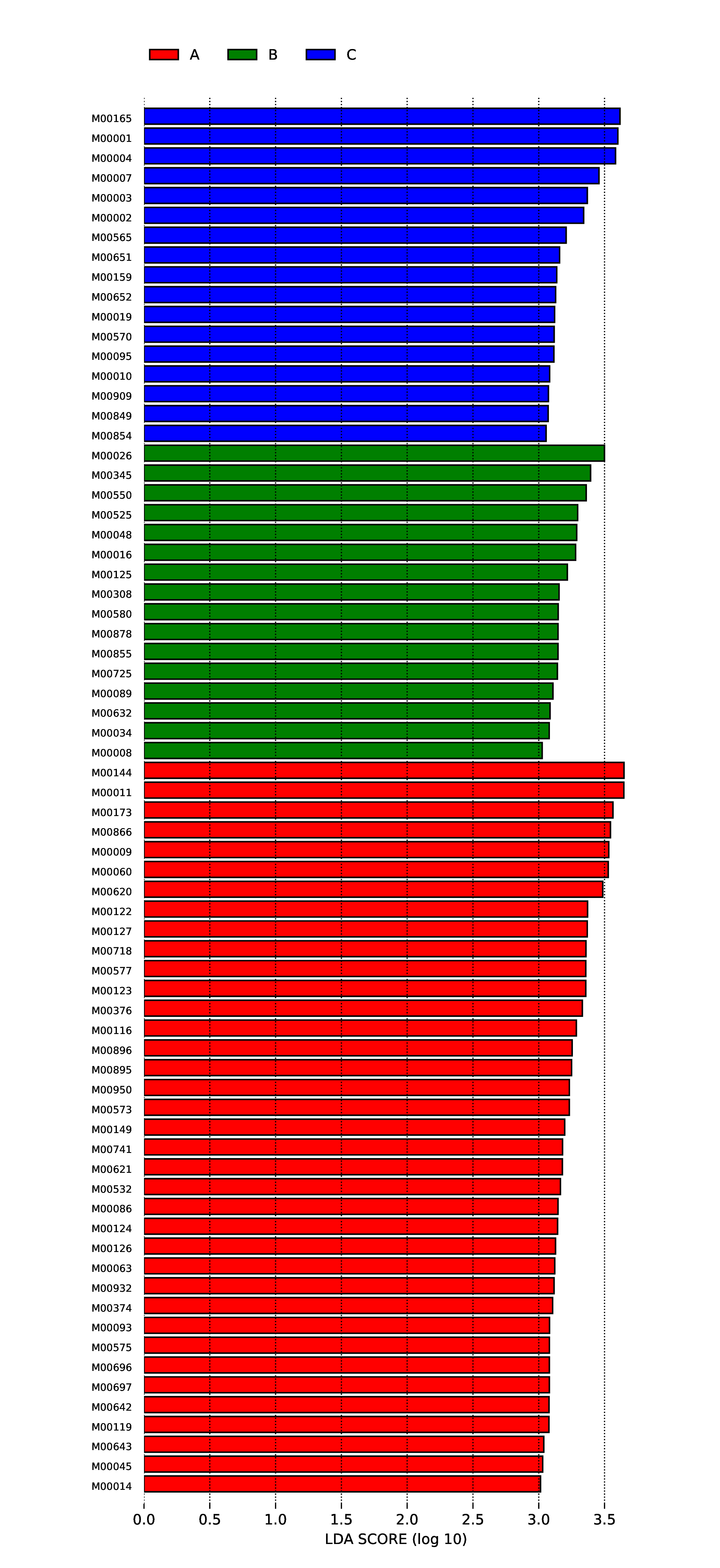
4.7.3.3.1 CARD
图4.87 CARD 的LDA值分布图
说明:图为差异功能的LDA值分布图,LDA值分布柱状图中展示了LDA Score大于设定值(默认设置为3)的功能,即组间具有统计学差异的Biomarker,柱状图的长度代表差异功能的影响大小(即为 LDA Score)。
结果目录 :
LEfSe分析结果见 : result/07.ResistantGene/CARD/StatisticalTest/LEfSe_group1/ARO
4.7.3.3.2 MGEs
图4.88 MGEs 的LDA值分布图
说明:图为差异功能的LDA值分布图,LDA值分布柱状图中展示了LDA Score大于设定值(默认设置为3)的功能,即组间具有统计学差异的Biomarker,柱状图的长度代表差异功能的影响大小(即为 LDA Score)。
结果目录 :
LEfSe分析结果见 : result/07.ResistantGene/MGES/*/StatisticalTest/LEfSe_group1/*
5 附录
5.1 分析软件列表
表5.1 分析软件列表
| 分析内容 | 软件 | 版本 | 参数 | 备注 |
|---|---|---|---|---|
| 数据质控 | fastp | 0.23.1 | -g -q 5 -u 50 -n 15 -l 150 --overlap_diff_limit 1 --overlap_diff_percent_limit 10 | |
| 过滤宿主 | bowtie2 | 2.5.4 | --end-to-end --sensitive --no-hd --no-sq -I 200 -X 400 --threads 8 | |
| 组装 | megahit | 1.2.9 | --presets meta-large -m 180000000000 -t 2 | |
| 基因预测 | GeneMark.hmm | 2.1 | -a -d -f G -p 1 | |
| 基因去冗余 | CD-HIT | 4.5.8 | -T 6 -G 0 -aS 0.9 -g 1 -d 0 -c 0.95 -n 5 -M 8000 | |
| reads mapping | bowtie2 | 2.2.4 | --end-to-end --sensitive --no-hd --no-sq -I 200 -X 400 --threads 8 | |
| 相关性热图 | R软件 | 2.15.3 | R package = corrplot | |
| corepan | R软件 | 2.15.3 | R package = ggplot2 | |
| venn图、花瓣图 | R软件 | v3.0.3 | R package = VennDiagram | |
| 基因数目差异分析 | R软件 | 2.15.3 | R package = ggplot2 | |
| 基于基因数目的样品间相关性分析 | R软件 | 2.15.3 | R package = corrplot | |
| Micro_NR比对注释 | diamond | 2.1.9 | -p 4 -e 1e-5 -k 50 | Micro_NR(Version: 2024.03) |
| KEGG比对注释 | diamond | 2.1.9 | -p 4 -e 1e-5 -k 50 --id 30 --sensitive | KEGG(Version: 2024.03) |
| eggNOG比对注释 | diamond | 2.1.9 | -p 4 -e 1e-5 -k 50 --id 30 --sensitive | eggNOG(Version: 5.0) |
| CAZy比对注释 | diamond | 2.1.9 | -p 4 -e 1e-5 -k 50 --id 30 --sensitive | CAZy(Version: 2024.03) |
| VFDB比对注释 | diamond | 2.1.9 | -p 4 -e 1e-5 -k 50 --id 30 --sensitive | VFDB(Version: 2024.06) |
| PHI比对注释 | diamond | 2.1.9 | -p 4 -e 1e-5 -k 50 --id 30 --sensitive | PHI(Version: 2023.03) |
| CARD比对注释 | RGI | 6.0.2 | -d wgs --clean | CARD(Version:3.2.6) |
| MGEs比对注释 | blastn | 2.12.0 | -num_threads 6 -outfmt 6 -evalue 1e-5 | Integrall(Version: 2021.09),Isfinder(Version: 2023.10),Plasmid(Version: 2021.06) |
| Alpha指数分析 | R软件 | 2.15.3 | R package = vegan | |
| PCA分析 | R软件 | 3.0.3 | R package = FactoMineR, ggplot2,grid | |
| NMDS | R软件 | 2.15.3 | R package = vegan,permute,lattice | |
| Anosim | R软件 | 2.15.3 | R package = vegan,permute,lattice | |
| PCoA | R软件 | 2.15.3 | R package = extrafont,ggplot2,grid | |
| 聚类热图 | R软件 | 3.1.0 | R package = pheatmap | |
| MetaGenomeSeq | R软件 | 2.15.3 | q value (Benjamini and Hochberg FDR) < 0.05 | |
| MetaGenomeSeq热图 | R软件 | 3.1.0 | R package = pheatmap | |
| MetaGenomeSeq combined | R软件 | 2.15.3 | R package = ggplot2 | |
| LEfSe | LEfSe软件 | 1.0 | 物种LDA Score>4,功能LDA Score>3 | |
| Krona | perl | 5.18.2 | use KronaTools | |
| 相对丰度柱形图 | perl | 5.18.2 | use SVG | |
| 聚类树 | perl | 5.18.2 | use SVG | |
| circos单圈图 | circos | v0.64 | ||
| 功能相对丰度柱形图 | perl | 5.18.2 | use SVG | |
| 代谢通路比较分析 | perl | 5.18.2 | use SVG |
5.2 Methods
5.3 结果文件解压方法及结果文件格式说明
表5.2 常见文件格式打开方法
| 文件格式 | 操作系统 | 描述 |
|---|---|---|
| *.gz 格式 | Unix/Linux/Mac | 使用gzip -d *.gz命令打开 |
| *.gz 格式 | Windows | 使用WinRAR、7-Zip等解压缩软件打开 |
| *.zip 格式 | Unix/Linux/Mac | 使用unzip*.zip命令打开 |
| *.zip 格式 | Windows | 使用WinRAR、7-Zip等解压缩软件打开 |
| *.tar 格式 | Unix/Linux/Mac | 使用tar -xvf *.tar命令打开 |
| *.tar 格式 | Windows | 使用WinRAR、7-Zip等解压缩软件打开 |
结果文件建议使用Excel或者EditPlus等专业文本编辑器打开。推荐使用火狐浏览器进行网页版结题报告浏览,下载地址:http://www.firefox.com.cn/download/
6 参考文献
Bäckhed F, Roswall J, Peng Y, et al. Dynamics and Stabilization of the Human Gut Microbiome during the First Year of Life. Cell Host Microbe. 2015;17(6):852. doi:10.1016/j.chom.2015.05.012
Breiman L. Random Forests. Machine Learning. 2001;45(1):5-32.
Buchfink B, Xie C, Huson DH. Fast and sensitive protein alignment using DIAMOND. Nat Methods. 2015;12(1):59-60. doi:10.1038/nmeth.3176
Cantarel BL, Coutinho PM, Rancurel C, Bernard T, Lombard V, Henrissat B. The Carbohydrate-Active EnZymes database (CAZy): an expert resource for Glycogenomics. Nucleic Acids Res. 2009;37(Database issue):D233-D238. doi:10.1093/nar/gkn663
Chen K, Pachter L. Bioinformatics for whole-genome shotgun sequencing of microbial communities. PLoS Comput Biol. 2005;1(2):106-112. doi:10.1371/journal.pcbi.0010024
Cotillard A, Kennedy SP, Kong LC, et al. Dietary intervention impact on gut microbial gene richness [published correction appears in Nature. 2013 Oct 24;502(7472)580]. Nature. 2013;500(7464):585-588. doi:10.1038/nature12480
Feng Q, Liang S, Jia H, et al. Gut microbiome development along the colorectal adenoma-carcinoma sequence. Nat Commun. 2015;6:6528. Published 2015 Mar 11. doi:10.1038/ncomms7528
Fu L, Niu B, Zhu Z, Wu S, Li W. CD-HIT: accelerated for clustering the next-generation sequencing data. Bioinformatics. 2012;28(23):3150-3152. doi:10.1093/bioinformatics/bts565
Handelsman J, Rondon MR, Brady SF, Clardy J, Goodman RM. Molecular biological access to the chemistry of unknown soil microbes: a new frontier for natural products. Chem Biol. 1998;5(10):R245-R249. doi:10.1016/s1074-5521(98)90108-9
Huson DH, Mitra S, Ruscheweyh HJ, Weber N, Schuster SC. Integrative analysis of environmental sequences using MEGAN4. Genome Res. 2011;21(9):1552-1560. doi:10.1101/gr.120618.111
Jaime Huerta-Cepas, Damian Szklarczyk, Kristoffer Forslund, Helen Cook, Davide Heller, Mathias C. Walter, Thomas Rattei, Daniel R. Mende, Shinichi Sunagawa, Michael Kuhn, Lars Juhl Jensen, Christian von Mering, Peer Bork; eggNOG 4.5: a hierarchical orthology framework with improved functional annotations for eukaryotic, prokaryotic and viral sequences, Nucleic Acids Research, Volume 44, Issue D1, 4 January 2016, Pages D286–D293
Jia B, Raphenya AR, Alcock B, et al. CARD 2017: expansion and model-centric curation of the comprehensive antibiotic resistance database. Nucleic Acids Res. 2017;45(D1):D566-D573. doi:10.1093/nar/gkw1004
Karlsson FH, Fåk F, Nookaew I, et al. Symptomatic atherosclerosis is associated with an altered gut metagenome. Nat Commun. 2012;3:1245. doi:10.1038/ncomms2266
Karlsson FH, Tremaroli V, Nookaew I, et al. Gut metagenome in European women with normal, impaired and diabetic glucose control. Nature. 2013;498(7452):99-103. doi:10.1038/nature12198
Kanehisa M, Furumichi M, Tanabe M, Sato Y, Morishima K. KEGG: new perspectives on genomes, pathways, diseases and drugs. Nucleic Acids Res. 2017;45(D1):D353-D361. doi:10.1093/nar/gkw1092
Kanehisa M, Goto S, Hattori M, et al. From genomics to chemical genomics: new developments in KEGG. Nucleic Acids Res. 2006;34(Database issue):D354-D357. doi:10.1093/nar/gkj102
Klipper-Aurbach Y, Wasserman M, Braunspiegel-Weintrob N, et al. Mathematical formulae for the prediction of the residual beta cell function during the first two years of disease in children and adolescents with insulin-dependent diabetes mellitus. Med Hypotheses. 1995;45(5):486-490. doi:10.1016/0306-9877(95)90228-7
Le Chatelier E, Nielsen T, Qin J, et al. Richness of human gut microbiome correlates with metabolic markers. Nature. 2013;500(7464):541-546. doi:10.1038/nature12506
Legendre P, Legendre L. Numerical ecology, 2nd edition[J].1998.
Li D, Liu CM, Luo R, Sadakane K, Lam TW. MEGAHIT: an ultra-fast single-node solution for large and complex metagenomics assembly via succinct de Bruijn graph. Bioinformatics. 2015;31(10):1674-1676. doi:10.1093/bioinformatics/btv033
Li J, Jia H, Cai X, et al. An integrated catalog of reference genes in the human gut microbiome. Nat Biotechnol. 2014;32(8):834-841. doi:10.1038/nbt.2942
Li W, Godzik A. Cd-hit: a fast program for clustering and comparing large sets of protein or nucleotide sequences. Bioinformatics. 2006;22(13):1658-1659. doi:10.1093/bioinformatics/btl158
Martínez JL, Coque TM, Baquero F. What is a resistance gene? Ranking risk in resistomes. Nat Rev Microbiol. 2015;13(2):116-123. doi:10.1038/nrmicro3399
McArthur AG, Waglechner N, Nizam F, et al. The comprehensive antibiotic resistance database. Antimicrob Agents Chemother. 2013;57(7):3348-3357. doi:10.1128/AAC.00419-13
Mende DR, Waller AS, Sunagawa S, et al. Assessment of metagenomic assembly using simulated next generation sequencing data [published correction appears in PLoS One. 2014;9(11):e114063]. PLoS One. 2012;7(2):e31386. doi:10.1371/journal.pone.0031386
Nielsen HB, Almeida M, Juncker AS, et al. Identification and assembly of genomes and genetic elements in complex metagenomic samples without using reference genomes. Nat Biotechnol. 2014;32(8):822-828. doi:10.1038/nbt.2939
Rao C R. The Use and Interpretation of Principal Component Analysis in Applied Research[J]. Sankhyā: The Indian Journal of Statistics, Series A (1961-2002), 1964, 26(4):329-358.
Oh J, Byrd AL, Deming C, et al. Biogeography and individuality shape function in the human skin metagenome. Nature. 2014;514(7520):59-64. doi:10.1038/nature13786
Ondov BD, Bergman NH, Phillippy AM. Interactive metagenomic visualization in a Web browser. BMC Bioinformatics. 2011;12:385. Published 2011 Sep 30. doi:10.1186/1471-2105-12-385
Raes J, Foerstner KU, Bork P. Get the most out of your metagenome: computational analysis of environmental sequence data. Curr Opin Microbiol. 2007;10(5):490-498. doi:10.1016/j.mib.2007.09.001
Qin J, Li R, Raes J, et al. A human gut microbial gene catalogue established by metagenomic sequencing. Nature. 2010;464(7285):59-65. doi:10.1038/nature08821
Qin J, Li Y, Cai Z, et al. A metagenome-wide association study of gut microbiota in type 2 diabetes. Nature. 2012;490(7418):55-60. doi:10.1038/nature11450
Qin N, Yang F, Li A, et al. Alterations of the human gut microbiome in liver cirrhosis. Nature. 2014;513(7516):59-64. doi:10.1038/nature13568
Scher JU, Sczesnak A, Longman RS, et al. Expansion of intestinal Prevotella copri correlates with enhanced susceptibility to arthritis. Elife. 2013;2:e01202. Published 2013 Nov 5. doi:10.7554/eLife.01202
Segata N, Izard J, Waldron L, et al. Metagenomic biomarker discovery and explanation. Genome Biol. 2011;12(6):R60. Published 2011 Jun 24. doi:10.1186/gb-2011-12-6-r60
Sunagawa S, Coelho LP, Chaffron S, et al. Ocean plankton. Structure and function of the global ocean microbiome. Science. 2015;348(6237):1261359. doi:10.1126/science.1261359
Tringe SG, Rubin EM. Metagenomics: DNA sequencing of environmental samples. Nat Rev Genet. 2005;6(11):805-814. doi:10.1038/nrg1709
Tringe SG, von Mering C, Kobayashi A, et al. Comparative metagenomics of microbial communities. Science. 2005;308(5721):554-557. doi:10.1126/science.1107851
Villar E, Farrant GK, Follows M, et al. Ocean plankton. Environmental characteristics of Agulhas rings affect interocean plankton transport. Science. 2015;348(6237):1261447. doi:10.1126/science.1261447
Zeller G, Tap J, Voigt AY, et al. Potential of fecal microbiota for early-stage detection of colorectal cancer. Mol Syst Biol. 2014;10(11):766. Published 2014 Nov 28. doi:10.15252/msb.20145645
Zhu W, Lomsadze A, Borodovsky M. Ab initio gene identification in metagenomic sequences. Nucleic Acids Res. 2010;38(12):e132. doi:10.1093/nar/gkq275